2. 中国科学院大学 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
2014年5月,Nature杂志报道了一项合成生物学的重大突破[1]。来自美国斯克利普斯研究所(The Scripps Research Institute)的Floyd E.Romesberg研究小组成功构建了包含一对人造碱基对的六核酸分子人工合成细菌,首次成功实现人造碱基对的体内复制。这一里程碑式的成果标志着人造碱基的研究工作正式从体外步入了体内。人造碱基不仅在结构上模拟天然碱基的特征,还从实际功能出发来进行设计和优化,其应用前景之广阔给人以无限遐想。
新碱基的引入可以丰富有限的DNA、RNA组成元件,利用工程菌及合成生物学技术合成自然界没有而又不易人工合成的抗生素、疫苗及其他产品。如在活疫苗开发领域,人们可以将含有人造碱基的人工合成细菌/病毒作为活疫苗接种,一方面这可以诱发免疫反应而产生疫苗作用;另一方面,因为人体内缺乏相应的人工碱基原料,所以该细菌/病毒不会在体内复制。因此,相比于天然细菌/病毒疫苗,含有人造碱基的人工合成细菌/病毒疫苗可能更为安全。对于目前还处于起始阶段的DNA计算机而言,在原有的ATCG的4字母碱基基础上新增加2个字母,就可以使DNA计算机从4进制升为6进制,其存储能力和运算能力都将得到极大提升,无疑会极大地促进此项技术的发展。
尽管上述设想距离最终实现还有很长的路要走,但六核酸分子人工合成细菌的成功实现无疑是“人工合成生命”领域的一次有益的尝试;其也会对化学合成生物学研究领域的发展起到极大的推动促进作用,并可能为合成生物学开辟出一条新的、更安全的别样健康发展之路;无疑也将对生物安全领域产生极为深远的影响。
1 人造碱基与合成生物学人造碱基的研究发展至今已有近30年的历史,它又被称为非天然碱基对或人工扩展碱基字母,是一种通过对碱基的改造,人工设计合成的可以行使或模拟天然核酸功能、而又具有相对独立性的人造DNA。它的研究属于合成生物学的一个重要分支——化学合成生物学的研究范畴[2]。化学合成生物学(Chemical Synthetic biology)是用以设计、描述并合成自然界不存在的具有新型化学结构的功能生物大分子元件(如新型结构的核酸、氨基酸、小泡结构等)[3]。正如Kool[2]在2000年美国化学会年会上阐述的那样,化学合成生物学关注的焦点并非在基因水平,而是非天然存在的功能性有机分子的化学结构,所以化学合成生物学又可以称为“Bottom-upSynthetic Biology”[4]。
化学合成生物大分子部件除了具有合成生物学生物部件模块化、标准化的特性外,其较强的独立性(即正交性)和可操控性是其与Venter JC类合成生命相比的最大优势所在[4, 5]。由于Venter JC类合成生命并非从头合成生命系统,其最小调节模块是建立在基因水平上的,而在分子水平采用的仍是天然的生物大分子元件(核酸、氨基酸等),所以此类“非从头合成生命”的生物安全问题一直为人们所担忧[6, 7]。与之相反,化学合成生物大分子元件由于在自然界中并不存在,完全独立(正交)于天然生物实体,所以由此类生物元件所组成的“从头合成生命系统(Synthesis of living systems fromscratch)”可能是更“纯洁的”,也是更易操控的[5, 8]。它们可以有效地避免与自然界中已有的生物体之间发生遗传信息重组、转移之类的风险所导致的污染、寄生等生物安全问题;即便它们通过非正常途径(如实验室泄漏)进入自然界,也会因为缺乏相关的人工养料基元(如相关的人工合成碱基、氨基酸等)而迅速死亡。基于此,欧盟生物安全观察员Schmidt博士[7]将之称为终极安全的生物学平台。
在人造碱基近30年的研究历史中,其研究并不仅限于人工扩展碱基相关的复制、转录、翻译,它们还被广泛扩展到核酸检测诊断技术、核酸适配体技术、核酸纳米材料等领域,下面将详述这一领域的代表性研究成果,着重介绍最新的研究进展及人工扩展碱基字母对的成功应用实例,并对这一研究领域的前景和面临的挑战进行展望。
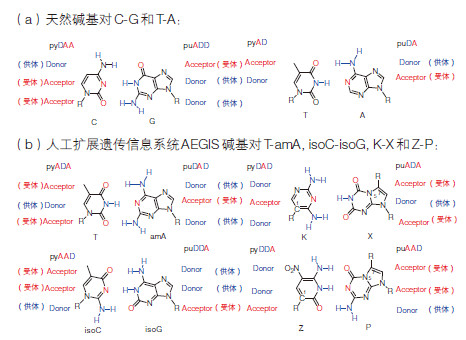
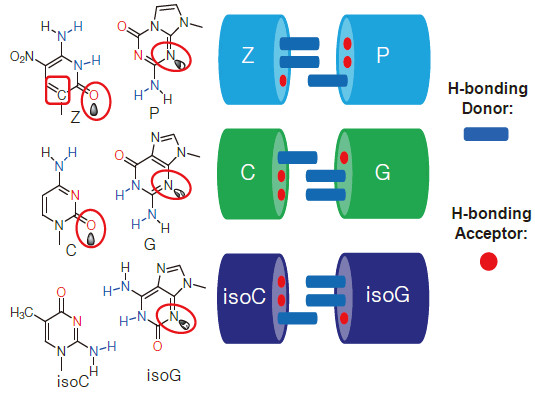
2 人造碱基对的起始及Steven A.Benner的工作Steven A.Benner博士是国际公认的人造碱基研究领域的创始人,他在20世纪80年代中期即开始了发展人工碱基字母对的尝试[4, 6]。在对磷酸基团和脱氧核糖的替换失败之后[3, 4],Benner将注意力转到碱基对上,他发现天然碱基对之间的Watson-Crick互补配对遵循以下两大原则(图 1a):
(1)形状互补——总是在体积较大的嘌呤碱基和体积较小的嘧啶碱基之间形成形状互补配对。
(2)氢键配对——总是通过一个碱基上的氢键供体与另一个碱基上的氢键受体相互作用形成氢键。
基于上述两大原则,如果将天然碱基上的氢键供体和氢键受体基团视作可以互换的元件,将它们进行重新洗牌,很容易就可以设计出多对新的人工模拟碱基对(图 1b)。此处,嘌呤和嘧啶环也可以进行适当的改造(如K-X,Z- P碱基对),这又可以极大地增加人工扩展碱基的种类。图 1b仅列出7种人工扩展碱基及4种新的独立于(正交于)天然碱基的碱基对,其实按照形状(大与小)和氢键互补(氢键供体和受体)原则,还可以得到更多人造碱基对[4, 7, 8]。设计人造碱基对后需要运用有机化学的合成技术来创造出这些新物质,并把它们装配到DNA分子中。观察其作为人造碱基的行为,主要是体外复制和转录特性。经过20多年的发展,目前,人工扩展DNA的生物学性能表征实验主要应包括:含有人造碱基对双链DNA的熔点检测、单侧引物延伸实验、六核酸分子PCR等;以上实验的另一重要目的是检验设计合成人造碱基对的正交性(相对独立性)(图 2)[9, 10, 11]。更进一步的验证实验还包括人造碱基对的体外转录及翻译(图 3)[12, 13, 14]。
 |
| 图 2 人造碱基的体外生物学性能及正交性检验实验(以Z-P碱基对反应体系为例) |
运用上述人造碱基对设计原则及生物学性能表征手段,1989年,Benner研究组[12]首先成功实现了包含人造碱基对isoG-isoC的DNA的体外复制和转录。1990年,他们又实现了包含另一对人造碱基对K-X的DNA的体外复制和转录[13]。随后在1992年,利用isoG-isoC互补配对,借助人工合成含有反密码子CU(isoG)的tRNA的帮助,Benner研究组进一步通过体外翻译系统将含有isoC的人工扩展密码子(isoC)AG翻译成一个非标准氨基酸3-碘化酪氨酸并成功的掺入到一段小肽中[14]。
通过单侧引物延伸实验,对以上两种人造碱基对的体外复制保真性进行检验时,其结果并不十分理想。从20世纪80年代末开始,Benner研究组始终致力于寻找性能更优异的人造扩展碱基。2006年,Benner研究组终于发现了一对比较优秀的人造碱基对Z-P[9]。笔者非常有幸在2005年加入了Benner的研究小组,并作为主要参加人员参与了Z-P碱基对的大部分研发工作。
Z和P采用pyDAA-puADD氢键配对方式(图 1)能够使Z-P碱基对绑定的更加紧密,形成氢键的稳定性要比C:G更强[9]。更为重要的是,基于Watson-Crick互补配对原则设计的Z-P碱基对更容易被DNA聚合酶所识别[15](图 4)。单侧引物延伸及PCR实验也证明了这一点,绝大多数商业化的DNA聚合酶都能识别Z-P碱基对,且保真性可高达99.8%[10, 16]。2006年至今,关于Z-P人造碱基对共有多篇文章发表在PNAS,JACS,Angew Chem.,Nucleic Acid Res.等国际著名生物和化学领域的顶级期刊上,其应用被广泛地扩展到六核酸分子适配体、多重巢式PCR反应、luminex核酸检测、分子信标检测等研究领域[16, 17, 18, 19, 20]。
1997—1998年,斯坦福大学的Kool ET创造出来一种疏水性人造碱基对(Z-F)[21, 22, 23]。他认为氢键配对并非人造碱基对设计所必须,相对而言化学结构的互补、碱基的堆积和静电排斥作用更为重要。他的研究小组正是利用这一设想,成功地创造了第一对疏水性人造碱基对Z-F(T: A碱基对的类似物)(图 5a)。不过Z-F配对的保真性并不好,而A: F以及Z:T之间的配对却更为高效[24]。随后,Kool ET研究组[4]又报道了另一对可被Klenow DNA聚合酶识别的P: Φ疏水性人造碱基对(图 5a)。Kool ET研究组的工作揭示了发展疏水性人工合成碱基对的可能性,从而吸引了包括Hirao I以及Romesberg FE在内的一批合成生物学家开始进行疏水性人造碱基对的研究。
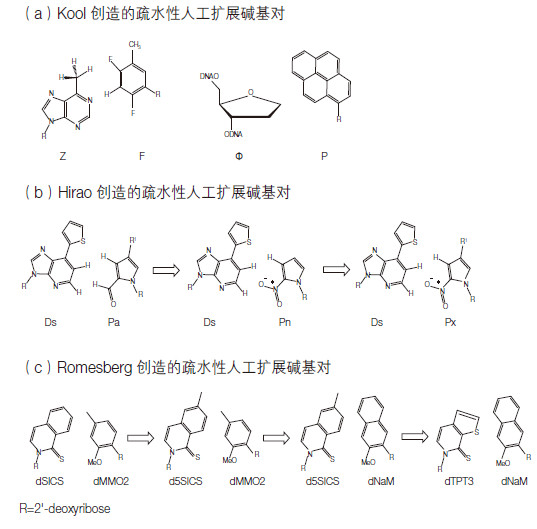 |
| 图 5 一些代表性的疏水性人造碱基对 |
2006年,Hirao等人[25]开发出了一对新的疏水性人工碱基对Ds和Pa(图 5b)。然而,很快他们就发现疏水性人造碱基普遍存在自身配对的问题。偶然地,一次实验上的疏忽让Hirao等人在合成dDsTP时意外合成了一种衍生物γ-氨基dDsTP(dDsTPNH2)。出于好奇,他们使用这一衍生物代替dDsTP进行了单侧引物延伸实验,结果发现在这种情况下,Ds只会掺入到与模板链上Pa相对应的位置而不再掺入到与Ds相对应的位置。基于上述结果,2006年该研究组报道了包含Ds-Pa的人工扩展DNA的高效PCR扩增,和包含这一碱基对及其衍生物(主要是包含各种修饰的Pa)的人工扩展DNA的体外转录[25]。这一工作第一次真正实现了包含一对非天然碱基对的人工扩展DNA的高效体外复制和转录[26]。2009年,Romesberg团队[7]从他们此前开发出的60种疏水性人工碱基中,筛选出一对在体外可以高效复制的疏水性碱基对dMMO2-dSICS(图 5c)。然而他们遇到了和Hirao研究组开发Ds-Pa时所遇到的类似难题,即dSICS的自我配对问题。所不同的是,他们通过在dSICS的5位引入一个甲基,成功地克服了这一难题,创造出了新的人造碱基对dMMO2-d5SICS(图 5c)[26]。
在此基础上,该团队进一步发展出了人造碱基对dNaM-d5SICS(图 5c)[8]。同年,Romesberg研究组报道了包含这两对疏水人工碱基对的PCR扩增以及体外转录[9, 10]。他们在对含有单个dNaM-d5SICS碱基对的模板进行PCR扩增的基础上,还实现了含有两个连续或者不连续的dNaMd5SICS碱基对的模板的高保真性PCR扩增(99.5%—99.6%每个循环)[11]。最近,通过对d5SICS碱基的进一步改造,该团队发展出了一对更为优异的非天然碱基对dNaM-dTPT3(图 5c)[12]。使用OneTaq进行PCR扩增时,dNaM-dTPT3复制的保真性超过了99.98%每个循环,这在某种程度上已经与天然碱基的复制保真性(10-4—10-7的错误率)相当接近了[8]。
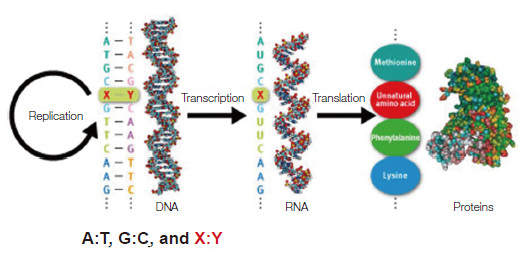
4 含有人工碱基对的六核酸分子合成生命系统的诞生至2012年前后,至少已经有包括Benner、Hirao、Romesberg 3个研究团队独立开发出了数对各方面性能都接近于天然碱基对的人造碱基对。这些工作为非天然碱基对的研究工作从体外走向体内,使用人造碱基对创造半合成生命奠定了坚实的基础。
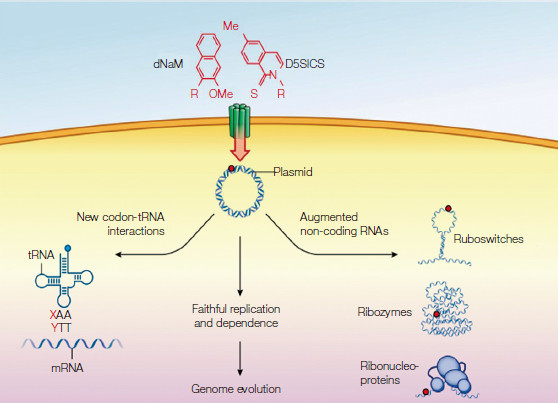
2014年,Romesberg团队首先在这一领域获得了突破性的进展,他们在实现了dNaM-d5SICS的人造碱基对的高效高保真体外PCR复制和转录之后,随即启动了将这一人工碱基对应用于大肠杆菌体内的研究计划。尽管在体外表现优异,但要将其成功的应用到生物体内显然还必须面对更多的挑战。首先,dNaM和d5SICS完全是由人工创造出来的非天然碱基,因而Romesberg团队面临的第一个难题是如何保证细胞内有足够的dNaMTP以及d5SICSTP单体以用于这种人工DNA的复制。为了解决这一问题,Romesberg FE研究组尝试了一系列不同来源的核苷三磷酸转运蛋白,最终发现一种海藻叶绿体的核苷三磷酸转运蛋白PtNTT2能够高效地将添加在培养基中的dNaMTP和d5SICSTP单体运输到细胞质中[1]。随后,通过向培养基中添加磷酸钾,该团队又有效缓解了这两种人工合成核苷三磷酸在培养基以及细胞内容易降解的问题[1]。
此后,该团队构建了一个含有一对人工碱基对的质粒pINF,并且系统地研究了这个质粒在大肠杆菌中的复制情况[1]。研究发现,在开启转运蛋白PtNTT2表达以及培养基中添加相应的外源人工核苷三磷酸的情况下,这一半合成质粒能够以适当的速度和准确度进行复制,并且几乎不阻碍大肠杆菌细胞生长,也没有明显表现出其他失去非天然碱基对的迹象[1]。研究结果显示,这一半合成质粒中的非天然碱基对的复制保真性超过了99.4%,这一保真性与某些病毒的DNA聚合酶相当。新碱基至少复制了24轮,并维持了近一周时间。当没有这一转运蛋白表达或是没有新碱基提供时,非天然碱基d5SICS和dNaM将会逐渐被天然碱基A、T、C、G所替换,并最终从基因组中完全消失。而这种碱基替换已被证明源于DNA复制中的碱基错配而不是细胞内的DNA修复系统(图 6)[1]。
2014年5月7日,Romesberg领导的研究团队在Nature上在线优先发表论文,宣告了包含一对人造碱基对(dNaM-d5SICS)的六核酸分子半合成大肠杆菌的诞生。同期Nature为这一工作配发了评论[17],5月9日出版的Science也对这一工作作出了评论[19]。这一工作可以说是迄今为止人造碱基研究领域最为重要的“里程碑”之一,从而标志着有关人造碱基对的研究工作正式从体外步入了体内。Romesberg的成功激发了人们的无限遐想:有没有可能添加更多的碱基呢?能不能用这些新零件创造出更复杂的生物呢?正如罗斯·泰尔在Nature的评论中所阐述的,“他们揭示了遗传学的一种新机制,使得诞生更加丰富的生物形态成为可能,并有可能创造更加美好的生物学未来”。Romesberg甚至想得更远,他认为虽然此次研究中的人工碱基对还不能参与制造新型蛋白质,但从理论上说,新增加两个字母,DNA就有望从4进制升格为6进制,6种碱基意味着更多的排列组合,更庞大的氨基酸编码库,即可将构成蛋白质的氨基酸从目前的20种提升至172种。
5 人造碱基对在生命健康等领域的应用人造碱基对的应用不仅仅限于从头合成生命,其应用还被扩展到了核酸检测诊断技术、六分子核酸适配体技术等领域,其在分支DNA检测(branched DNAassay)诊断试剂盒中的应用是一个的典型范例。此诊断试剂盒目前已在多个国家上市,被广泛应用于对HIV、HPV和HCV的临床检测诊断,仅在美国,每年就有400 000人应用这一诊断试剂盒,年产值超过1亿美元。
分支DNA检测技术产生得很早,依据核酸杂交原理对目标基因DNA或RNA进行检测。该系统主要由目标检测系统(包括Target probe和capture probe等)、branched DNA信号放大系统(包括label probe、preamplifier和amplifier)组成(图 7a)[21, 22]。Benner等人成功地将第一对人造碱基对(isoG和isoC)应用此技术(又被称为三代branched DNA检测技术),他们向上述各种探针中掺入大约30%的非天然碱基isoC和isoG,改善了由于多种不同的探针在同一体系中使用所产生的背景噪音信号从而使检测灵敏度从100 000个HIV分子/毫升升至1 000个HIV分子/毫升[20, 23, 27]。
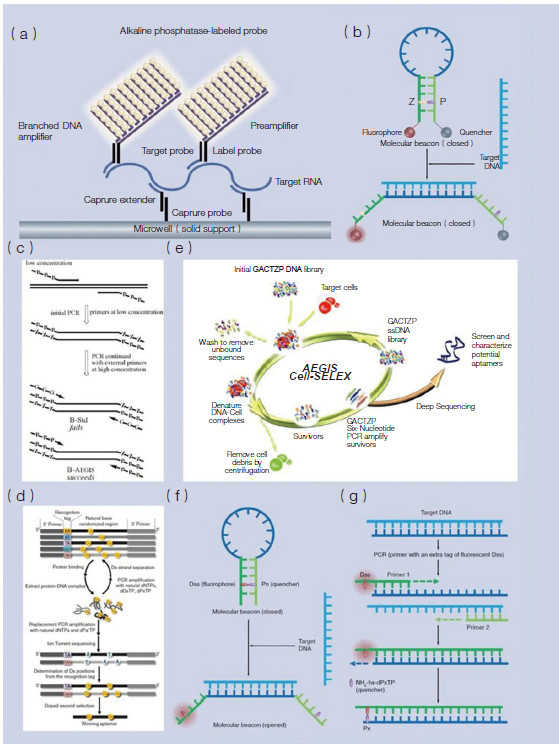 |
| 图 7 一些人工扩展碱基的应用实例 |
人造碱基对在分子信标(molecular beacon)核酸检测技术中的应用也被广泛尝试。2008年,Benner研究组报道了一种新型的分子信标(图 7b)[28]。在这种分子信标中,非天然碱基对Z-P被引入到分子信标的茎部。由于Z-P碱基对与天然碱基对之间存在正交性,使得各种非特异性结合明显减少,从而显著地提高了分子信标的检测灵敏度[28]。2010年Benner研究组还将Z-P碱基对引入到反向巢式PCR的引物中,同样取得了非常理想地提高灵敏度的效果(图 7c)[29]。
科学家们也尝试将人工扩展碱基应用于核酸适配体(aptamer)研究领域。2013年,Hirao研究组首次将非天然碱基引入到了aptamer筛选中(图 7d)[30]。2014年,Benner研究组首次将完全随机的人工扩展DNA文库成功用于aptamer筛选(图 7e)[31]。结果证明,相对于传统aptamer动辄15—20轮次的筛选工作量而言,采用人工扩展DNA文库进行筛选的轮次和工作量大大降低,筛选得到的aptamer性能也更好[30, 31]。
对于人造碱基的应用研究,除了上述提到的基于正交性和多样性所产生的各种应用之外,科学家的一些偶然发现也促成了一些新的应用。比如Hirao研究组在优化各种非天然碱基对时,发现一种Ds碱基的衍生物Dss可以发出强烈的荧光,这种碱基同样可以与Pn或者Px形成稳定配对,更为重要的是,一旦配对形成,Dss碱基所发出的荧光会被完全淬灭[32]。基于这一发现,Hirao I研究组成功开发了利用这一新型碱基对作为荧光和淬灭基团的分子信标(图 7f),并将这一碱基对成功地用在了实时定量PCR中(图 7g)[32]。
6 总结和展望除了人工改造碱基之外,科学家们也一直在进行氨基酸的人工改造。以斯克利普斯研究所Peter Schultz为代表的一些合成生物学家已实现了向蛋白质中定点掺入各种非天然氨基酸以实现各种功能,不过他们是利用天然碱基的无义密码子TAG来进行编码的,因而一次只能掺入一种非天然氨基酸。要想同时向蛋白质中掺入多种不同的非天然氨基酸,必然需要对现有的遗传信息系统进行人工扩展。从Benner[2]率先提出并开展有关人造碱基对的研究工作以来,已经过去了20多年,一系列性能优越的人造碱基对被不同的研究团队创造出来。2014年含有非天然碱基对的半合成生命的诞生更是标志着这一领域的研究正式从体外走向了体内[1, 17, 19]。然而要想真正实现在生物体内使用人造碱基对,利用人工扩展密码子编码各种非天然氨基酸,从而按需定制各种非天然功能蛋白质的长远目标,这一领域还有很长的路要走。
首先,需要设计并创造性能更为优越的人造碱基对。必须承认即便是现在最为成功的人造碱基对的复制保真性较天然碱基对而言也仍然存在着明显的差距。其次,需要进一步优化与转录、翻译相关的各种酶及工作元件。尽管在20多年前,Bernner研究组就已经利用早期人造碱基对isoC-isoG在体外实现了从复制到转录、再到翻译的中心法则整个流程。但在那之后,相对于复制,有关人造碱基对的转录尤其是翻译的研究工作就大大滞后了。
总而言之,Romesberg的成功使得合成生物学家们的目标更加明晰——就是在生物体内扩展遗传密码,提高遗传密码的多样性,进而可以根据需要定制出各种含有人工扩展碱基的功能DNA、RNA及含有各种非天然氨基酸的功能蛋白质的新型生命形式。当然,这项技术和任何技术一样(如基因工程、核物理等),既可以造福人类,也可以毁灭人类(生物武器、核武器)。技术本身并没有问题,关键是看应用技术的人的动机。希望在未来科学家可以创造出更多形式和功能的可控新型生命,造福全人类。
| [1] | Malyshev D A, Dhami K, Lavergne T, et al. A semi-synthetic organism with an expanded genetic alphabet. Nature, 2014, 509 (7500): 385-388. |
| Click to display the text | |
| [2] | Benner S A, Sismour A M. Synthetic biology. Nat. Rev. Genet.,2005, 6 (7): 533-543. |
| Click to display the text | |
| [3] | Hirao I, Kimoto M, Yamashige R. Natural versus artificial creation of base pairs in DNA: origin of nucleobases from the perspectives of unnatural base pair studies. Acc. Chem. Res., 2012, 45 (12): 2055-2065. |
| Click to display the text | |
| [4] | Matray T J, Kool E T. A specific partner for abasic damage in DNA. Nature, 1999, 399 (6737): 704-708. |
| Click to display the text | |
| [5] | Moran S, Ren X F, Kool E T. A thymidine triphosphate shape analog lacking Watson-Crick pairing ability is replicated with sequence selectivity. Proc. Natl. Acad. Sci. USA, 1997, 94 (20): 10506-10511. |
| Click to display the text | |
| [6] | Sismour A M, Benner S A. The use of thymidine analogs to improve the replication of an extra DNA base pair: a synthetic biological system. Nucleic Acids Res., 2005, 33 (17): 5640-5646. |
| Click to display the text | |
| [7] | Leconte A M, Hwang G T, Matsuda S, et al. Discovery, characterization, and optimization of an unnatural base pair for expansion of the genetic alphabet. J. Am. Chem. Soc., 2008, 130 (7): 2336-2343. |
| Click to display the text | |
| [8] | Seo Y J, Hwang G T, Ordoukhanian P, et al. Optimization of an unnatural base pair toward natural-like replication. J. Am. Chem. Soc., 2009, 131 (9): 3246-3252. |
| Click to display the text | |
| [9] | Malyshev D A, Seo Y J, Ordoukhanian P, et al. PCR with an expanded genetic alphabet. J. Am. Chem. Soc., 2009, 131 (41): 14620-14621. |
| Click to display the text | |
| [10] | Seo Y J, Matsuda S, Romesberg F E. Transcription of an expanded genetic alphabet. J. Am. Chem. Soc., 2009, 131 (14): 5046-5047. |
| Click to display the text | |
| [11] | Malyshev D A, Dhami K, Quach H T, et al. Efficient and sequence-independent replication of DNA containing a third base pair establishes a functional six-letter genetic alphabet. Proc. Natl. Acad. Sci. USA, 2012, 109 (30): 12005-12010. |
| Click to display the text | |
| [12] | Li L, Degardin M, Lavergne T, et al. Natural-like replication of an unnatural base pair for the expansion of the genetic alphabet and biotechnology applications. J. Am. Chem. Soc., 2014, 136 (3): 826-829. |
| Click to display the text | |
| [13] | Yang Z, Hutter D, Sheng P, et al. Artificially expanded genetic in formation system: a new base pair with an alternative hydrogen bonding pattern. Nucleic Acids Res., 2006, 34 (21): 6095-7101. |
| Click to display the text | |
| [14] | Yang Z, Sismour A M, Sheng P, et al. Enzymatic incorporation of a third nucleobase pair. Nucleic Acids Res., 2007, 35 (13): 4238-4249. |
| Click to display the text | |
| [15] | Chen F, Yang Z, Yan M, et al. Recognition of an expanded genetic alphabet by type-II restriction endonucleases and their application to analyze polymerase fidelity. Nucleic Acids Res., 2011, 39 (9): 3949-3961. |
| Click to display the text | |
| [16] | Leal N A, Kim H J, Hoshika S, et al. Transcription, reverse transcription, and analysis of RNA containing artificial genetic components. ACS Synth Biol, 2015, 4 (4): 407-413. |
| Click to display the text | |
| [17] | Thyer R, Ellefson J. Synthetic biology: New letters for life's alphabet. Nature. 2014, 509 (7500): 291-292. |
| Click to display the text | |
| [18] | Kim H J, Leal N A, Hoshika S, et al. Ribonucleosides for an artificially expanded genetic information system. J. Org. Chem., 2014, 79 (7): 3194-3199. |
| Click to display the text | |
| [19] | Service R F. Synthetic biology. Designer microbes expand life's genetic alphabet. Science, 2014, 344 (6184): 571. |
| Click to display the text | |
| [20] | Collins M L, Irvine B, Tyner D, et al. A branched DNA signal amplification assay for quantification of nucleic acid targets below 100 molecules/ml. Nucleic Acids Res., 1997, 25 (15): 2979-2984. |
| Click to display the text | |
| [21] | Collins M L, Dailey P J, Shen L P, et al. Branched DNA (bDNA) technology for direct quantification of nucleic acids: Design and performance. In: Ferré F, Ed. Gene quantification. Boston: Birkhäuser, 1998: 205-223. |
| Click to display the text | |
| [22] | Kolberg J A, Ludtke D N, Shen L P, et al. Branched DNA (bDNA) technology for direct quantification of nucleic acids: Research and clinical applications. In: Ferré F, Ed. Gene quantification. Boston: Birkhäuser, 1998: 327-342. |
| Click to display the text | |
| [23] | Elbeik T, Markowitz N, Nassos P, et al. Simultaneous runs of the Bayer VERSANT HIV-1 version 3.0 and HCV bDNA version 3.0 quantitative assays on the system 340 platform provide reliable quantitation and improved work flow. J. Clin. Microbiol, 2004, 42 (7): 3120-3127. |
| Click to display the text | |
| [24] | Morales J C, Kool E T. Efficient replication between non-hydrogen- bonded nucleoside shape analogs. Nat. Struct. Biol., 1998, 5 (11): 950-954. |
| Click to display the text | |
| [25] | Hirao I, Kimoto M, Mitsui T, et al. An unnatural hydrophobic base pair system: site-specific incorporation of nucleotide analogs into DNA and RNA. Nat. Methods, 2006, 3 (9): 729-735. |
| Click to display the text | |
| [26] | Leconte A M, Romesberg F E. Amplify this! DNA and RNA get a third base pair. Nat. Methods, 2006, 3 (9): 667-668. |
| Click to display the text | |
| [27] | Elbeik T, Surtihadi J, Destree M, et al. Multicenter evaluation of the performance characteristics of the Bayer VERSANT HCV RNA 3. 0 assay (bDNA). J. Clin. Microbiol, 2004, 42 (2): 563-569. |
| Click to display the text | |
| [28] | Sheng P, Yang Z, Kim Y, et al. Design of a novel molecular beacon: modification of the stem with artificially genetic alphabet. Chem. Commun., 2008, 41 (7): 5128-5130. |
| Click to display the text | |
| [29] | Yang Z, Chen F, Chamberlin S G, et al. Expanded genetic alphabets in the polymerase chain reaction. Angew Chem. Int. Ed. Engl., 2010, 49 (1): 177-180. |
| Click to display the text | |
| [30] | Kimoto M, Yamashige R, Matsunaga K-I, et al. Generation of high-affinity DNA aptamers using an expanded genetic alphabet. Nat. Biotechnol, 2013, 31 (5): 453-457. |
| Click to display the text | |
| [31] | Sefah K, Yang Z, Bradley K M, et al. In vitro selection with artificial expanded genetic information systems. Proc. Natl. Acad. Sci. USA, 2014, 111 (4): 1449-1454. |
| Click to display the text | |
| [32] | Kimoto M, Mitsui T, Yamashige R, et al. A new unnatural base pair system between fluorophore and quencher base analogues for nucleic acid-based imaging technology. J. Am. Chem. Soc., 2010, 132 (43): 15418-15426. |
| Click to display the text |

 , ,
, , 
