2. 中国科学院北京生命科学研究院 北京 100101;
3. 中国科学院青岛生物能源与过程研究所 青岛 266101
2. Beijing Institute of Life Sciences, Chinese Academy of Sciences, Beijing 100101, China;
3. Qingdao Institute of Bioenergy and Bioprocess Technology, Chinese Academy of Sciences, Qingdao 266101, China
微生物组(Microbiome)是指一个特定环境中的总的微生物群落,通过在一定环境空间的相互作用和平衡,形成了相对稳定的生态环境并具有一定的生理功能。长期以来,微生物群落被认为在营养代谢、污染物降解、维持动植物和人体生态系统平衡中发挥着关键作用,然而,对其中的作用机制并不清楚。高通量测序技术的广泛应用,为在群落水平上研究微生物的功能和作用机制开辟了新的思路,使得我们能够从全基因组角度研究自然和人体环境样品中微生物的组成和功能,为我们寻找新基因、开发新的生物活性物质、研究环境中微生物多样性和进化提供了重要手段,也使得这一领域迅速成为研究热点。海量测序数据的产生,使得微生物组学成了一门真正的大数据科学。以人体微生物组为例,它包含了数万亿个细胞,占人体总细胞的90%以上,涵盖上千个物种,至少2 000万个独特的微生物基因,其数目远远超过人的基因数目(大约2万至2.5万个基因[1])。人类微生物组项目(Human Microbiome Project, HMP)自2008年启动至2012年第一阶段结束期间,共完成5 177个16SrDNA样本,681个全基因组序列(Whole Genome Sequences,WGS)样本和3 000余个高质量的参考基因组测序[2]。然而,当测序成本已经不再成为微生物组学发展的主要限制因素时,数据分析就成了微生物组研究面对的最大挑战。
本文围绕微生物组数据管理与分析这一关键问题,分析了目前的现状和需求,总结了国内外发展趋势和问题,提出了我国微生物组数据中心建设的思考和建议。
1 微生物组数据管理和分析的现状和需求(1)在目前元基因组研究的各个过程,从样本的采集、提取、测量方法(如高通量测序技术、质谱、核磁等),到数据的分析和整合的各个环节,都缺乏标准化的协议。而元基因组数据标准不统一以及整合技术的缺失,使得不同研究课题、不同采样来源、不同数据平台的样本数据只能简单地按照采样信息对其进行汇总,而无法根据结构特征和功能进行集成和统一挖掘分析,因此也无法从大范围的数据中获得其蕴含的生物意义。
(2)微生物组数据及其分析的特点,对复杂数据的整合提出了很高的要求。微生物组研究产生了大量的复杂数据,既有对环境、样本进行描述的元数据,也有原始的测序文件,还包括格式各异的序列注释和功能研究产生的数据,由此而形成的对大规模复杂数据的组织、存储、访问、共享以及与关联数据进行整合能力的要求,也是一个巨大的挑战。此外,不同的生态系统(如肠道、土壤、海洋等)、不同结构和功能特征的数据整合和对比分析也有着非常重要的价值,能够对跨生态系统的分析、物种分布与环境因素的相互作用机制提供数据支持。
(3)微生物组数据分析缺乏高质量的参考序列。因为元基因组研究中物种识别和基因注释都依赖于已知参考基因组及相关注释信息, 即便是已经有大量系统研究的人体微生物组,也仍然有将近一半预测的开放阅读框架(Open Reading Frame, ORF)无法找到相应的相似性序列来进行功能研究[3]。相对于研究基础较多的人体微生物组,新环境下元基因组的研究更缺乏有效的实验和计算手段。目前,国际上也陆续建立起了如土壤微生物[4],发酵食品[5]等环境相关的高质量参考数据库,为功能注释提供了重要的参考,也对数据整合起到了极大的帮助。
此外,元基因组快速对比与海量数据搜索技术的缺失,爆发式增长的元基因组数据的存储和分析对成本和计算能力的需求等问题,都迫切需要通过新型硬件(如GPU等)、云计算、关联数据整合方法、高效搜索算法等相结合,提出创新解决方案。
2 国际微生物组数据平台建设情况2016年5月13日,美国政府颁布了投资5.21亿美元的“美国国家微生物组计划”,试图通过对各种不同环境中微生物生态系统的综合研究,深入揭示微生物组的组成、结构及功能,促进对健康微生物组功能的保护和恢复。截至2016年,国际上已陆续启动了由美国国立卫生研究院(National Institutes of Health,NIH)支持的HMP、由欧盟支持的人肠道微生物组(MetaHIT)等13项与人类健康相关的微生物组项目及包括“地球微生物组计划”(Earth Microbiome Project, EMP)、海洋微生物B3计划(Micro B3 Biodiversity, Bioinformatics, and Biotechnology)等在内的9个环境微生物组研究计划[6]。这些项目大多建立了完善的数据集成机制和数据管理平台,通过对人体和环境样本进行测序分析,全方位理解微生物群落的多样性及功能。
HMP是由美国国立卫生研究院支持的项目,一期从2008年至2012年,2014年起开始了第二期的研究工作。该项目的主要目标是探索人体微生物组与人类健康和疾病的关系,主要集中在呼吸道、口腔、皮肤、肠道、阴道5个方面。该项目分别在贝勒医学院(Baylor College of Medicine,BCM)和华盛顿医学院(Washington University School of Medicine)两个临床中心从242个人的身上获取上千个样本,在BCM人类基因组测序中心、麻省理工学院Broad研究所测序中心、文特研究所和华盛顿大学医学院4个测序中心进行了16S和WGS测序[7]。由于样本采集和测序都是由不同的机构分别进行的,项目开发了针对测序和数据分析的标准协议和质量控制过程。HMP项目也建立了数据分析和管理中心(Data Analysis and Coordination Center, DACC)来存储所有项目产生的16S、WGS和参考基因组序列。同时,DACC也发布新闻、通知公告、项目的统计数据,并与测序中心合作,共同进行数据的分析和注释工作。项目产生的所有数据,也同时提交到美国国立生物信息中心进行公开。
2010年8月,地球微生物组计划(EMP)正式启动,计划旨在通过对全球典型的环境样本进行宏基因组测序,包括土壤、海洋、空气、淡水等生态系统,从而全方位地分析微生物群落的多样性及其功能。项目在设立之初,就将建立一个用以解决地球生态系统基础问题的集成样本、基因、蛋白质的数据库作为3个主要目标之一[8]。为了实现对元数据和数据的质量控制,EMP项目推荐使用基因组最小数据规范(Minimum Information about a Genome Sequence Specification, MIGS)[9]和环境序列最小数据规范(Minimum Information about an ENvironmental Sequence specification,MIENS)[10]作为数据标准,并且定义了关于元数据、DNA提取、16S、18S、ITS等不同测序目标的标准和协议[11]。项目产生的数据通过定量微生物生态系统数据库(Quantitative Insights into Microbial Ecology database,QIIME)进行管理和共享。截至2014年8月,项目已经有超过200个合作者提供数据,样本覆盖超过40种不同的生态环境[12]。
除了项目建立的数据中心,一些主要的测序和研究机构也建立了微生物组的数据平台。其中由美国能源部联合基因组研究中心建立的整合微生物基因组(Integrated Microbial Genomes, IMG)[13]和阿贡国家实验室建立的Metagenome-RAST(MG-RAST)平台[14],目前得到了较为广泛的应用。
IMG平台支持美国能源部联合基因组研究中心进行测序的数据进行注释、分析和管理, 逐步对全球科学家免费开放。在数据标准方面,IMG及其数据管理平台Genome Online[15](图 1)都使用国际基因组标准委员会(Genomic Standards Consortium,GSC)[16]领导制定的一系列关于环境测序样本描述的最小数据集,因此,整合的数据能够按照生态系统、环境、宿主或工程改造进行分类组织。此外,平台目前还提供一系列的对基因组和元基因组数据的分析工具。
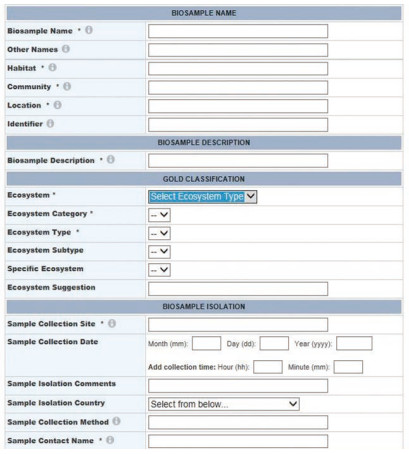
|
| 图 1 IMG数据管理平台 |
MG-RAST主要目的是为用户提供基于高性能计算资源的元基因组数据的系统发育和功能注释等分析流程,对于非生物信息学专业的用户来说,可以简单地通过一个工作流(图 2),得到元基因组数据关于注释的基本信息。同时,MG-RAST也提供了数据管理的平台,用户可以对自有的元数据和序列文件进行管理,并且可以选择公开或者对数据保持私有。
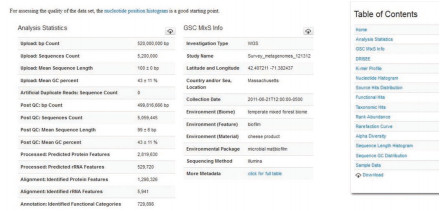
|
| 图 2 MG-RAST通过自动化的分析工具形成的分析结果 |
因此,可以看到在国际上,尤其是美国,已经在微生物组研究及其数据分析方面具有了比较好的工作基础,形成了有一定影响力的数据管理和分析平台,能够对大型测序项目的数据进行有效管理,并通过相对统一的标准和质量控制程序,来保证数据产生的质量。然而,它们也仍然存在一系列的问题,如数据中心主要以基因组和元基因组数据为主,缺乏与其他数据的整合;以纵向数据整合为主,跨领域和不同生态系统的数据集很少;同时也缺乏高质量的参考数据集、高效的计算资源及快速的数据分析平台等[17]。
3 我国微生物组数据平台建设的现状及需求中国一方面积极参与了国际EMP计划,另一方面,早在21世纪初,中科院微生物所有关专家就开始推动“微生物地球”研究计划;2014年,中科院组织并启动了土壤微生物相关的先导专项研究计划。中国科学家已经在人体微生物组、酿造微生物组、微生物数据资源等方面,取得了很好的成绩。从论文的发文量来看,中国已仅次于美国,居于全球第二位,但是与第一名美国,还有较大的差距(图 3)。
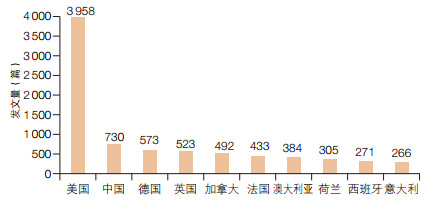
|
| 图 3 2010—2015年全球微生物组论文发文量排名前十的国家 (数据来源: Web of Sciences) |
以中科院为核心的团队,在微生物组的数据平台建设和数据分析方面,具有较好的基础。在以微生物为核心的数据平台建设方面,落户于中科院微生物所的世界微生物数据中心是我国生命科学领域第一个世界数据中心。中科院微生物所马俊才团队建立的全球微生物资源目录数据平台(Global Catalogue of Microorganism, GCM),目前集成了来自美国、法国、德国、荷兰等43个国家110个国际微生物资源保藏机构,超过30万的微生物实物资源的详细信息,其中不乏来自特殊生态环境、具有重要的科研和工业应用价值的微生物[18]。此外,马俊才团队还建立了食源性病原微生物、极端环境微生物等高质量基因组参考数据库,整合了海量国际微生物组数据和分析工作流,形成了一个基于云环境的微生物组分析系统。最近,中科院北京生科院赵方庆团队建立了RiboFR-seq[19]、metaSort[20]、inGAP-sf[21]和inGAP-CDG[22]等多种微生物组学研究的新技术和新方法,这些工具分别针对微生物组分析中的拼接、序列归类和注释,以及微生物间相互作用等问题,为高效解读微生物组提供了全新的技术手段。中科院青岛生物能源与过程所苏晓泉、宁康等团队开发了元基因组高性能计算分析软件Parallel-META 3[23]以及元基因组比较算法Meta-Storms[24]和GPU-Meta-Storms[25],能够深入、全面、快速地将数量庞大的未知微生物组进行结构与功能解析,从而允许从大数据的角度剖析疾病或生态灾害下微生物组的变化规律。中科院青岛生物能源与过程所徐健等团队提出了“拉曼组”(Ramanome)与“元拉曼组”(Meta-ramanome)的概念,能够在单个微生物细胞精度、非标记式、快速表征与测量细胞群体或群落的状态与功能;它们与元基因组等“基因型”数据有着本质区别,与元转录组、元蛋白组和元代谢组等现有“表型”数据相比,在单细胞精度、非破坏性、通量和成本等方面也具不可替代的优势,代表着一种崭新的微生物组大数据类型。
然而,我国微生物组相关研究的数据资源散落于各实验室,尚无国家层面的微生物组数据库体系和数据管理机制,同时在数据管理中还存在着标准不统一、数据产出和数据分析脱节、数据集成和保存困难、分析技术与方法不完善、数据深度挖掘技术缺乏等问题,缺乏高效、稳定、可用的计算平台,无法从海量数据中发现有价值的生物学信息,严重阻碍着微生物组技术的发展与应用。
4 思考与建议数据资源是微生物组研究的关键,更是重要的战略资源。与基因组研究相比,微生物组研究在国际上处于起跑阶段。应立足于我国微生物组研究的现状,解决微生物组数据管理和分析的关键问题,并逐步形成自己的核心优势。特提出以下建议:
(1)构建微生物组数据标准化及数据管理系统。建立一套完整的微生物组研究的技术标准(样本采集、保存、数据产出、分析、质量控制)及管理规范和机制(数据共享、存储、知识产权等);实现标准化的数据接口和存储方案、标准化的分析方法和流程、标准化计算、存储方案的评价体系和标准化数据安全及分级体系。在此基础上,开发微生物组数据管理系统,逐步整合国内相关研究产生的人体、环境、工农业等微生物组数据资源,实现对我国微生物组数据资源的有效管理和高效集成。
(2)建立微生物组大数据计算、存储和共享平台。搜集和整理海量公共微生物组数据,整合样本的多组学信息,实现微生物组大数据的广泛、深层次整合;建立高质量的微生物组参考数据库;实现高效的大数据搜索与相似度分析算法的开发;建立高效的微生物组数据处理流程,实现对微生物组数据的系统管理、高效分析及整合利用。
(3)开发微生物组大数据挖掘的新方法。建立适合元基因组物种谱注释和全基因组序列拼接方法,开发基于降低物种复杂度策略的宏基因拼接和序列归类算法,建立基于多序列联配的远缘元基因组数据的功能注释方法,发展基于菌群结构和功能相似性的微生物组大数据搜索引擎,并结合人工智能发展针对慢性疾病和生态灾害的微生物组诊断和预警技术。开发适用于高性能计算平台的数据处理方法,实现大规模数据及分析结果的可视化。
(4)加强以我为主的国际合作。以数据平台为基础,参与国际标准的制定,积极引领满足国家重大需求的国际微生物组数据合作计划,形成更大范围的数据共享体系,提升我国在微生物组研究领域的国际影响力和贡献度。
各国已将微生物组研究置于空前重要的位置,并形成比较完善的工作基础。我国在微生物资源、测序能力等方面具有显著优势,但在微生物组大数据的收集、存储、功能挖掘、开发利用等关键技术上,仍存在诸多薄弱环节,这也是制约我国微生物组研究的关键问题。因此,我们建议适时启动“中国微生物组”计划,建立中国微生物组数据中心,实现我国微生物组数据资源的系统管理和高效利用。
| [1] | Grice E A, Segre J A. The Human Microbiome:our second Genome. Annu Rev Genomics Hum Genet, 2012, 13(1): 151–170. DOI:10.1146/annurev-genom-090711-163814 |
| [2] | Gevers D, Knight R, Petrosino J F, et al. Human Microbiome Project Consortium:A framework for human microbiome research. Nature, 2012, 486(7402): 215–221. DOI:10.1038/nature11209 |
| [3] | Kurokawa K, Itoh T, Kuwahara T, et al. Comparative metagenomics revealed commonly enriched gene sets in human gut microbiomes. DNA Res, 2007, 14: 169–181. DOI:10.1093/dnares/dsm018 |
| [4] | Choi J, Yang F, Stepanauskas R, et al. Strategies to improve reference databases for soil Microbiomes.The ISME Journal, 2016, 1-6. |
| [5] | Almeida M, Hébert A, Abraham A L, et al. Construction of a dairy microbial genome catalog opens new perspectives for the metagenomics analysis of dairy fermented products. BMC Genomics, 2014, 15: 1101. DOI:10.1186/1471-2164-15-1101 |
| [6] | Stulberg E, Fravel D, Proctor L M, et al. An assessment of US microbiome research. Nat Biotechnol, 2016, 1(1): 15015. |
| [7] | Lita M P. The National Institutes of Health Human Microbiome Project. Seminars in Fetal & Neonatal Medicine, 2016, 21(6): 368–372. |
| [8] | Gilbert J A, Meyer F, Jansson J, et al. The Earth Microbiome Project:Meeting report of the "1st EMP meeting on sample selection and acquisition" at Argonne National Laboratory. Standards in Genomic Sciences, 2010, 3(3): 249–253. DOI:10.4056/aigs.1443528 |
| [9] | Field D, Garrity G, Gray T, et al. The minimum information about a genome sequence (MIGS) specification. Nat Biotechnol, 2008, 26(5): 541–547. DOI:10.1038/nbt1360 |
| [10] | Yilmaz P, Kottmann R, Field D, et al. The "Minimum Information about an Environmental Sequence" (MIENS) specification. Nat Biotechnol, 2011, 29: 415–420. DOI:10.1038/nbt.1823 |
| [11] | EMP.[2016-12-3]. http://www.earthmicrobiome.org/empstandard-protocols/. |
| [12] | Gilbert J A, Jansson J K, Knight R, et al. The Earth Microbiome project:successes and aspirations. BMC Biol, 2014, 12(1): 69. DOI:10.1186/s12915-014-0069-1 |
| [13] | Chen I A, Markowitz V M, Chu K, et al. IMG/M:integrated genome and metagenome comparative data analysis system. Nucleic Acids Res, 2017, 45(D1): D507–D516. DOI:10.1093/nar/gkw929 |
| [14] | Meyer F, Paarmann D, D'Souza M, et al. The metagenomics RAST server-a public resource for the automatic phylogenetic and functional analysis of metagenomes. BMC Bioinformatics, 2008, 9(1): 386. DOI:10.1186/1471-2105-9-386 |
| [15] | Mukherjee S, Stamatis D, Bertsch J, et al. Genomes OnLine Database (GOLD) v.6:data updates and feature enhancements. Nucleic Acids Res, 2017, 45(D): D446–D456. |
| [16] | Field D, Sterk P, Kottmann R, et al. Genomic Standards Consortium Projects. Standards in Genomic Sciences, 2014, 9(3): 599–601. DOI:10.4056/sigs.5559680 |
| [17] | Kyrpides N C, Eloe-Fadrosh E A, Ivanova N N. Microbiome data science:understanding our microbial planet. Trends Microbiol, 2016, 24(6): 425–427. DOI:10.1016/j.tim.2016.02.011 |
| [18] | Wu L, Sun Q, Desmeth P, et al. World data centre for microorganisms:an information infrastructure to explore and utilize preserved microbial strains worldwide. Nucleic Acids Res, 2017, 45(D): D611–D618. |
| [19] | Zhang Y, Ji P, Wang J, et al. RiboFR-Seq:a novel approach to linking16S rRNA amplicon profiles to metagenomes. Nucleic Acids Res, 2016, 44(10): e99. DOI:10.1093/nar/gkw165 |
| [20] | Ji P, Zhang Y, Wang J, et al. MetaSort untangles metagenome assembly by reducing microbial community complexity. Nat Commun, 2017, 8: 14306. DOI:10.1038/ncomms14306 |
| [21] | Shi W, Ji P, Zhao F. The combination of direct and paired link graphs can boost repetitive genome assembly. Nucleic Acids Res, 2016. DOI:https://doi.org/10.1093/nar/gkw1191. |
| [22] | Peng G, Ji P, Zhao F. A novel codon-based de Bruijn graph algorithm for gene construction from unassembled transcriptomes. Genome Biol, 2016, 17(1): 232. DOI:10.1186/s13059-016-1094-x |
| [23] | Jing G, Sun Z, Wang H, et al. Parallel-META 3:Comprehensive taxonomical and functional analysis platform for efficient comparison of microbial communities. Sci Rep, 2017, 7: 40371. DOI:10.1038/srep40371 |
| [24] | Su X, Xu J, Ning K. Meta-Storms:Efficient Search for Similar Microbial Communities Based on a Novel Indexing Scheme and Similarity Score for Metagenomic Data. Bioinformatics, 2012, 28(19): 2493–2501. DOI:10.1093/bioinformatics/bts470 |
| [25] | Su X, Wang X, Jing G, et al. GPU-Meta-Storms:Computing the structure similarities among massive amount of microbial community samples using GPU. Bioinformatics, 2014, 30(7): 1031–1033. DOI:10.1093/bioinformatics/btt736 |



