2. 中国科学院植物生理生态研究所 合成生物学重点实验室 上海 200032
2. Key Laboratory of Synthetic Biology, Institute of Plant Physiology & Ecology, Shanghai Institutes for Biological Sciences, Chinese Academy of Sciences, Shanghai 200032, China
自然科学是研究自然现象和自然界各种物质形态、结构、性质及其运动和发展的各门学科的总称,包括天文学、物理学、化学、地学和生物学等。数学因其研究对象的特殊性(从自然到社会的一切“数”的问题的抽象)和思维逻辑的特殊性(形式逻辑的“自洽”),不属于自然科学范畴,却是各门学科的重要基础。物质存在和运动的状态可以从空间、时间两个维度来观测和研究,因此,自然科学各学科也可以在这两个维度上“划分”各自的领域。
与各门学科一样,生物学起步于观测和描述。由于生命现象自身的复杂性,19世纪末,虽然人们在以往对生物及生物运动(如,生长、生殖、发酵等)观测与研究的基础上,开始对细胞形态结构的认识、对遗传规律的抽象、对生物进化概念的理解上有了革命性的突破,但生物学尚未能实现在整体上向研究其普遍的构成本质及运动规律的“生命科学”的飞跃。20世纪上半叶,微观层面的生物化学、细胞生物学和发育生物学,宏观层面的生态学和进化生物学等的形成与发展,推动生物学从以分类描述为特征的传统科学向以机制研究为核心的现代科学转变。以此为基础,20世纪中叶以来,生命科学研究在技术、认知和能力方面接连爆发了“分子生物学”“基因组学”和“会聚”研究三次革命。
“生命是什么?生命如何起源,又如何演化?”这既是涉及人类健康生存与繁衍、社会发展与和谐的基本科学问题,也是人类每个文明体系都必须回答的哲学问题。尽管以还原论为主导的现代生命科学技术,特别是以DNA双螺旋结构解析为基础的分子遗传学研究,确定了生命运动的“中心法则”(central dogma),又通过以基因组全面解析以及相关“组学”数据为基础的系统生物学分析,对生物体组成和生命运动规律的认识达到了前所未有的深度和精度,但是对生命起源、演化(进化)以及生命本质的认识还远远不够。21世纪初,工程学思想策略与现代生物学、系统科学及合成科学的融合,形成了以采用标准化表征的生物学部件,在理性设计指导下,重组乃至从头合成新的、具有特定功能的人造生命为目标的“合成生物学”。合成生物学的崛起,突破了生物学以发现描述与定性分析为主的所谓“格物致知”的传统研究范式,为生命科学提供了“建物致知”的崭新研究思想,开启了可定量、可计算、可预测及工程化的“会聚”研究新时代。它不仅将人类对生命的认识和改造能力提升到一个全新的层次,也为解决与人类社会相关的全球性重大问题提供了重要途径。
1 合成生物学的内涵合成生物学区别于其他传统生命科学的核心,是其“工程学本质”,主要体现在两个方面。①其“自下而上”的正向工程学“策略”。因此,元件标准化→模块构建→底盘适配,包括对生命过程的途径、网络的组成及其调控的认识及“正交化生命”的设计与构建,是其最核心的研究内容;而人工线路(包括新一代的“代谢工程”)的构建,就是其最重要的工程化平台。②目标导向的构(重构)建(建造)“人造生命”。因此,“自上而下”地构建“最小基因组”或“自下而上”地合成“人工基因组”,是合成生物学另一个最核心的研究内容;大片段基因组操作和改造,以及大规模、高精度、低成本DNA合成,是其最重要的两大使能技术;而基因组(包括原型细胞的合成等“细胞工程”)的构建,是其最重要的工程化平台。这两个方向,基本抓住了合成生物学的工程化研究所包含的“定量生物学”“分子生物学”与“系统生物学”等会聚来源的本质,原则上是比较全面和准确的;当然,我们也可以在下述3个方向上更具体地阐述其内涵(图 1)。
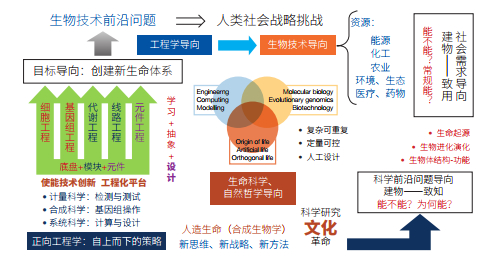
|
| 图 1 合成生物学的内涵 |
合成生物学内涵的核心,是它的“会聚特性”。合成生物学会聚了科学研究带来的“发现能力”,工程学理念带来的“建造能力”以及颠覆性技术带来的“发明能力”,从而全面提升社会的“创新能力”。合成生物学的工程学内涵就是,在人工设计的指导下,采用正向工程学“自下而上”的原理,对生物元件进行标准化的表征,建立通用型的模块,在简约的“细胞”或“系统”底盘上,通过学习、抽象和设计,构建人工生物系统并实现其运行的定量可控。
1.2 生物技术内涵合成生物学是在分子水平上对生命系统的重新设计和改造,线路工程、基因组工程、细胞代谢工程等是其核心的技术与工程展现。因此,在一定意义上,可以认为合成生物学也就是生物技术在基因组和系统生物学时代的延伸。当然,这种延伸有质的飞跃。一方面,合成生物学将原有的生物技术上升到系统化和标准化的高度,有可能把生物技术推向平台化的工程生物学层次。另一方面,在全基因组和系统生物学基础上创建新生命体系的工程化生物技术,不仅能完成传统生物技术难以胜任的任务,还将在学科交叉和技术整合的基础上,孕育技术创新的飞跃。这就是合成生物学的生物技术内涵。
1.3 科学内涵合成生物学从其发端到现在的实践乃至将来的发展,还有另一层重要的内涵,就是与“自上而下”的系统生物学相辅相成,从“合成”的理念和策略出发,突破生命科学传统研究从整体到局部的“还原论”策略,通过“从创造到理解”的方式,开启理解生命本质的新途径,建立生命科学研究的新范式。这就是合成生物学的科学内涵。
2 合成生物学学科发展及成果合成生物学,从20世纪初,由合成科学而得以“隐喻”(metaphor)[1],到由分子生物学的科学知识与DNA重组技术突破成为“愿景”(vision)[2],直至现代在基因组蓝图基础上、工程学理念介入而“定名” [3],进而成为“会聚”研究的标志,大体经历了3个阶段[4]。第一阶段,创建时期(2000—2003年):产生了许多具备领域特征的研究手段和理论,特别是基因线路工程的建立及其在代谢工程中的成功运用。第二阶段,扩张和发展期(2004—2007年):这一阶段的特征是领域有扩大趋势,但工程技术进步比较缓慢。第三阶段,快速创新和应用转化期(2008—2013年):这一阶段涌现出的新技术和工程手段使合成生物学研究与应用领域大为拓展,特别是人工合成基因组的能力提升到了接近Mb(染色体长度)的水平,而基因组编辑技术出现前所未有的突破(图 2)。
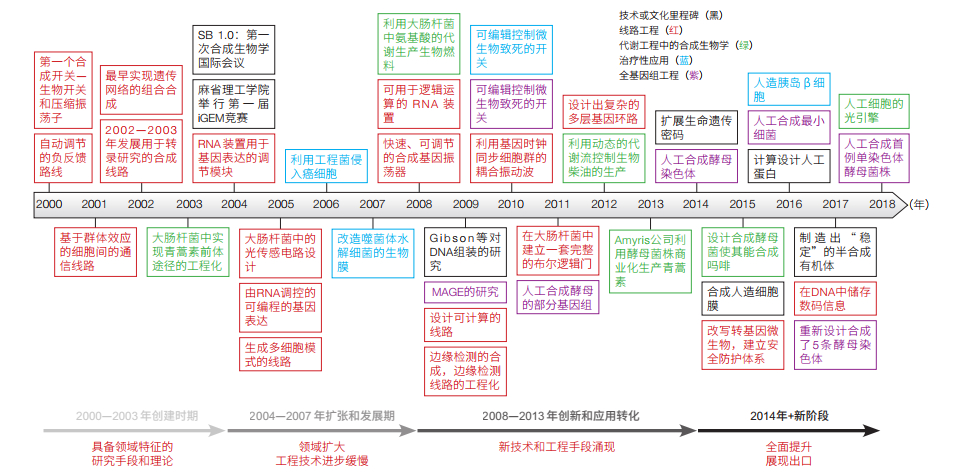
|
| 图 2 2000—2018年合成生物学研究的代表性进展 |
在此基础上,2014年,美国科学院提出“会聚”研究为生命科学第三次革命[5],以使能技术的工程化平台建设与生物医学大数据的开源应用相结合带来的“工程生物学”(Engineering Biology),正在全面推动生物技术、生物产业和生物医药“民主化”发展的新阶段,也可以说,是在实现人类“能力提升”的宏伟目标上迈出了坚实的一步。
本期《中国科学院院刊》专刊“合成生物学:回顾与展望”所刊载的各篇综述,简要介绍了近20年来合成生物学在元件工程、线路工程、代谢工程、基因组与细胞工程等方面的进展,特别是相关使能技术的重要突破;进而专门描述了承载工程化研究与技术服务的实验室与中心(设施)的建设情况。最后,阐述了合成生物学在医药、化工、农业、环保等领域应用的重要进展以及保障合成生物学科技与产业发展的相关政策与管理。
2.1 元件工程合成生物学按照工程学的理念,将生命体系中发挥功能的最简单、最基本的单元,统称为生物元件(biological part),鉴于其生物大分子本质,氨基酸序列(蛋白质的一级结构)或者核苷酸序列(核酸的一级结构)以及在序列上相关的修饰应该是生物元件最基本的构成。当今“合成生物学”概念的提出,就是基于生物“元件”构建人工逻辑“线路”的创新工作。在此基础上,形成了合成生物学中“生物元件”的定义——“遗传系统中最简单、最基本的生物积块(BioBrick),是具有特定功能的氨基酸或者核苷酸序列,可以在更大规模的设计中与其他元件进一步组合成具有特定生物学功能的生物学装置(Device)” [2, 6]。随着合成生物学研究领域的拓展,生物元件的内涵已经不再能局限于原先的“遗传系统”,也不仅仅服务于利用“模式系统”以构建线路工程。“生物元件”中非核酸部分(特别是非DNA部分,大部分就是蛋白质多肽)在一般的生物学体系中,特别是在细胞体系中,基本都是由“基因”即DNA序列所编码的。因此,“生物元件”中核酸序列的很大部分,就是编码蛋白质(其中既有发挥调控功能的蛋白因子,又有发挥结构功能的结构蛋白,还有大量具有催化功能的酶)的基因。
目前,生物元件主要来源于自然界,基于生物全基因组或转录组测序和信息挖掘的生物元件的筛选与鉴定是其研究主流。通过对基因组中的功能蛋白、转录和翻译特征序列分析,可以得到丰富的启动子、核糖体结合位点、蛋白质编码序列以及终止子等生物元件资源。在“调控元件”预测方面,已经有FPROM、TSSG、SCOPE等软件;在“功能元件”预测方面,也有Pfam等用于比对蛋白质家族和结构域的数据库。然而,从DNA序列到其编码元件的功能解读和利用还存在巨大鸿沟,生物合成功能元件的鉴定往往非常困难且效率低。以紫杉醇在红豆杉中合成步骤及相关“生物元件”的挖掘为例,从1997年揭示紫杉醇生物合成的第一步反应[7],到现在已经对后续步骤中涉及的8个细胞色素P450单加氧酶、5个酰基/芳香基转移酶以及1个氨基酸变位酶进行了解析,但仍有5个基因功能有待研究[8]。这种关键生物元件的缺失直接导致了紫杉醇的合成生物学制造尚未实现。
为实现对目标生物器件或生物系统的预测、设计、构建与优化,有必要对现有生物元件的结构与功能进行改造。由美国科学家建立的蛋白质的定向进化技术(获得2018年诺贝尔化学奖)目前仍然是生物元件改造的主要策略。构建各种不同强度的启动子文库也是实现基因精确调控的有力工具,典型例子是在工程化的产番茄红素菌株的优化过程中,对酿酒酵母的TEF1启动子改造,建立突变启动子库,得到一系列强度不同的突变启动子[9],并在此基础上进一步研究其中11个启动子特性,发现这些突变启动子活性是野生型的8%—120%[10]。此外,通过改造大肠杆菌核糖体,可以利用大肠杆菌深入研究核糖体的机制,研究抗生素和核糖体的相互作用;如果进一步扩展细胞的遗传编码方式,可以用这些工程改造的核糖体来合成新的多聚物,还可能将细胞转化成多用途的“细胞工厂” [11]。
当然,更为吸引人及更具有挑战意义的是设计合成自然界不存在的元件,例如,剑桥大学利用人造遗传物质合成出世界上第一个人工酶[12]。随着高性能计算技术、量子力学和分子动力学理论及方法学的发展,计算蛋白质设计技术在核心元件酶催化设计方面发挥出巨大作用[13],使酶工程迎来发展新阶段。例如,利用计算迭代方法从非活性蛋白质支架HG-1为起始点计算,获得的8种设计酶均表现出显著的催化活性,大幅提高了计算设计酶分子的成功率[14]。最近,中国科学研究人员利用密码子扩展方法,改造一个28 kD的荧光蛋白,成功模拟了天然光合作用系统的光能吸收,并将二氧化碳还原形成一氧化碳,为利用人工光合作用途径、最终实现建立的能源获取方式跨出了关键一步[15]。
合成生物学“自下而上”的正向工程本质决定了建立元件库的必然性和至关重要性。在应用实践中,合成生物学的定量预测、精准化设计、标准化合成与精确调控技术能力的提升,依赖于合成生物学工程化平台和标准元件库的支撑,以此推动实现利用合成生物学手段规模化高效率地解决生物工程问题,回应社会需求。2012年建立的BioBricks™元件库,第一次从法律层面允许个人、公司及科研院校制作标准化生物元件,并在相关的协议架构下进行免费共享。当然,在这个方向上,还有很长的路要走。
2.2 线路工程合成生物学学科形成的标志性工作就是人工基因线路的设计与合成。利用成熟表征的基因元件,按照电子工程学原理和方式设计、模拟,构建简单的、可被调控的基因线路模块。这些简单基因线路可被相对应的简单数学模型描述并利用环境信号加以调控,应用这样的模型,研究人员能够对其模块设计方式进行评估并可重设计、重合成,实现优化。2000年,Gardner等[16]构建的基因拨动开关,是构建具备设计功能的工程基因线路的开创性工作。Elowitz和Leibler[17]设计的振荡器,利用3个基因模块彼此间的抑制和解抑制作用实现输出信号的规律振荡。Weiss和Basu[18]建立了工程转录逻辑门的方法,并为线路的语言设计作出了重要贡献。通过基因线路可以了解原核、真核生物基因表达和分子噪声之间的关系,这也体现了合成生物学能够帮助人们深化对基础生物学的认识。合成生物学1.0会议以后,提出通过构建组合型基因线路,以提高生物系统的工程化水平的目标[19]。一些有关大肠杆菌信号线路和元件设计的研究,已将合成线路设计的范围从以转录调控为主,扩大到转录后和翻译调控[20]。通过设计出群体感应线路,开始用于多细胞模式的构建[21]。
工程设计和构建方法的不断优化,加速了线路工程的发展进程。在大肠杆菌中构建的快速、具有鲁棒性、可持续振荡的基因振荡子[22],是振荡线路设计和理论研究方面的重大突破。哺乳动物细胞振荡子的合成,首次在哺乳动物细胞中实现了对基因表达的周期性调控[23]。合成具有计数功能的基因线路,利用重组酶介导的DNA重排形成永久记忆,这是线路工程长期以来的一个目标[24]。在这期间,基于RNA的线路工程也不断发展,生物传感为RNA运算提供了方法,构建出可用于对基因表达的逻辑进行调控的RNA器件[25]。
近几年来,模块、线路设计能力不断提升,以单基因簇为单位对基因逻辑线路进行优化,可将原核生物的基因线路移植到真核细胞中[26];利用细胞之间的信号转导机制来调节多种细胞的基因表达,实现了双信号偶联的正、负反馈循环[27]。合成线路赋予细胞更强大的功能,有力促进了蛋白质线路在生物技术领域的应用[28];Andrews等[29]通过定量手段在细胞内设计可组合的具有反馈回路的时序逻辑,代表着在细胞内执行高级计算的研究迈出了关键一步。
2.3 代谢工程代谢工程研究的主要目的是通过对底盘(体外—分子机器;体内—细胞工厂)代谢途径或网络的设计、改造、构建,使其能够产生符合人类要求的产物,并逐步提高其效率。由于细胞代谢网络的复杂性,很难从上千个代谢基因及其调控线路中找到合适的改造靶点,而通过对大规模代谢网络的计算分析,设计出特定生物产品的最优合成途径,可以帮助人们找出合适的代谢工程改造策略,更快地得到适合的菌株。最具代表性的工作就是对青蒿素前体途径的工程化优化构建(包括元件的适配)[30, 31],最终形成优化的酵母青蒿酸合成途径,并授权赛诺菲(Sanofi)公司生产,成为合成生物学应用于实践的一个重要里程碑[32]。2015年,研究人员又在酵母中实现阿片类药物全合成,这是目前在微生物中构建的最长的植物天然化合物代谢途径[33]。
代谢工程已达到构建可预测合成途径模型的水平,能够利用有关宿主细胞代谢体系的信息,结合所有已知、预测的酶功能信息,来确定感兴趣的代谢途径。通过基因组挖掘获得的外源酶功能信息,对模型途径进行正向工程改造,填补宿主细胞代谢体系的空白。多家团队在大肠杆菌底盘基础上,改变其氨基酸生物合成途径,成功地产出异丁醇[34, 35]、脂肪酸类生物柴油[36]、汽油[37],以及生物塑料1, 4-丁二醇[38]。研究人员还将合成调控通路整合到生产株系中,实现了代谢途径随代谢中间产物或环境条件的动态调控[39]。当然,如何使工程线路在与细胞系统的互作中进行代谢流的动态调控,仍然是代谢工程要面对的挑战之一[40]。
随着我国在能源材料、环境生态和人民健康方面的需求不断增长,迫切期望利用合成生物学技术提高代谢工程的效率。一方面,在过去几年中,以中国科学院天津工业生物技术研究所为代表,在我国生物工程方面积累的基础上,迅速形成了一批可用于工业转化的代谢工程的研发成果。另一方面,结合中医药学的丰厚积累及我国在天然药物开发方面形成的化学生物学基础,我国科学家近年来在植物天然化合物的细胞工厂合成方面也取得重要进展。一批药食用天然化合物(如萜烯类、甾体类、黄酮化合物、抗生素等)的器件挖掘、集成及异源合成与调控均有重要突破,打通了从珍稀植物基因组测序、基因挖掘到重组合成的通道,获得不同性能的重组细胞。在酵母细胞工厂中实现了从葡萄糖分别到稀有人参皂苷CK、Rh2、Rg3、F1和Rh1的从头合成并通过优化使产量有了突破性进展,其中一部分正进入药物开发研究[41-43]。此外,创建了全新的多酚类氨基酸衍生药物的生物合成路径,实现了丹参素的高效生物合成(7.1 g/L)[44];突破了从简单糖到甾体激素前体的生物合成,实现了7-脱氢胆固醇(49.9 mg/L)、菜油甾醇(355 mg/L)等多个甾醇药物前体的高效合成。上述工作,不仅具有从微生物到动植物天然化合物的“新草本”创新性特色,而且具有生物学与药物化学及生理病理学结合的“生物医药”转化性特色,代表了我国代谢工程发展的重要方向。
2.4 基因组与细胞工程“人造生命”的合成是合成生物学发展史上的里程碑式事件,也为合成生物学的大规模发展奠定了最基本的使能技术基础。前者的代表性案例可追溯至20世纪60年代中国完成具有生理活性的牛胰岛素的全人工合成,而后者则可经典地追溯至DNA重组技术(包括原核与真核的基因克隆技术)的成功引发著名波兰遗传学家Waclaw Szybalski于1974—1978年数次提出的“合成生物学”愿景。20世纪90年代以来,基因组测序注释技术的突破,原则上实现了“读”基因组的可能性,自然也衍生出“设计”的可能性;各类定向性的DNA突变、扩增及克隆技术(乃至近年来的编辑技术)原则上实现了对基因组“编”即改造或重编程的可能性;而DNA的大规模合成与组装及构建能力的提高,则在原则上实现了对基因组“写”即合成的可能性。于是,以合成基因组及对基因组编辑为目标的基因组工程以及与此相关联的细胞工程自然成为过去20年中,合成生物学最为紧迫也最为受挑战的任务之一。
早在2002年,研究人员就用化学方法合成了与脊髓灰质炎病毒基因组RNA互补的cDNA,使其在体外RNA聚合酶的作用下转录成病毒的RNA,最终重新装配成具有侵染能力的病毒[45];此后又通过寡核苷酸的合成与逐步组装,得到一个全化学合成的φX174噬菌体基因组后,实现了支原体之间基因组DNA转移和支原体基因组人工合成技术的突破[46]。2010年,Gibson等[47]设计、合成和组装了1.08 Mb的蕈状支原体基因组,并把它移植到山羊支原体受体细胞中,创造了世界上第一个仅由人工化学合成染色体控制的、具自我复制能力的“新细胞——Synthia”。同年,Gibson等[48]通过与酶和化学试剂的混合物相结合,首次化学合成了小鼠线粒体基因组。此后,研究人员又使用基因组合成方法化学合成了2条酿酒酵母染色体臂,这是世界上首次成功合成真核生物的部分基因组[49]。从2011年开始,来自世界多个国家的研究人员开始实施第一个真核生物基因组合成计划——合成酵母基因组计划(Sc2.0),并在2014年成功合成酵母染色体synIII[50],尽管合成的仅仅是酿酒酵母16条染色体中最小的一条,但这是通往构建一个完整的真核细胞生物基因组的关键一步,特别是建立了利用计算机辅助设计染色体序列的技术。2017年3月,参与Sc2.0研究的各国科学家完成了2、5、6、10和12号染色体的合成与组装,在真核生物基因组设计与化学合成方面取得重大突破[51]。2018年,我国科研人员充分利用CRISPR-Cas等基因编辑使能技术及合成生物学的“设计—合成—检测”的工程学理念,成功实现了单染色体啤酒酵母细胞的人工创建,是合成生物学基因组工程与细胞工程方面的里程碑式突破;它不仅为人类对生命本质的研究(即“真核生物能不能以一条染色体编码基因组”的科学问题)开辟了新方向,也为研究人类端粒功能及细胞衰老提供了很好的模型[52]。
2.5 从使能技术创新到工程化平台建设颠覆性使能技术是支撑合成生物学发展的关键,而DNA合成以及高效基因组编辑技术都是其核心使能技术。现有基因合成的主流方法是基于寡核苷酸合成仪来合成寡核苷酸,然后在此基础上利用PCR等手段来进行基因合成;该技术的工程化使合成通量大幅度提高,催生了众多生物公司开展基因合成业务,合成价格也因此极大降低。但是,为进一步降低超长序列(如基因组)合成的成本,不少团队正在研发基于芯片法合成高精度寡核苷酸池,配以不同的拼接手段实现最后拼接的方法。除化学合成寡核苷酸的方法外,科学家也在探索利用末端转移酶(terminal deoxynucleotidyl transferase, TdT)直接快速合成DNA,有希望直接合成比目前方法长10倍的DNA链[53],且不需要使用毒性化学物。当然,随着DNA合成价格的降低,基于DNA的信息存储也将是未来一个很有前景的发展方向。
科学家一直在探索实现对基因组(特别是高等生物基因组)的精准编辑,曾研发了锌指核酸酶(Zincfinger nucleases, ZFN)和转录激活因子样效应因子核酸酶(transcription activator-like effector nucleases, TALEN)等方法。由于CRISPR系统的高效、方便、廉价等优点,这两种方法在CRISPR系统发展起来之后被逐渐淘汰。从2012年起,科学家利用CRISPR-Cas体系的可编程和精准切割等特点陆续发展了一系列基因组编辑的工具[54],其宿主范围目前已经覆盖了从细菌到高等生物,而且还在不断增加中。
CRISPR-Cas介导的基因组编辑的基本原理是利用向导RNA介导Cas蛋白在特定的靶标序列处引起dsDNA的断裂,然后利用同源重组方法进行精准的DNA序列替换或利用非同源末端连接方法进行靶标基因的中断。在此基础上,一系列衍生方法得以发展,如利用只切割一条链的Cas9切口酶(Cas9-nickase)突变体连组合来降低基因组编辑的脱靶率[55]。最近,基于CRISPR发展起来的单碱基编辑器在不造成靶标DNA断裂的情况下,通过对特定的碱基进行脱氨来进行基因组的精确编辑,被认为有较好的发展前景[56]。此外,利用失活的Cas蛋白还可以进行靶标基因的转录调控、表观遗传修饰研究和基因组的成像等[57]。值得一提的是,除了基因组编辑外,Cas13、Cas12以及Cas14等蛋白在结合了靶标DNA后会诱发其旁路切割活力,进而被开发成下一代分子诊断技术;在该方向上,我国的科学家也做出了原创性的成果[58-62]。
生命体具有高度复杂性,人工设计的基因线路很难完全按照预期工作,往往需要长时间的反复调谐。克服这一难题的最有效手段,是建立工程化研究平台,大批量测试多种元件、线路、底盘的组合,获取海量实验数据,以指导进一步工程优化与理性设计。工程化平台的核心合成生物研究的自动化设施,亦称为生物铸造厂,依照“设计—构建—测试—学习”的闭环策略组织工艺流程,进行工程化的海量试错,从而快速获得具有目标功能的合成生命体。如美国劳伦斯伯克利国家实验室的Agile BioFoundry,美国伊利诺伊大学的iBioFAB,美国麻省理工学院的MIT-Broad Foundry,英国帝国理工学院的London DNA Foundry,以及产业界的Amyris公司、Zymergen公司、Ginkgo公司等。在此背景下,我国规划由中国科学院深圳先进技术研究院承担建设全球最大的合成生物研究重大科技基础设施。值得指出的是,从原创发现到产业化应用,仅仅依赖于自动化设施是不够的。例如,人工酵母细胞生产青蒿素,是合成生物学领域目前最成功的产业化案例。项目领导者美国Jay Keasling教授,围绕工程化平台建立了完整的研发体系——由劳伦斯伯克利国家实验室负责上游原创发现,联合生物能源研究所(JBEI)负责中游技术开发,Amyris公司负责下游产业应用的创新链条。这些围绕工程化平台的体制与机制创新,值得深入研究与思考,为充分发挥我国重大科技基础设施对合成生物学研究、创新与产业的巨大推动作用提供重要借鉴。
2.6 转化与应用研究利用合成生物学技术,有可能解决长期困扰基因治疗和生物治疗的一系列技术难题,为癌症、糖尿病等复杂疾病开发出更多有效的药物和治疗手段;也有可能突破生物燃料发展的技术瓶颈,模拟乃至设计出更加简单高效的生物过程,生产出更复杂的天然产品,合成出更多的有机化工产品。
在医疗领域,除目前已广为关注的CAR-T等细胞治疗技术[63, 64],设计合成促进细菌入侵肿瘤细胞的线路也是合成生物学理念指导下细胞工程的一个开拓性事例,也是细胞治疗的一个早期例子[65]。此后,工程噬菌体疗法和细胞疗法也不断成熟,如通过工程手段构建益生大肠杆菌,能够识别并消灭绿脓假单胞菌[66],也可以通过表达异源群体感应信号,阻断霍乱弧菌的毒害[67]。
近年来,我国科学家利用合成生物学技术改造的若干高产药物的菌株开始投入工业化应用,发酵水平达到国际或国内领先水平,新增销售额近10亿元人民币。利用基因表达浓度梯度调控技术改造纳他霉素合成酶之间的适配性,纳他霉素发酵水平最高达15.0 g/L[68];解析和重组了玫瑰孢链霉菌达托霉素生物合成过程的级联调控机制和关键基因,达托霉素发酵单位达2.5 g/L以上[69];重构筑波链霉菌中的他克莫司合成模块,实现他克莫司最高发酵单位1 405 μg/mL[70]。
生物燃料领域,研究人员利用合成拨动开关和群体感应系统,协调生物量扩张和乙醇生产[71];设计并构建了生产生物柴油的大肠杆菌,并实现了多功能模块的集成,通过在大肠杆菌中引入外源酶,使其同时具有合成脂肪酯、脂肪醇及蜡,并可利用简单五碳糖为底物的多种功能[72],开辟了微生物工程化炼制能源的新途径。
在化学品生物合成方面,基于化学反应特征和胞内调控位点,计算设计与合成表征了满足数百种重要化学品生物合成的功能元件,为化学分子合成途径和高效人工合成细胞创建奠定了物质基础功能元件。利用DNA组装和精确表达调控技术[73],创建和优化了从葡萄糖到丁二酸、戊二胺、己二酸、5-氨基乙酰丙酸等途径。其中,5-氨基乙酰丙酸产量达到50 g/L,比国际水平高5倍;丁二酸产量达到125 g/L,产物对底盘葡萄糖的转化率为105%(可部分固定CO2)[74],技术指标处于国际领先水平,并已经开始产业转化。在对光合蓝细菌底盘细胞的生理调控机制进行系统研究和形成较为完整的理解和认识的基础上,构建了一系列获得抗逆性能提高的光合蓝细菌底盘细胞[75],通过光合模块、CO2固定和生物合成模块的重构和优化,实现了从CO2生物合成酮、醇、酸等典型化合物,为发展和利用新的碳资源提供了可能,其中丙酮、D-乳酸和3-羟基丁酸均为国际上首次报道[76, 77]。
3 合成生物学的科技支撑与社会治理合成生物学所包含的多学科“融合”已不仅仅是原先意义上的“交叉”,而是科学、技术、工程乃至自然科学与社会科学、管理科学的“会聚”。这种“会聚”不仅使传统的、以学科为特征的研究模式面临巨大挑战,也代表着组织管理与文化建设的重大变革。因此,合成生物学的发展,需要构建与会聚研究能力相适应的生态系统,这样的“会聚”生态系统涉及科研、教育、管理、合作以及资助等各方面[78],需要从政策引导、基础设施建设、机制配套、创新人才培养、工程技术队伍集聚等方面切入,形成产学研创新价值链等多层次、综合性的协作网络。同时,合成生物学作为新兴颠覆性领域,还需要对制度、法规和公众舆论引导等方面进行长期研究,并逐渐形成系统性的风险管控体系,以保证并促进其健康快速的发展。
3.1 研究与开发体系 3.1.1 促进创新发展的政策引导和能力建设合成生物学的发展不仅需要国家和政府部门经费的支持和政策引导,更需要充分调动产学研金各方的积极性,形成有科技创造活力、产业转化能力和社会经济效益的创新价值链。要根据价值链不同阶段的不同目标,采取不同的支持方式,积极寻求多元化的资金支持,实现从基础研究到技术创新,从工程平台建设到产品开发、产业转化的多层次、分阶段的快速与稳定的发展。在投入方面,政府要特别强调支持用于转化研究的、具有合成生物学特色的元件库、数据库,以及工程技术平台等的建设。通过国家级的基础设施或国家实验室的建设,不仅能解决学科会聚融合的问题,还能更有效地发挥研发机构、资助机构和监管机构的作用。另外,要通过政策激励鼓励企业,一方面增加研发投入,以支持人才队伍建设来支撑企业研发能力的提升;另一方面,以支持合成生物学先驱企业在技术和产品的(包括技术服务产品)研发推广过程中,加强标准建立和推广,引导产业变革。
3.1.2 有利于“会聚”研究的机制体制和文化氛围由于会聚研究在很大程度上依赖于多个领域、多个合作伙伴的专业知识的整合,因而,开放与包容的文化、组织结构与管理、通用的概念与标准,以及共同的目标,对于支持这种密切合作关系十分必要。首先,制定支持交叉会聚的相关政策,建立统一协调的机制,设置相应的研究单元(机构)、为其量身定制的管理架构,推动传统组织架构间的跨学科研究。另外,要建立有效的组织文化,创造思想交流的机会、提高对学科差异的理解;制定公平、灵活的预算与费用分摊政策,配套相应的资源投入与经费使用机制,改革人员招聘、晋升、考评制度,以更好地支持跨学科的“会聚”研究。
3.2 教育与人才培养 3.2.1 夯实多学科专业基础的学科教育合成生物学的会聚发展模式,需要创新的教育模式和人才培养模式。合成生物学的教育,不仅应教授具体的理论与实验技术,还应传递合成生物学的价值观和理念。这些价值观包括通过超越传统生物学科的新型合作以及支持开源资源的开发和利用,致力于跨学科的创新研究。应精心设计和推行“会聚”的教育计划和培训项目。需要注意的是,在目前阶段上,并非一定要设立“合成生物学”专业,而是要多方协同教育资源,夯实多学科专业基础,强调各“融合”学科相关部分之间的“会聚”;通过教学相长,建立适应合成生物学发展的师资队伍,进一步完善本科生、研究生的创新教育体系。同时,注重学科建设与人才培养相结合,强调基地建设与队伍建设相结合。
3.2.2 培养具备跨学科工作的能力和素质在合成生物学的人才培养中,应强调和倡导勇于创新、开放、合作的意识和理念[79],才能培养新一代的合成生物学家。起源于2003年美国麻省理工学院合成生物学课程的国际基因工程机器大赛(iGEM),每年吸引着全球众多高校乃至中学生团队参与。iGEM通过学生自主选题、导师提供实验室和指导意见的模式,使学生不但可以将学到的知识运用到实际科研工作中,而且对其科学思维、自主学习、人际交往、团队协作、跨学科交流等能力进行了全面锻炼[80]。通过这种“会聚”研究能力的培养,保持独特的创新文化和合作分享文化,培育造就跨学科的研究梯队和系列人才,集中解决合成生物学的关键科学问题和产业转化问题。
3.3 社会影响研究与治理合成生物学技术的快速发展,挑战了传统伦理观念,直接带来涉及开源共享与知识产权/生物安全安保等问题。目前尚缺少伦理、法律、社会学等方面的研究和讨论,以及支撑标准建立的相关技术及方法的研究。
3.3.1 推动产品研发与产业应用的标准和规范推进合成生物学产品研发与产业应用,需要建立相关元器件和技术服务的标准。在合成生物学技术源头研发阶段,应重点加强知识产权(包括标准化)的保护与管理,促进资源开放共享;在市场应用发展到一定程度时,要及时研究制定相关的技术/科学标准、环境/安全标准、过程可重复的计量标准等,并加强与国际标准机构的交流合作。
合成生物学产品的市场准入,是保证合成生物学技术推动产业转型,服务社会进步的重要节点。目前,对于纯化产品(如药物)和非食用类产品,原则上可沿用现有的监管原则。对于非纯化的食用类产品,应尽早利用这一机遇,建立科学、理性、安全、高效的评估规范,明确新产品的申报与审批路径,统一市场准入标准和审查制度,推动新产品早日进入市场。
3.3.2 促进健康发展的监督和管理如何保障合成生物学技术和相关产品的生物安全,防止技术的滥用,以及防止伦理方面的冲突已成为极其重要且亟待解决的问题。应密切跟踪合成生物学发展所带来的新的生物安全、伦理等问题,大力开展相关的风险研究,研究新特征、新变化;梳理现有管理政策中存在的问题、漏洞和空白,适时进行补充、修改;还应提高研究人员的安全意识并重视对公众的宣传和公众的参与,完善基于信用档案的资质审核制度,促进自我监管。
| [1] |
Leduc S. The Mechanism of Life. Whitefish: Kessinger Legacy Reprint, 1911.
|
| [2] |
Way J C, Collins J J, Keasling J D, et al. Integrating biological redesign:Where synthetic biology came from and where it needs to go[J]. Cell, 2014, 157(1): 151-161. DOI:10.1016/j.cell.2014.02.039 |
| [3] |
Benner S A, Sismour A M. Synthetic biology[J]. Nature Reviews Genetics, 2005, 6(7): 533-543. DOI:10.1038/nrg1637 |
| [4] |
Cameron D E, Bashor C J, Collins J J. A brief history of synthetic biology[J]. Nature Reviews Microbiology, 2014, 12: 381-389. DOI:10.1038/nrmicro3239 |
| [5] |
National Research Council. Convergence:Facilitating Transdisciplinary Integration of Life Science, Physical Science, Engineering, and Beyond[M]. Washington DC: The National Academies Press, 2014.
|
| [6] |
Purnick P E, Weiss R. The second wave of synthetic biology:from modules to systems[J]. Nature Reviews Molecular Cell Biology, 2009, 10(6): 410-422. DOI:10.1038/nrm2698 |
| [7] |
Hezari M, Ketchum R E, Gibson D M, et al. Taxol production and taxadiene synthase activity in Taxus canadensis cell suspension cultures[J]. Archives of Biochemistry and Biophysics, 1997, 337: 185-190. DOI:10.1006/abbi.1996.9772 |
| [8] |
Ajikumar P K, Xiao W H, Tyo K E, et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli[J]. Science, 2010, 330: 70-74. DOI:10.1126/science.1191652 |
| [9] |
Alper H, Fischer C, Nevoigt E, et al. Tuning genetic control through promoter engineering[J]. Proceedings of the National Academy of Sciences of USA, 2005, 102(36): 12678-12683. DOI:10.1073/pnas.0504604102 |
| [10] |
Nevoigt E, Kohnke J, Fischer C R, et al. Engineering of promoter replacement cassettes for fine-tuning of gene expression in Saccharomyces cerevisiae[J]. Applied and Environmental Microbiology, 2006, 72(8): 5266-5273. DOI:10.1128/AEM.00530-06 |
| [11] |
Orelle C, Carlson E D, Szal T, et al. Protein synthesis by ribosomes with tethered subunits[J]. Nature, 2015, 524: 119-124. DOI:10.1038/nature14862 |
| [12] |
Taylor A I, Pinheiro V B, Smola M J, et al. Catalysts from synthetic genetic polymers[J]. Nature, 2015, 518(7539): 427-430. DOI:10.1038/nature13982 |
| [13] |
Kiss G, Çelebi N, Moretti R, et al. Computational enzyme design[J]. Angewandte Chemie International Edition, 2013, 52: 5700-5725. DOI:10.1002/anie.201204077 |
| [14] |
Privett H K, Kiss G, Lee T M, et al. Iterative approach to computational enzyme design[J]. Proceedings of the National Academy of Sciences of USA, 2012, 109: 3790-3795. DOI:10.1073/pnas.1118082108 |
| [15] |
Liu XH, Kang FY, Hu C, et al. A genetically encoded photosensitizer protein facilitates the rational design of a miniature photocatalytic CO2-reducing enzyme[J]. Nature Chemistry, 2018. DOI:10.1038/s41557-018-0150-4 |
| [16] |
Gardner T S, Cantor C R, Collins J J. Construction of a genetic toggle switch in Escherichia coli[J]. Nature, 2000, 403: 339-342. DOI:10.1038/35002131 |
| [17] |
Elowitz M B, Leibler S. A synthetic oscillatory network of transcriptional regulators[J]. Nature, 2000, 403: 335-338. DOI:10.1038/35002125 |
| [18] |
Weiss R, Basu S. The device physics of cellular logic gates. First Workshop on Non-Silicon Computing.[2002-01-01]. http://www.hpcaconf.org/hpca8/nsc.pdf.
|
| [19] |
Endy D. Foundations for engineering biology[J]. Nature, 2005, 438: 449-453. DOI:10.1038/nature04342 |
| [20] |
Isaacs F J, Dwyer D J, Ding C, et al. Engineered riboregulators enable post-transcriptional control of gene expression[J]. Nature Biotechnology, 2004, 22: 841-847. DOI:10.1038/nbt986 |
| [21] |
Levskaya A, Chevalier A A, Tabor J J, et al. Synthetic biology:engineering Escherichia coli to see light[J]. Nature, 2005, 438: 441-442. DOI:10.1038/nature04405 |
| [22] |
Stricker J, Cookson S, Bennett M R, et al. A fast, robust and tunable synthetic gene oscillator[J]. Nature, 2008, 456: 516-U39. DOI:10.1038/nature07389 |
| [23] |
Tigges M, Marquez-Lago T T, Stelling J, et al. A tunable synthetic mammalian oscillator[J]. Nature, 2009, 457: 309-312. DOI:10.1038/nature07616 |
| [24] |
Friedland A E, Lu T K, Wang X, et al. Synthetic gene networks that count[J]. Science, 2009, 324: 1199-1202. DOI:10.1126/science.1172005 |
| [25] |
Budin I, Devaraj N K. Membrane assembly driven by a biomimetic coupling reaction[J]. Journal of the American Chemical Society, 2012, 134: 751-753. DOI:10.1021/ja2076873 |
| [26] |
Smanski M J, Bhatia S, Zhao D, et al. Functional optimization of gene clusters by combinatorial design and assembly[J]. Nature Biotechnology, 2014, 32: 1241-1249. DOI:10.1038/nbt.3063 |
| [27] |
Chen Y, Kim J K, Hirning A J, et al. Emergent genetic oscillations in a synthetic microbial consortium[J]. Science, 2015, 349: 986-989. DOI:10.1126/science.aaa3794 |
| [28] |
Gao X J, Chong L S, Kim M S, et al. Programmable protein circuits in living cells[J]. Science, 2018, 361(6408): 1252-1258. DOI:10.1126/science.aat5062 |
| [29] |
Andrews L B, Nielsen A A K, Voigt C A. Cellular checkpoint control using programmable sequential logic[J]. Science, 2018, 361(6408): pii:eaap8987. DOI:10.1126/science.aap8987 |
| [30] |
Martin V J, Pitera D J, Withers S T, et al. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids[J]. Nature Biotechnology, 2003, 21: 796-802. DOI:10.1038/nbt833 |
| [31] |
Win M N, Smolke C D. Higher-order cellular information processing with synthetic RNA devices[J]. Science, 2008, 322: 456-460. DOI:10.1126/science.1160311 |
| [32] |
Paddon C J, Westfall P J, Pitera D J, et al. High-level semisynthetic production of the potent antimalarial artemisinin[J]. Nature, 2013, 496: 528-532. DOI:10.1038/nature12051 |
| [33] |
Galanie S, Thodey K, Trenchard I J, et al. Complete biosynthesis of opioids in yeast[J]. Science, 2015, 349: 1095-1100. DOI:10.1126/science.aac9373 |
| [34] |
Atsumi S, Hanai T, Liao J C. Non-fermentative pathways for synthesis of branched-chain higher alcohols as biofuels[J]. Nature, 2008, 451: 86-89. DOI:10.1038/nature06450 |
| [35] |
Huo Y X, Cho K M, Rivera J G L, et al. Conversion of proteins into biofuels by engineering nitrogen flux[J]. Nature Biotechnology, 2011, 29: 346-351. DOI:10.1038/nbt.1789 |
| [36] |
Steen E J, Kang Y, Bokinsky G, et al. Microbial production of fatty-acid-derived fuels and chemicals from plant biomass[J]. Nature, 2010, 463: 559-562. DOI:10.1038/nature08721 |
| [37] |
Choi Y J, Lee S Y. Microbial production of short-chain alkanes[J]. Nature, 2013, 502: 571-574. |
| [38] |
Yim H, Haselbeck R, Niu W, et al. Metabolic engineering of Escherichia coli for direct production of 1, 4-butanediol[J]. Nature Chemical Biology, 2011, 7: 445-452. DOI:10.1038/nchembio.580 |
| [39] |
Holtz W J, Keasling J D. Engineering static and dynamic control of synthetic pathways[J]. Cell, 2010, 140: 19-23. DOI:10.1016/j.cell.2009.12.029 |
| [40] |
Callura J M, Cantor C R, Collins J J. Genetic switchboard for synthetic biology applications[J]. Proceedings of the National Academy of Sciences of USA, 2012, 109: 5850-5855. DOI:10.1073/pnas.1203808109 |
| [41] |
Yan X, Fan Y, Wei W, et al. Production of bioactive ginsenoside compound K in metabolically engineered yeast[J]. Cell Research, 2014, 24: 770-773. DOI:10.1038/cr.2014.28 |
| [42] |
Wang P, Wei Y, Fan Y, et al. Production of bioactive ginsenosides Rh2 and Rg3 by metabolically engineered yeasts[J]. Metabolic Engineering, 2015, 29: 97-105. DOI:10.1016/j.ymben.2015.03.003 |
| [43] |
Wei W, Wang P, Wei Y, et al. Characterization of panax ginseng UDP-Glycosyltransferases catalyzing protopanaxatriol and biosyntheses of bioactive ginsenosides F1 and Rh1 in metabolically engineered yeasts[J]. Molecular Plant, 2015, 8: 1412-1424. DOI:10.1016/j.molp.2015.05.010 |
| [44] |
Yao Y F, Wang C S, Qiao J, et al. Metabolic engineering of Escherichia coli for production of salvianic acid A via an artificial biosynthetic pathway[J]. Metabolic Engineering, 2013, 19(5): 79-87. |
| [45] |
Cello J, Paul A V, Wimmer E. Chemical synthesis of poliovirus cDNA:Generation of infectious virus in the absence of natural template[J]. Science, 2002, 297: 1016-1018. DOI:10.1126/science.1072266 |
| [46] |
mith HO1, Hutchison CA 3rd, Pfannkoch C, et al. Generating a synthetic genome by whole genome assembly:phiX174 bacteriophage from synthetic oligonucleotides[J]. Proceedings of the National Academy of Sciences of USA, 2003, 100(26): 15440-15445. DOI:10.1073/pnas.2237126100 |
| [47] |
Gibson D G, Glass J I, Lartigue C, et al. Creation of a bacterial cellcontrolled by a chemically synthesized genome[J]. Science, 2010, 329: 52-56. DOI:10.1126/science.1190719 |
| [48] |
Gibson D G, Smith H O, Hutchison C A, et al. Chemical synthesis of the mouse mitochondrial genome[J]. Nature Methods, 2010, 7(11): 901-903. DOI:10.1038/nmeth.1515 |
| [49] |
Dymond J S, Richardson S M, Coombes C E, et al. Synthetic chromosome arms function in yeast and generate phenotypic diversity by design[J]. Nature, 2011, 477: 471-476. DOI:10.1038/nature10403 |
| [50] |
Annaluru N, Muller H, Mitchell L A, et al. Total synthesis of a functional designer eukaryotic chromosome[J]. Science, 2014, 343: 1426-1429. DOI:10.1126/science.343.6178.1426 |
| [51] |
Mercy G, Mozziconacci J, Scolari V F, et al. 3D organization of synthetic and scrambled chromosomes[J]. Science, 2017, 355(6329): eaaf4597. DOI:10.1126/science.aaf4597 |
| [52] |
Shao Y Y, Lu N, Wu Z F, et al. Creating a functional singlechromosome yeast[J]. Nature, 2018, 560: 331-335. DOI:10.1038/s41586-018-0382-x |
| [53] |
Palluk S, Arlow D H, de Rond T, et al. De novo DNA synthesis using polymerase-nucleotide conjugates[J]. Nature Biotechnology, 2018, 36: 645-650. DOI:10.1038/nbt.4173 |
| [54] |
Cong L, Ran F A, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [55] |
Ran F A, Hsu P D, Lin C Y, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J]. Cell, 2013, 154(6): 1380-1389. DOI:10.1016/j.cell.2013.08.021 |
| [56] |
Komor A C, Kim Y B, Packer M S, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature, 2016, 533: 420-424. DOI:10.1038/nature17946 |
| [57] |
Wang H, Russa M L, Qi L S. CRISPR/Cas9 in genome editing and beyond[J]. Annual Review of Biochemistry, 2016, 85: 227-264. DOI:10.1146/annurev-biochem-060815-014607 |
| [58] |
Li S Y, Cheng Q X, Wang J M, et al. CRISPR-Cas12a-assisted nucleic acid detection[J]. Cell Discovery, 2018, 4: 20. |
| [59] |
Li S Y, Cheng Q X, Liu J K, et al. , CRISPR-Cas12a has both cis-and trans-cleavage activities on single-stranded DNA[J]. Cell Research, 2018, 28(4): 491-493. DOI:10.1038/s41422-018-0022-x |
| [60] |
Harrington L B, Burstein D, Chen J S, et al. Programmed DNA destruction by miniature CRISPR-Cas14 enzymes[J]. Science, 2018, eaav4294. DOI:10.1126/science.aav4294 |
| [61] |
Chen J S, Ma E, Harrington L B, et al. , CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity[J]. Science, 2018, 360(6387): 436-439. DOI:10.1126/science.aar6245 |
| [62] |
Gootenberg J S, Abudayyeh O O, Lee J W, et al. Nucleic acid detection with CRISPR-Cas13a/C2c2[J]. Science, 2017, 356(6336): 438-442. DOI:10.1126/science.aam9321 |
| [63] |
June C H, O'Connor R S, Kawalekar O U, et al. CAR T cell immunotherapy for human cancer[J]. Science, 2018, 359(6382): 1361-1365. DOI:10.1126/science.aar6711 |
| [64] |
Singh N, Shi J, June C H, et al. Genome-editing technologies in adoptive T cell immunotherapy for cancer[J]. Current Hematologic Malignancy Reports, 2017, 12(6): 522-529. DOI:10.1007/s11899-017-0417-7 |
| [65] |
Anderson J C, Clarke E J, Arkin A P, et al. Environmentally controlled invasion of cancer cells by engineered bacteria[J]. Journal of Molecular Biology, 2006, 355: 619-627. DOI:10.1016/j.jmb.2005.10.076 |
| [66] |
Gupta S, Bram E E, Weiss R. Genetically programmable pathogen sense and destroy[J]. ACS Synthetic Biology, 2013, 2: 715-723. DOI:10.1021/sb4000417 |
| [67] |
Duan F, March J C. Engineered bacterial communication prevents Vibrio cholerae virulence in an infant mouse model[J]. Proceedings of the National Academy of Sciences of USA, 2010, 107: 11260-11264. DOI:10.1073/pnas.1001294107 |
| [68] |
Jiang H, Wang Y Y, Fan W M, et al. Improvement of natamycin production by engineering of phosphopanteth transferases in streptomyces chattanoogensis L10[J]. Applied and Environmental Microbiology, 2013, 79(11): 3346-3354. DOI:10.1128/AEM.00099-13 |
| [69] |
Mao X M, Luo S, Zhou R C, et al. Transcriptional regulation of the daptomycin gene cluster in streptomyces roseosporusby an autoregulator, AtrA*[J]. Journal of Biological Chemistry, 2015, 290: 7992-8001. DOI:10.1074/jbc.M114.608273 |
| [70] |
Zhang X S, Lou H D, Tao Y, et al. FkbN and Tcs7 are pathwayspecific regulators of the FK506 biosynthetic gene cluster in Streptomyces tsukubaensis L19[J]. Journal of Industrial Microbiology & Biotechnology, 2016, 43: 1693-1703. |
| [71] |
Anesiadis N, Cluett W R, Mahadevan R. Dynamic metabolic engineering for increasing bioprocess productivity[J]. Metabolic Engineering, 2008, 10: 255-266. DOI:10.1016/j.ymben.2008.06.004 |
| [72] |
Steen E J, Kang Y, Bokinsky G, et al. Microbial production of fatty-acid-derived fuels and chemicals from plant biomass[J]. Nature, 2010, 463: 559. DOI:10.1038/nature08721 |
| [73] |
Yu X Y, Liu T G, Zhu F Y, et al. In vitro reconstitution and steadystate analysis of the fatty acid synthase from Escherichia coli[J]. Proceedings of the National Academy of Sciences of USA, 2011, 108(46): 18643-18648. DOI:10.1073/pnas.1110852108 |
| [74] |
Zhu X N, Tan Z G, Xu H T, et al. Metabolic evolution of two reducing equivalent-conserving pathways for high-yield succinate production in Escherichia coli[J]. Metabolic Engineering, 2014, 24: 87-96. DOI:10.1016/j.ymben.2014.05.003 |
| [75] |
Jin H, Chen L, Zhang W. Engineering biofuel tolerance in nonnative producing microorganisms[J]. Biotechnology Advances, 2014, 32(2): 541-548. DOI:10.1016/j.biotechadv.2014.02.001 |
| [76] |
Zhou J, Zhang H F, Zhang Y P, et al. Designing and creating a modularized synthetic pathway in cyanobacterium Synechocystis enables production of acetone from carbon dioxide[J]. Metabolic Engineering, 2012, 14(4): 394-400. DOI:10.1016/j.ymben.2012.03.005 |
| [77] |
Wang B, Pugh S, Nielsen D R, et al. Engineering cyanobacteria for photosynthetic production of 3-hydroxybutyrate directly from CO2[J]. Metabolic Engineering, 2013, 16: 68-77. DOI:10.1016/j.ymben.2013.01.001 |
| [78] |
National Research Council. Convergence:Facilitating Transdisciplinary Integration of Life Sciences, Physical Sciences, Engineering, and Beyond[M]. Washinton DC: National Academies Press, 2014.
|
| [79] |
Farny N G. A vision for teaching the values of synthetic biology[J]. Trends Biotechnol, 2018, 36(11): 1097-1100. DOI:10.1016/j.tibtech.2018.07.019 |
| [80] |
谭静, 胡启文, 肖文刚, 等. 国际基因工程机器大赛对本科生创新能力培养的启示[J]. 卫生职业教育, 2018(1): 1-3. |



