2. 江南大学 食品科学与技术国家重点实验室 无锡 214122;
3. 江南大学 工业生物技术教育部重点实验室 无锡 214122;
4. 江南大学 粮食发酵工艺与技术国家工程实验室 无锡 214122
2. State Key Laboratory of Food Science and Technology, Jiangnan University, Wuxi 214122, China;
3. Key Laboratory of Industrial Biotechnology(Ministry of Education), Jiangnan University, Wuxi 214122, China;
4. National Engineering Laboratory for Cereal Fermentation Technology, Jiangnan University, Wuxi 214122, China
LIU Liming Professor at Jiangnan University.He received his Ph.D.in fermentation engineering from Jiangnan University in 2006.From 2009 to 2010, he worked at Chalmers University of Technology as a postdoctoral fellow, focusing on the research of systems biology.His group focuses on the research of systems biology, metabolic engineering, and enzyme engineering.He was supported by the Scientific and Technological Innovation Leading Talents of National "Ten Thousand Talents Program" of Ministry of Science and Technology of China and the National Natural Science Foundation of China.E-mail:mingll@jiangnan.edu.cn
SUN Jibin Professor at Tianjin Institute of Industrial Biotechnology, CAS since 2008, winner of the Hundred Talents Program of CAS.He got his Ph.D.from German Research Centre for Biotechnology/Technical University of Braunschweig in 2004.His researches mainly focus on systems and synthetic biotechnology of industrial microorganisms in terms of tools development and industrial application.E-mail:sunjibin@tib.cas.cn
SUN Jibin
基因工程技术的出现使得人类不再受限于自然变异和筛选,通过有目的的基因组改造可以大幅度地提升菌株的生产性能。例如,通过对代谢网络中酶基因的引入、敲除或精细调控可以创造新的细胞,将廉价的原料转化为有价值的目标产品。这正是目前代谢工程和合成生物学研究的主要内容。从原料到产品的生物转化途径常常包括几十步的酶反应,目前常用的代谢工程改造策略主要就是过表达和优化产物合成途径中的关键酶、敲除副产物生成途径、解除产物合成抑制等。通过这种传统的代谢工程改造策略,人们已经获得大量工程菌株应用于生物化学品(如氨基酸、有机酸、维生素、抗生素)、生物燃料(如乙醇)、生物材料(如聚羟基脂肪酸酯、聚乳酸等)的生产,以可再生的原料和绿色的生产方式,推动人类工业经济发展进入一个新的阶段。
代谢工程研究遇到的主要瓶颈就是细胞代谢网络的复杂性。自然存在的细胞是长期进化的结果,常包括上千个酶基因并具有复杂的调控机制,仅仅对原料到产品合成途径上的少数基因进行改造很多时候并不能达到预期的结果。随着基因组测序技术的快速发展,完成全基因组测序的生物越来越多;通过基因组注释信息可以快速确定其基因组能够编码哪些酶、进行哪些反应,由此构建出该生物的代谢网络模型[1]。这些基因组规模代谢网络模型和长期积累形成的跨物种代谢反应数据库,如KEGG和MetaCyc等为代谢网络计算模拟和设计奠定了基础,为合成生物学和代谢工程提供了新的策略,从而大幅度加快了菌种定向开发和工业化应用进程。
1 合成新产品:从无到有通过引入外源基因使某一生物合成其原来不能合成的新产品,这是一种常用的代谢工程手段。例如,Galanie等[2]通过将来源于6种不同生物(包括植物、哺乳动物和细菌)的10余个基因引入酵母中实现了鸦片类药物的人工合成。通过微生物细胞工厂合成植物来源的天然产物,其好处是微生物生长更快,单位时空内产量更高,可以通过较小的发酵工厂代替大面积的农业种植,大大降低生产成本。
从无到有的设计首先需要确定在底盘细胞中引入哪些外源基因才能打通该新产品的合成途径。因此,需要将底盘细胞代谢网络与代谢反应数据库(如KEGG、MetaCyc等)相结合,通过一定的计算方法找出需要在底盘细胞中引入的新反应,形成新的途径,以实现新产品的合成。例如,Zhang等[3]将KEGG反应引入到大肠杆菌代谢网络中,构建了包含7 316个反应的复合代谢网络模型;基于该模型,以葡萄糖为底物进行通量平衡分析发现,1 777个异源产品可以通过在大肠杆菌中引入适当外源反应合成。这一研究为选择合适外源基因构建大肠杆菌的人工细胞工厂提供了指导。该方法还可以计算其他微生物为底盘时的结果,并通过比较分析确定针对某一特定产品的最优底盘生物(需引入外源基因少,相应前体合成能力强等)。Chatsurachai等[4]分别以大肠杆菌、谷氨酸棒杆菌和酿酒酵母为底盘细胞,通过逐级扩展算法将来源于KEGG、BRENDA和ENZYME 3个数据库中的异源代谢物和反应添加到3个菌株代谢模型中以合成新的产物。该扩展算法首先从数据库中找出那些反应物在(但产物不在)该菌株代谢网络中的反应,通过将这些反应添加到模型中,使该模型可以生成那些新反应中的产物。随后在扩展的模型基础上重复该新反应添加过程,直到没有新反应可以添加。最终,针对每一个底盘细胞得到其可以合成的新产品列表及生成某一新产品需要引入的新反应。作者通过该方法预测出大肠杆菌和酿酒酵母是1, 3-丙二醇合成的可行宿主,并且只需要在这两种株菌中引入甘油脱氢酶和1, 3-丙二醇氧化还原酶便可实现产品合成。
上述方法均需基于特定代谢反应数据库扩展特定底盘细胞的代谢网络,要合成的代谢物和相关反应必须存在于数据库中才能设计出相关途径,而自然界中还存在许多未知的生化反应并未包含在这些数据库中。例如,很多酶,如醛缩酶等均具有广谱催化活性,除了已知反应外还可能催化其他代谢物的转化。通过酶改造,构建具有新催化活性的酶可以实现新的转化过程获得新的生物产品,甚至是合成出自然界不存在的全新生物分子。针对这一问题,人们提出了基于生化反应规则和化合物结构特征来设计新反应的方法。例如,Hatzimanikatis的研究组基于化学信息学,在KEGG数据库中反应的基础上扩展酶反应,创建了ATLAS数据库[5],其中包含13万多个在已知反应基础上通过结构相似性设计得到的新生化反应。利用该数据库,他们设计了多条可以生成生物燃料丁酮的全新途径,并对不同途径的理论得率进行了比较分析,以获得最优途径[6]。
2 提高产品得率:从有到优在赋予了细胞新的合成能力,实现了从无到有的改造之后,代谢工程的下一个目标就是以最小的原料消耗生产最多的产品。消耗单位原料(底物)可以生成的产物的量常用“得率”来表征,其是反映原料成本的一个重要指标,尤其对大宗化学品来说原料成本是影响产品生产成本的最主要因素。对某些通过简单途径就可以合成得到的产品,如乙醇、乳酸等,通过直观观察就可以得到得率最大化的代谢途径;但对大部分代谢产物,如核苷酸、芳香族氨基酸等,由于其合成途径复杂,涉及副产物及多个前体,很难通过简单观察或手工计算得到其理论得率。此外,在产品合成过程中还涉及能量(ATP)和还原力的产生和消耗,需要额外消耗底物产生能量和还原力,或通过某种途径消耗掉多余的能量还原力,这使得产物得率的计算更为复杂。基于代谢网络模型,人们提出了通量平衡分析(Flux Balance Analysis,FBA)的计算分析方法,以准确计算该网络中产物的理论得率及能达到该理论得率的最优转化途径[7]。FBA基于代谢网络模型中的生化反应计量关系(可以理解为一个反应中各种反应物和产物的摩尔比)和反应的不可逆性约束,通过求解约束最优化问题,得到使产物生成最大化并且满足物料和能量平衡的产品合成最优途径。由于求得的途径是通过对整体网络计算分析得到的,比由生化知识得出的常规合成途径更为全面,该方法能考虑能量平衡、辅因子平衡、多前体的平衡以及合成途径中副产物的再回收利用[8]。该方法可以获得实现同样最大得率的多种不同策略,可能超越我们对生物化学知识的理解,得到与已知传统代谢途径不同的新途径,从而提供全新的改造靶点和策略。例如,很多生物产品都是以乙酰辅酶A为前体合成得到,但以葡萄糖为底物时,乙酰辅酶A一般由糖酵解终产物丙酮酸脱羧生成,这导致葡萄糖中的2个碳原子都以CO2形式损失掉而不能进入产物,1个葡萄糖(含6个碳原子)最多得到2个乙酰辅酶A(共4个碳原子)。因此,即使全部乙酰辅酶A中的碳都转化到产品中,产物的最大碳摩尔得率也只能达到0.67(图 1a)。而以大肠杆菌代谢网络模型iJO1366通过FBA对乙酰辅酶A衍生产品聚3-羟基丁酸酯(P3HB)最优得率计算结果却可达到0.86,这表明在其代谢网络存在一些新途径可以减少碳损失。求得的最优途径中的通量分布如图 1b所示,可见在该途径中除了常规葡萄糖经过EMP途径生成丙酮酸进而生成乙酰辅酶A途径外,还有一条称之为苏氨酸循环的途径回收甲酸和CO2,由磷酸烯醇式丙酮酸再回到丙酮酸同时生成一个乙酰辅酶A用于产品合成,从而提高了产品得率。基于该途径计算结果,Lin等[9]在大肠杆菌中通过解调控等激活了该循环途径,将P3HB得率提高了将近一倍。
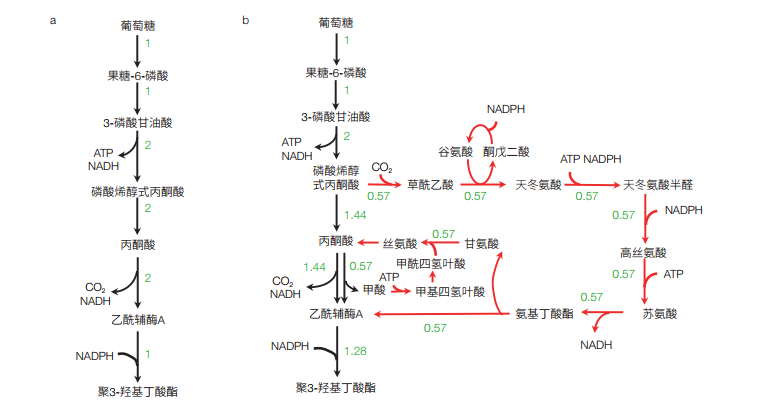
|
| 图 1 由代谢网络分析确定聚3-羟基丁酸酯(P3HB)合成最优途径 (a)常规途径由于丙酮酸脱氢步骤的碳损失1个葡萄糖(6个碳原子)只能生成1个P3HB(4个碳原子),碳摩尔得率为0.67;(b)通过代谢网络模型计算得到新途径通过苏氨酸循环由1个葡萄糖得到1.28个P3HB,碳摩尔得率提高到0.85。新途径的实现可显著降低原料成本 |
外源基因的引入不但可以使细胞从无到有获得合成新产品的能力,也可以形成新的途径提高已有产品的得率。Bogorad等[10]通过在大肠杆菌中引入来源于B. adolescentis的戊糖/己糖磷酸转酮酶(Fxpk)构建了非氧化酵解途径(non-oxidative glycolysis,NOG)。通过该途径可以实现1个葡萄糖生成3个乙酸,即完全没有碳损失(图 2)。我们在大肠杆菌iJO1366模型中引入该反应后通过FBA分析对上百种生物产品的理论得率进行了计算,发现至少有几十种生化产品的得率都有了显著提高。在这一计算结果指导下,构建了含NOG途径的大肠杆菌菌株并分别用于P3HB、丙酮和异丙醇的生产。改造后的菌株与出发菌株相比得率均有显著提高[11, 12]。最近Meadows等[13]将NOG途径引入到酿酒酵母细胞中构建了法尼烯合成细胞工厂,最终菌株法尼烯对葡萄糖的碳摩尔得率由0.52提高到0.65。
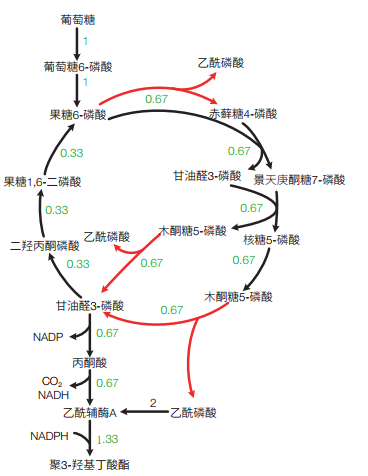
|
| 图 2 引入NOG途径后乙酰辅酶A衍生产品合成最优途径 |
基于FBA模型获得的反应列表及其通量代表了理想状态下反应速度最佳比例,可以作为代谢工程改造的终极目标——酶的实际表达量与活性要刚好满足实现最佳代谢通量的要求,不多也不少。实际代谢工程实践中,容易做到酶活性的“有无”调控或者叫“静态控制”,比如通过基因敲除彻底失活某个生化反应,或者通过外源基因的引入增加某个反应,但是难以做到按照FBA模型预测的通量要求精确地调控每个酶的活性大小。实际上,细胞在长期进化中已经形成多种多样的动态调控机制,根据不同的环境,精准地调控酶的表达(诱导与阻遏),精准地控制酶的活性(激活与抑制),确保代谢网络能够适应环境,实现全局最优化。例如,在发酵培养过程中细胞适应环境变化不断调整自身的代谢状态,形成延迟期、指数生长期和产物生成期等不同阶段,每个阶段对基因蛋白表达水平、代谢物浓度和通量分布都有不同要求。学习自然进化形成的动态调控机制,近年来人们提出了动态代谢工程(Dynamic Metabolic Engineering)的概念和方法,即根据胞内状态和环境条件的变化对基因表达和活性进行动态调控,避免中间代谢产物的积累或不足,满足不同阶段的生长和产物合成对代谢通量分布的差异化需求。基因回路或称转录调节回路,是最常用的动态调控手段,借助于小分子与转录因子(一种蛋白质)的相互作用和转录因子与特殊DNA区域(如启动子、增强子、沉默子)的相互作用,将胞内外环境信号转化为对酶蛋白表达的精准控制,确保细胞随时调节代谢活动以适应环境变化[14-16]。通过动态控制酶的水平,可以实现代谢途径流向的动态转向调控(图 3a),或实现代谢活性的精准平衡(图 3b)。
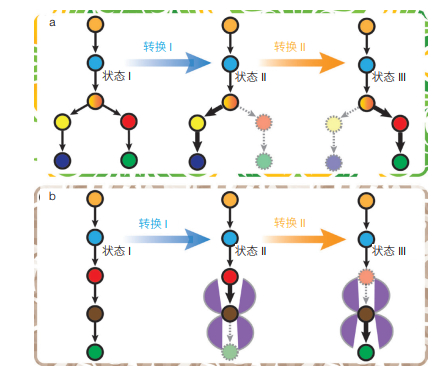
|
| 图 3 代谢途径中代谢流的动态调控 (a)动态调控代谢途径流向;(b)动态调控代谢途径平衡 |
基因回路可以在特定的时间和细胞密度下关闭或开启基因表达,实现代谢流向在细胞生长的内源代谢途径和化学品生产的异源途径之间的切换。切换控制主要集中在生长必需基因上,因为通过它们可以实现生物量形成和产品生物合成之间最直接的控制。基因回路除了可以用于控制酶的合成速度,还可以精细调节酶的降解速度[17]。例如,葡萄糖-6-磷酸(G6P)是一个代谢流控制的重要节点,既可以通过磷酸果糖激酶-1(Pfk-1)进入糖酵解途径从而进入内源代谢,也可以通过合成途径进入异源代谢。通过基因回路控制Pfk-1的活性就可能将碳代谢流的流向从“生长模式”切换到“生产模式”。为实现这一目的,Brockman和Prather[18]设计了一种蛋白质降解回路,将Pfk-1与SsrA降解标签融合,SsrA标签能够控制适配蛋白SspB的结合、ClpXP蛋白酶招募和蛋白水解降解,从而实现Pfk-1降解速度的动态控制。将该回路用于大肠杆菌生产肌醇,与静态控制相比,肌醇的积累量提高了2倍。基因回路通过控制必需基因的表达水平实现代谢途径的动态调控,成为工业菌株代谢工程设计改造的有效手段:通过对内源代谢网络进行重新布线,替代传统静态控制,实现基因表达的时间依赖性控制以及多个基因或途径的平衡互作等,拥有广泛的应用前景[17]。
代谢中间产物的过量积累可能引发副反应,占据重要细胞资源(如磷酸盐、辅因子),以及产生直接或间接的细胞毒性,对细胞生长或产物合成不利。基因回路可以借助对中间产物浓度响应的生物传感器,激活代谢途径下游酶,阻遏代谢途径上游酶,实现中间代谢物合成和利用速度适配,提高整个代谢途径的合成效率。例如,莽草酸是芳香族氨基酸代谢途径的中间体,莽草酸的积累会与芳香族氨基酸的代谢产生竞争。可以通过响应莽草酸的基因回路控制莽草酸代谢途径关键酶——莽草酸激酶(AroK)实现代谢途径各分支的平衡,实现碳代谢流在莽草酸积累与芳香族氨基酸代谢之间的平衡。Mahr等[19]通过筛选和组装适当的生物传感器元件,包括半乳糖响应启动子和苯丙氨酸响应启动子,构建了代谢产物响应回路网络。该回路的应用使得工程菌株E. coli K-12 MG1655/pJC1-mtr sensor-type1的苯丙氨酸产量与出发菌株E. coli K-12 MG1655相比提高了4.3倍。除了直接响应中间代谢物浓度以外,细胞生长状态、辅因子失衡程度等都可以用于构建生物传感器。
生物合成速度的最大化取决于单位菌体生产能力和菌体总量。通常情况下,生长和生产竞争使用同样的细胞资源,需要进行合理的安排和均衡,才能实现生物合成速度整体的最大化。能够对细胞密度进行响应的基因回路——群体感应系统(quorum sensing,QS)为调控生长和生产的关系提供了新的手段。例如,科研人员借助特征性启动子和不同强度核糖体结合位点,组装源自Pantoea stewartii的部分量子感应元件,实现了在特定时间和细胞密度下pfk-1等基因表达的开与关,工程菌株E. coli L19S的肌醇产量与静态控制相比提高了5.5倍[20]。借助QS回路控制aroK表达的工程菌株E. coli L19SA,其莽草酸的产量由0提高到105 mg/L[20]。
4 结语代谢网络模型及基于代谢网络模型的计算设计方法为代谢工程菌株改造提供了新的方法和策略。通过这些计算设计方法,人们可以选择合适的外源基因构建新途径实现从无到有的新产品合成,也可以确定改造靶点优化已有产品的得率,或者通过基因回路将细胞发酵培养过程中的状态环境变化与特定基因的表达相关联,使细胞适应不同的目标而始终保持最优状态,最大化目标产物的生成。随着人们对细胞机理和相关调控机制的了解不断深入,有可能建立全细胞模型对细胞内代谢物、蛋白和基因表达水平的变化进行精确描述和准确预测,实现完全模型指导下的代谢工程策略设计。
| [1] |
Thiele I, Palsson B O. A protocol for generating a high-quality genome-scale metabolic reconstruction[J]. Nat Protoc, 2010, 5(1): 93-121. DOI:10.1038/nprot.2009.203 |
| [2] |
Galanie S, Thodey K, Trenchard I J, et al. Complete biosynthesis of opioids in yeast[J]. Science, 2015, 349(6252): 1095-1100. DOI:10.1126/science.aac9373 |
| [3] |
Zhang X, Tervo C J, Reed J L. Metabolic assessment of E. coli as a Biofactory for commercial products[J]. Metab Eng, 2016, 35: 64-74. DOI:10.1016/j.ymben.2016.01.007 |
| [4] |
Chatsurachai S, Furusawa C, Shimizu H. An in silico platform for the design of heterologous pathways in nonnative metabolite production[J]. BMC Bioinformatics, 2012, 13: 93. DOI:10.1186/1471-2105-13-93 |
| [5] |
Hadadi N, Hafner J, Shajkofci A, et al. ATLAS of biochemistry:A repository of all possible biochemical reactions for synthetic biology and metabolic engineering studies[J]. ACS Synth Biol, 2016, 5(10): 1155-1166. DOI:10.1021/acssynbio.6b00054 |
| [6] |
Tokic M, Hadadi N, Ataman M, et al. Discovery and evaluation of biosynthetic pathways for the production of five methyl ethyl ketone precursors[J]. ACS Synth Biol, 2018, 7(8): 1858-1873. DOI:10.1021/acssynbio.8b00049 |
| [7] |
Orth J D, Thiele I, Palsson B O. What is flux balance analysis?[J]. Nat Biotechnol, 2010, 28(3): 245-248. DOI:10.1038/nbt.1614 |
| [8] |
McCloskey D, Palsson B O, Feist A M. Basic and applied uses of genome-scale metabolic network reconstructions of Escherichia coli[J]. Mol Syst Biol, 2013, 9: 661. |
| [9] |
Lin Z, Zhang Y, Yuan Q, et al. Metabolic engineering of Escherichia coli for poly(3-hydroxybutyrate) production via threonine bypass[J]. Microb Cell Fact, 2015, 14: 185. DOI:10.1186/s12934-015-0369-3 |
| [10] |
Bogorad I W, Lin T S, Liao J C. Synthetic non-oxidative glycolysis enables complete carbon conservation[J]. Nature, 2013, 502(7473): 693-697. DOI:10.1038/nature12575 |
| [11] |
Yang X, Yuan Q, Zheng Y, et al. An engineered non-oxidative glycolysis pathway for acetone production in Escherichia coli[J]. Biotechnol Lett, 2016, 38(8): 1359-1365. DOI:10.1007/s10529-016-2115-2 |
| [12] |
Zheng Y, Yuan Q, Yang X, et al. Engineering Escherichia coli for poly-(3-hydroxybutyrate) production guided by genome-scale metabolic network analysis[J]. Enzyme Microb Technol, 2017, 106: 60-66. DOI:10.1016/j.enzmictec.2017.07.003 |
| [13] |
Meadows A L, Hawkins K M, Tsegaye Y, et al. Rewriting yeast central carbon metabolism for industrial isoprenoid production[J]. Nature, 2016, 537(7622): 694-697. DOI:10.1038/nature19769 |
| [14] |
Wall M E, Hlavacek W S, Savageau M A. Design of gene circuits:lessons from bacteria[J]. Nat Rev Genet, 2004, 5(1): 34-42. DOI:10.1038/nrg1244 |
| [15] |
Zhang F, Keasling J. Biosensors and their applications in microbial metabolic engineering[J]. Trends Microbiol, 2011, 19(7): 323-329. DOI:10.1016/j.tim.2011.05.003 |
| [16] |
Xu P. Production of chemicals using dynamic control of metabolic fluxes[J]. Curr Opin Biotechnol, 2017, 53: 12-19. |
| [17] |
Brockman I M, Prather K L. Dynamic metabolic engineering:New strategies for developing responsive cell factories[J]. Biotechnol J, 2015, 10(9): 1360-1369. DOI:10.1002/biot.201400422 |
| [18] |
Brockman I M, Prather K L. Dynamic knockdown of E. coli central metabolism for redirecting fluxes of primary metabolites[J]. Metab Eng, 2015, 28: 104-113. DOI:10.1016/j.ymben.2014.12.005 |
| [19] |
Mahr R, von Boeselager R F, Wiechert J, et al. Screening of an Escherichia coli promoter library for a phenylalanine biosensor[J]. Appl Microbiol Biotechnol, 2016, 100(15): 6739-6753. DOI:10.1007/s00253-016-7575-8 |
| [20] |
Gupta A, Reizman I M, Reisch C R, et al. Dynamic regulation of metabolic flux in engineered bacteria using a pathway-independent quorum-sensing circuit[J]. Nat Biotechnol, 2017, 35(3): 273-279. DOI:10.1038/nbt.3796 |




