2. 中国科学院 重大科技任务局 北京 100864;
3. 中国科学院深圳先进技术研究院 合成生物学研究所 定量合成生物学研究中心 深圳 518055
2. Bureau of Major R & D Programs, Chinese Academy of Sciences, Beijing 100864, China;
3. Center for Quantitative Synthetic biology, Institute of Synthetic Biology, Shenzhen Institutes of Advanced Technology, Chinese Academy of Sciences, Shenzhen 518055, China
DAI Junbiao Director of Shenzhen Key Laboratory of Synthetic Genomics and Center for Synthetic Genomics, Institute of Synthetic Biology (iSynBio), Shenzhen Institutes of Advanced Technology (SIAT), Chinese Academy of Sciences (CAS).He received his Bachelor degree from Nanjing University in 1997 and Ph.D.in molecular, cellular and developmental biology from Iowa State University in 2006.His research interests lie in synthetic biology and he is one of the key members in synthetic yeast consortium (Sc2.0).More than 40 original research papers have been published on Science, Cell, Nature, and so on, and 8 patents have been applied/authorized.He is the winner of Albert Lehninger Research Award from Johns Hopkins University and the Thousand Youth Talents Program in 2011.In 2017, he was awarded the National Science Fund for Distinguished Young Scholars.E-mail:junbiao.dai@siat.ac.cn
生命科学的迅速发展使得我们从生物遗传信息的“读取”阶段进入到后基因组时代,基因组的“改写”乃至“全新设计”正逐渐成为现实。以设计创造新生命体为目标的合成生物学在此背景下迅速发展,并在医药、制造、能源等领域显现了巨大的应用前景。基因组的从头合成和针对天然基因组的规模改造分属合成基因组学和基因编辑领域,均为当前合成生物学研究的热点。合成基因组学涉及基因组的从头设计、构建和功能表征等,属于自下而上的生物学研究策略;而基因编辑侧重于通过对现有基因组进行删除、替换、插入等分子操作,进而改写遗传信息,属于自上而下的生物学研究策略。以上两者的有机结合将极大地推动生物制造、疾病治疗等领域的革新;同时二者也将为后基因组时代,功能基因组学的研究提供强有力的技术手段。新生命体系的从头设计与合成不仅需要基因组序列的合成、拼接及转移等技术,也需要高效率、低脱靶率的编辑技术以实现在基因组上进行大规模的编辑改造。在不断的探索研究中,基因编辑技术已经从最初依赖细胞自然发生的同源重组,发展到几乎可在任意位点进行的靶向切割,其操作的简易和高效极大地推动了物种遗传改造的发展。基因编辑可为合成生命的进一步改造提供手段,为新物种的创造提供更多的可能性。
1 基因编辑的原理基因编辑技术是对生物体DNA断裂的现象及其修复机制的应用。作为一种常见的分子生物学事件,在分裂活跃的哺乳动物细胞中,DNA双链断裂(DNA double-strand breaks,DSBs)每天会发生[1-4]。DSBs发生后细胞可以通过多种方式进行修复,包括经典的非同源末端修复(non-homologous end joining,NHEJ),选择性末端修复(alternative end joining,a-EJ),单链退火修复(single-strand annealing,SSA)和同源重组修复(homologous recombination,HR)[4]。HR可以进行精确无误的修复,但是需要同源模板的存在[5-7];NHEJ则是将很大程度上没有同源性的两个DNA末端直接连接实现修复[7],此过程中,两个末端在大多数情况下都会发生若干核苷酸的缺失,是一种不精确的修复机制[8]。而作为辅助性的修复机制,a-EJ和SSA均需要更大幅度的末端单链切除[4],这也会导致遗传信息的丢失。基于DNA断裂修复的原理,如果在细胞中人为提供特定的同源重组模板,待目标DNA自然发生或者人为诱导产生DSBs,触发同源重组修复,就有机会把特定DNA序列进行删除或者插入外源基因。在不提供同源模板的情况下,利用NHEJ、a-EJ及SSA的不精确修复机制可以实现基因的突变和敲除。传统的基因编辑借助细胞内自然发生的DSBs实现靶向整合[9],达到基因敲除、替换等目的。然而,在真核生物细胞里面,通过自发双链断裂实现目的基因编辑的概率通常低至百万分之一[10, 11]。人为使用化学诱导剂或者辐射处理等方法,或者使用转座子技术也可以实现基因的突变,但是这些突变是随机的,需要后续进行大量的筛选工作来获得所需的基因型。定点基因编辑技术是进行基因功能研究和物种定向改造的优选策略。
2 重组核酸酶介导的基因编辑技术 2.1 锌指核酸酶技术人工核酸酶技术的发展使人为定点诱导DSBs成为现实,其中锌指核酸酶技术(zinc finger nucleases,ZFNs)就是一个里程碑式的突破,也称为第一代基因编辑技术。ZFN由锌指蛋白(zinc finger protein,ZFP)和FokⅠ内切酶的核酸酶结构域组成,前者负责识别,后者负责切割DNA。ZFP是自然存在的蛋白结构,其由锌指结构(zinc finger,ZF)组成,ZF能识别特定的3个连续碱基对[11](图 1a),因此可通过串联ZF的数量调整ZFN的识别特异性。FokⅠ通过N端与ZFP连接,由于FokⅠ以二聚体的形式发挥切割作用,ZFN使用时需要成对设计[12]。作为新型基因编辑工具,ZFN从2001年开始被陆续用于不同物种的基因编辑[11, 13]。
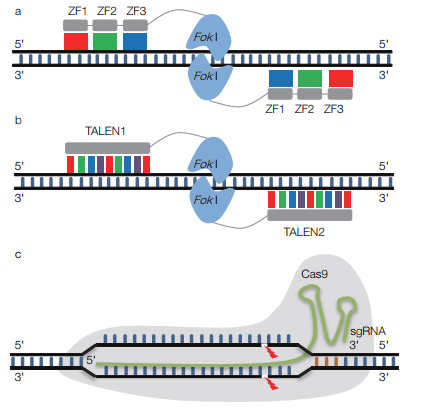
|
| 图 1 ZFN、TALEN及CRISPR/Cas9基因编辑技术 (a)锌指核酸酶(ZFN)基因编辑技术;(b)TALEN基因编辑技术;(c)CRISPR/Cas9基因编辑技术 |
ZFN技术将基因编辑引领进了不再单纯依赖自然发生DSBs的时代,但其存在很大的局限性,如成本高、难以实现多靶点编辑等[11]。而TALE(transcription activator like effector)基序的发现催生了第二代基因编辑技术——TALENs(TALE nucleases)。TALEN的构造与ZFN类似,由TALE基序串联成决定靶向性的DNA识别模块,与FokⅠ结构域连接而成。与ZF基序不同,一个TALE基序识别一个碱基对(图 1b),因此串联的TALE基序与所识别的碱基对是一一对应的关系[11]。研究发现,对于相同的靶点TALENs有与ZFNs相同的切割效率,但是毒性通常比ZFNs的低,另外其构建也比ZFNs容易[11]。然而,TALENs在尺寸上要比ZFNs大得多,而且有更多的重复序列,其编码基因在大肠杆菌中组装更加困难[11]。
3 RNA引导的基因编辑技术 3.1 CRISPR/Cas9技术CRISPR/Cas系统原本是细菌和古菌进化出来用于抵御外来病毒及质粒DNA的适应性免疫系统。Ⅱ型CRISPR/Cas系统依赖于外源DNA片段在规律成簇的短间隔回文重复(clustered regularly interspaced short palindromic repeat,CRISPR)位点整合,其经过转录及剪切后产生短的CRISPR RNAs(crRNAs),crRNA与反式转录的crRNA(trans-activating crRNA,tracrRNA)退火结合,然后引导Cas9(CRISPR associated protein 9,Cas9)蛋白介导序列特异性的外源DNA降解[11, 14-16]。Jinek等[17]发现Cas9发挥靶向切割作用所依赖的crRNA和tracrRNA可以融合为一体,作为sgRNA(single guide RNA)(图 1c)。随后,若干研究组陆续报道CRISPR/Cas9系统可用于人类细胞的靶向基因编辑[18-21]。与ZFNs和TALENs技术相比,CRISPR/Cas9的设计要简单得多,而且成本很低,对于相同的靶点,CRISPR/Cas9有相当甚至更好的靶向效率[11]。
3.2 CRISPR/Cas9衍生技术随着CRISPR/Cas系统的研究不断深入,Cas9核酸酶的催化机制也被揭示[17],并且可以通过特定位点氨基酸的突变获得单链靶向剪切功能的Cas9切刻酶(Cas9 nickase,Cas9n)或者全失活的Cas9(dead Cas9,dCas9),而这些不同的Cas9核酸酶,衍生出了适用范围更为广泛的基因编辑系统。
(1)CRISPR/Cas9-nickase基因编辑技术。 CRISPR/Cas9系统使用的crRNA能容受一定程度的错配导致脱靶效应的产生,限制了CRISPR/Cas9系统在高精度编辑中的应用[22]。为了提高Cas9编辑系统的精度,Ran等[22]巧妙利用Cas9 D10A突变体具备切刻酶活性的特性,设计了“一个位点,双sgRNA靶向”的策略,即CRISPR/Cas9-nickase基因编辑技术(图 2a)。其原理与ZFN和TALEN类似,两个Cas9n/sgRNA复合物同时靶向一个位点,分别切割其中一条DNA链,实现双链断裂,诱导NHEJ或者HR修复。使用这种策略,细胞系中基因编辑的脱靶效率最高可以降低近4个数量级。
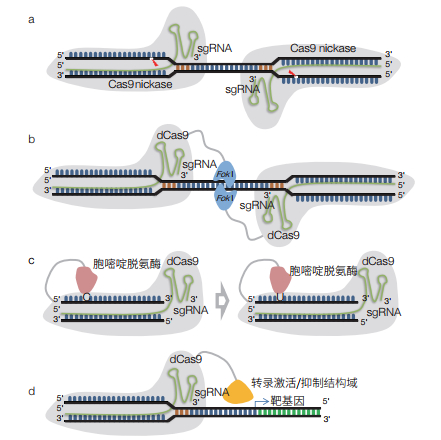
|
| 图 2 系列CRISPR/Cas9衍生基因编辑技术 (a)CRISPR/Cas9-nickase(Cas9切刻酶)基因编辑技术;(b)CRISPR/dCas9-FoKⅠ,基因编辑技术;(c)基于CRISPR/dCas9的单碱基编辑技术;(d)基于CRISPR/dCas9的转录调控技术 |
(2)CRISPR/dCas9-FoKⅠ基因编辑技术。同样是为了解决CRISPR/Cas9技术的脱靶问题,Guilinger等[23]采用了基于dCas9的策略。理论上dCas9/sgRNA只能起到单纯的靶向引导作用,无法诱导DNA的断裂,类似于ZFN或者TALEN中的DNA结合结构域。为了实现DNA的切割,其引入的FokⅠ核酸内切酶的切割结构域(图 2b),与dCas9连接做成融合蛋白fCas9,这与ZFN及TALEN的设计策略如出一辙。在人类细胞的基因编辑中,fCas9的特异性比野生型Cas9要高出140倍以上。而在高度类似的脱靶位点上,fCas9的特异性要比Cas9n至少高出4倍。fCas9的应用将进一步丰富Cas9工具箱,提供更完善的基因编辑工具。
(3)基于CRISPR/dCas9的单碱基编辑技术。人类大多数的遗传病与基因的点突变有关[24],如果能通过精确的手段进行修复,可能会带来新的治疗策略。在提供同源重组模板的情况下,定点突变可以通过CRISPR/Cas9来实现,但是其诱导的NHEJ修复可能带来的碱基随机插入、缺失是潜在的危险因素。Cas9的突变体Cas9n、dCas9无切割双链DNA的功能,但可以发挥寻靶定位作用;如果有能催化特定碱基转换的蛋白/结构域可用,则可参考CRISPR/dCas9-FoKⅠ蛋的设计,构建CRISPR/Cas9n/dCas9导向的单碱基编辑技术。Liu课题组将源自大鼠的胞嘧啶脱氨酶(APOBEC1)与dCas9融合(图 2c),发现其可以定点将C转变为U,然后在后续的DNA复制或者修复作用下实现C : G碱基对到T :A的转变[24]。随后日本神户大学的Kondo课题组[25]及我国上海交通大学的常兴课题组[26]也报道了类似研究成果。为了实现A: T到G : C的转换,Liu课题组采用蛋白质进化工程手段对大肠杆菌的tRNA腺苷脱氨酶(TadA)进行改造,他们获得的第7代腺嘌呤碱基编辑器(adenine base editors,ABEs)能高效介导A : T碱基对到G : C的转变[27]。即,C到T以及G到A碱基之间的自由转换已经实现,将来有可能做到4种碱基的任意转换。
(4)基于CRISPR/dCas9的基因表达调控技术。除了直接对DNA进行编辑,CRISPR/Cas系统在基因表达调控上也可发挥作用[28, 29]。例如,利用dCas9无核酸内切酶活性但仍能与DNA结合的特点,可直接阻碍其结合DNA与其他因子的结合,影响基因的表达(图 2d)。如果将转录抑制因子或者激活因子与dCas9融合,则可以实现靶基因的抑制与激活,为基因功能研究提供灵活的操作手段。
3.3 CRISPR/Cas12a基因编辑技术CRISPR/Cas9技术受富含G碱基的PAM序列限制,不能实现任意序列的靶向。同时Cas9蛋白分子量过大,某些情况下不便使用。实际上几乎所有的古细菌和众多的细菌都采用CRISPR/Cas机制进行免疫防御[30],其中包含多种CRISPR/Cas系统。已经表征过的Ⅱ类CRISPR/Cas系统,均属于采用Cas9家族核酸酶作为效应因子的Ⅱ型CRISPR/Cas系统,而在普氏菌和弗朗西斯氏菌属(Prevotella和Francisella 1)中存在另一个Ⅱ类CRISPR/Cas系统,其被归为Ⅴ型CRISPR/Cas系统[30]。2015年,张锋团队报道Ⅴ型系统中的Cpf1(CRISPR from Prevotella and Francisella 1)(现称“Cas12a”)是有功能的细菌免疫机制并能在人类细胞中介导有效的基因编辑[30]。CRISPR/Cas12a具有CRISPR/Cas9没有的优点[30],其中之一就是Cas12a需要的是富含T碱基的PAM序列,有助于其在基因组富含A/T碱基的物种中使用。上海科技大学的陈佳课题组将无DNA切割活性的dCas12a与大鼠源的胞嘧啶脱氨酶(APOBEC1)融合,发现其与基于Cas9的碱基编辑器类似,能有效地催化人类细胞中C到T碱基的转换[31]。由于识别的是富含T碱基的PAM序列,基于Cas12a的碱基编辑系统能与基于Cas9的碱基编辑系统互补,为相关基础研究及将来的临床应用提供更全面的技术条件。
3.4 基于CRISPR/Cas系统的RNA编辑技术除了对DNA进行编辑,张锋课题组发现CRISPR蛋白家族的C2c2(现称Cas13a)可以靶向切割RNA[32],随后他们证实Cas13a可在哺乳动物细胞中靶向降低RNA的水平[33]。利用Cas13a的RNA靶向功能,CRISPR/Cas13a系统被开发为RNA检测器用于疾病诊断[34]。张锋团队后续又发现了Cas13b[35],其同样具有RNA靶向和编辑功能。另外,Cas13c和Cas13d也具有类似功能[36]。RNA编辑技术的建立,进一步拓展了CRISPR/Cas基因编辑技术的应用范围。
4 DNA介导的基因编辑工具根据碱基互补配对原则,及引物设计原理,Oligo DNA理论上也可以用于引导核酸内切酶靶向切割双链DNA。类似于ZFN和TALEN,可用Oligo DNA替代其DNA结合结构域,关键是需要找到Oligo DNA与切割结构域FoKⅠ的合适连接方式。或者是寻找类似于Cas9的天然蛋白,本身具备核酸内切酶功能,同时能结合一定长度的引导Oligo DNA。由于DNA本身有更好的稳定性,Oligo DNA制备成本低,可以简化基因编辑的操作程序,Oligo DNA引导的基因编辑工具有其他编辑工具不可比拟的优点,值得深入研究开发。特别是当前主流基因编辑工具全为国外专利,对今后国内基因编辑相关产品的上市不利,具备自主知识产权的基因编辑技术亟待开发。
2016年9月,Genome Biology刊登了我国南京大学研究团队研发的新型基因编辑工具:结构引导的核酸内切酶(structure-guided endonuclease,SGN)[37]。SGN是典型的Oligo DNA引导基因编辑工具,本质上也是重组核酸酶的设计与应用。其N端是能识别DNA 3′末端翘翼(3′ flap)的核酸内切酶(flap endonuclease-1,FEN-1),C端则是FoKⅠ核酸内切酶的DNA切割结构域(Fn1)。SGN通过识别引导DNA(guide DNA,gDNA)与特点靶点结合产生的3′ flap进行定位切割。不同于CRISPR/Cas9及其系列衍生技术,SGN没有PAM限制,理论上可以靶向任何序列。实验结果显示SGN的切割活性不依赖于靶点的序列,其可用于斑马鱼基因编辑但是效率尚有很大的提升空间。SGN作为我国科学家的原创性研究结果,其优点无疑显著,如:gDNA非常容易获得并且可根据需要精确控制其用量;可以通过调整gDNA的长度将错配的可能降到最低等。
5 我国基因编辑技术发展面临的挑战与机遇近年来,我国在基因编辑技术方面取得了瞩目的进展。但是我们要清醒地认识到,目前基因编辑,尤其是CRISPR/Cas9相关的核心专利基本都是掌握在其他国家手中。未来基因编辑技术及基因编辑的细胞制品等走向临床应用,以及基因编辑农作物走向市场所产生的巨大利润都会因此而受到重大损失。开发具备自主知识产权的核心技术才有可能在这场生物技术革命中获得发展,包括对现有基因编辑技术的缺陷进行修正,以及通过技术组合等,抢先获得现有基因编辑技术的改进版本和增强版。同时也借助生物信息学手段挖掘潜在的CRISPR核酸酶和DNA引导的核酸酶等,开发新型基因编辑技术。此外,基因编辑技术,尤其是CRISPR/Cas技术已经广泛用于各物种的基因编辑,除了常见的模式生物,如线虫、果蝇、斑马鱼、小鼠等,还有猪、狗、猴等大型动物,以及水稻、小麦等常见农作物。将现有基因编辑技术拓展到其他生物也是新的突破,尤其对于我国特有的生物资源,其过程往往涉及技术的调整和改进,一方面可以产生新的技术,另一方面可为目标生物的功能基因组研究以及遗传改良提供有效手段。
目前,CRISPR/Cas9等基因编辑技术已经显示巨大的应用价值,但是其在编辑效率、精确度及脱靶效应等方面,以及其走向临床应用等尚有很多问题需要解决。病毒载体可应用于人类基因治疗,但是其装载容量有限,常规的Cas9蛋白过大,不利于使用。一方面可以从自然界寻找更小的Cas9蛋白,另一方面可以通过基因工程手段进行适当的删减。而在减少脱靶效应方面,除了上述提到的CRISPR/Cas9-nickase和CRISPR/dCas9-FoKⅠ基因编辑技术,还可以对Cas9蛋白本身进行定点突变等来增强其特异性。这些工作充满着挑战性,但也是我们发展的机遇和突破口。
近年来,我国科学家在基因编辑领域取得了长足进展。上述提及常兴课题组和陈佳课题组在碱基编辑技术上的突破,此外,哈尔滨工业大学黄志伟课题组和中国科学院生物物理研究所王艳丽课题组在Cas/sgRNA复合物的结构解释上也作出了突出贡献,可为基因编辑原理的理解提供重要参考。中国科学院遗传与发育生物学研究所高彩霞课题组等则在植物基因编辑上取得重要进展。中国科学院广州生物医药与健康研究院赖良学课题组、昆明理工大学季维智课题组、中国科学院神经科学研究所杨辉课题组等,利用基因编辑技术成功获得多种疾病动物模型。我国在新型基因编辑技术开发上也取得了一定突破,DNA引导的基因编辑技术有着独特的优势,值得加大投入以解决编辑效率低等问题。
此外,合成生物学作为新近迅速发展的交叉学科,已在生物医药、能源、新材料等领域展现越来越广泛的应用潜力。基因组合成和基因编辑,涉及的操作广度、深度不同,技术体系也不同。但其本质上都是通过遗传改造,获得具有特定功能的生命体,服务科研与生产,二者有机融合是水到渠成的趋势。比如,在合成的基因组中可以引入基因编辑体系,为新生物体进一步改造及应用提供更多的可能性。SCRaMbLE(synthetic chromosome rearrangement and modification by loxP-mediated evolution)[38]是一种快速的染色体重排修饰技术,可以加速生物体的进化,其本质上是一种非定点的诱导型基因编辑技术。生物的进化往往需要漫长的历史过程,难以快速获得具备某类性状的物种。合成生物学可以通过基因组设计,引入特定的功能模块,以获得目标工程细胞;然而,基于目前的成本和技术限制,尽管可以有很多候选基因组设计,想要获得一个大容量合成细胞库并从中筛选获得最优设计的可能性不大。而SCRaMbLE是一种潜在的高性价比手段。Sc2.0计划拟在合成的酵母基因组中插入约5 000个loxPysm位点[39]。该计划近期发表的系列成果[40-46]表明,SCRaMbLE系统可以诱导获得增强型工业用酵母菌株,提目标代谢物的产量等。
当前,首个全人工合成的真核生物——Sc2.0已经接近完成,更高等生物的全基因组合成也已经提上日程[47]。然而,超大基因组的合成尚有很多挑战。通过基因编辑对现有物种进行局部基因组改造实际为传统基因工程的升级,其不大可能实现全面的基因组改写。就目前而言,基因组合成与基因组编辑的结合将是一个十分具有潜力的研究方向,可解决目前超大基因组从头合成面临的各项困难,为合成基因组学在超大基因组物种基础研究及物种改造中的应用提供可能。这也是我国在合成生物学领域及基因编辑领域可以把握的契机。
| [1] |
Martin G M, Smith A C, Ketterer D J, et al. Increased chromosomal aberrations in first metaphases of cells isolated from the kidneys of aged mice[J]. Isr J Med Sci, 1985, 21(3): 296-301. |
| [2] |
Lieber M R, Karanjawala Z E. Ageing, repetitive genomes and DNA damage[J]. Nat Rev Mol Cell Biol, 2004, 5(1): 69-75. DOI:10.1038/nrm1281 |
| [3] |
Lieber M R. The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway[J]. Annu Rev Biochem, 2010, 79: 181-211. DOI:10.1146/annurev.biochem.052308.093131 |
| [4] |
Chang H H Y, Pannunzio N R, Adachi N, et al. Non-homologous DNA end joining and alternative pathways to double-strand break repair[J]. Nat Rev Mol Cell Biol, 2017, 18(8): 495-506. DOI:10.1038/nrm.2017.48 |
| [5] |
Paques F, Haber J E. Multiple pathways of recombination induced by double-strand breaks in Saccharomyces cerevisiae[J]. Microbiol Mol Biol Rev, 1999, 63(2): 349-404. |
| [6] |
Sung P, Klein H. Mechanism of homologous recombination:mediators and helicases take on regulatory functions[J]. Nat Rev Mol Cell Biol, 2006, 7(10): 739-750. DOI:10.1038/nrm2008 |
| [7] |
Hartlerode A J, Scully R. Mechanisms of double-strand break repair in somatic mammalian cells[J]. Biochem J, 2009, 423(2): 157-168. DOI:10.1042/BJ20090942 |
| [8] |
Lieber M R, Ma Y, Pannicke U, et al. Mechanism and regulation of human non-homologous DNA end-joining[J]. Nat Rev Mol Cell Biol, 2003, 4(9): 712-720. DOI:10.1038/nrm1202 |
| [9] |
Lee A H, Symington L S, Fidock D A. DNA repair mechanisms and their biological roles in the malaria parasite Plasmodium falciparum[J]. Microbiol Mol Biol Rev, 2014, 78(3): 469-486. DOI:10.1128/MMBR.00059-13 |
| [10] |
Capecchi M R. Altering the genome by homologous recombination[J]. Science, 1989, 244(4910): 1288-1292. DOI:10.1126/science.2660260 |
| [11] |
Chandrasegaran S, Carroll D. Origins of programmable nucleases for genome engineering[J]. J Mol Biol, 2016, 428(5): 963-989. DOI:10.1016/j.jmb.2015.10.014 |
| [12] |
Remy S, Tesson L, Menoret S, et al. Zinc-finger nucleases:a powerful tool for genetic engineering of animals[J]. Transgenic Res, 2010, 19(3): 363-371. DOI:10.1007/s11248-009-9323-7 |
| [13] |
Bibikova M, Carroll D, Segal D J, et al. Stimulation of homologous recombination through targeted cleavage by chimeric nucleases[J]. Mol Cell Biol, 2001, 21(1): 289-297. DOI:10.1128/MCB.21.1.289-297.2001 |
| [14] |
Nishimasu H, Ran F A, Hsu P D, et al. Crystal structure of Cas9 in complex with guide RNA and target DNA[J]. Cell, 2014, 156(5): 935-949. DOI:10.1016/j.cell.2014.02.001 |
| [15] |
Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea[J]. Science, 2010, 327(5962): 167-170. DOI:10.1126/science.1179555 |
| [16] |
Mali P, Esvelt K M, Church G M. Cas9 as a versatile tool for engineering biology[J]. Nat Methods, 2013, 10(10): 957-963. DOI:10.1038/nmeth.2649 |
| [17] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dualRNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [18] |
Mali P, Yang L, Esvelt K M, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033 |
| [19] |
Cong L, Ran F A, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [20] |
Cho S W, Kim S, Kim J M, et al. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease[J]. Nat Biotechnol, 2013, 31(3): 230-232. DOI:10.1038/nbt.2507 |
| [21] |
Jinek M, East A, Cheng A, et al. RNA-programmed genome editing in human cells[J]. ELife, 2013, 2: e00471. DOI:10.7554/eLife.00471 |
| [22] |
Ran F A, Hsu P D, Lin C Y, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J]. Cell, 2013, 154(6): 1380-1389. DOI:10.1016/j.cell.2013.08.021 |
| [23] |
Guilinger J P, Thompson D B, Liu D R. Fusion of catalytically inactive Cas9 to FokⅠ nuclease improves the specificity of genome modification[J]. Nat Biotechnol, 2014, 32(6): 577-582. DOI:10.1038/nbt.2909 |
| [24] |
Komor A C, Kim Y B, Packer M S, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature, 2016, 533(7603): 420-424. DOI:10.1038/nature17946 |
| [25] |
Nishida K, Arazoe T, Yachie N, et al. Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems[J]. Science, 2016, 353(6305). DOI:10.1126/science.aaf8729 |
| [26] |
Ma Y, Zhang J, Yin W, et al. Targeted AID-mediated mutagenesis (TAM) enables efficient genomic diversification in mammalian cells[J]. Nature methods, 2016, 13(12): 1029-1035. DOI:10.1038/nmeth.4027 |
| [27] |
Gaudelli N M, Komor A C, Rees H A, et al. Programmable base editing of A·T to G·C in genomic DNA without DNA cleavage[J]. Nature, 2017, 551(7681): 464-471. DOI:10.1038/nature24644 |
| [28] |
Qi L S, Larson M H, Gilbert L A, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022 |
| [29] |
Gilbert L A, Larson M H, Morsut L, et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes[J]. Cell, 2013, 154(2): 442-451. DOI:10.1016/j.cell.2013.06.044 |
| [30] |
Zetsche B, Gootenberg J S, Abudayyeh O O, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system[J]. Cell, 2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038 |
| [31] |
Li X, Wang Y, Liu Y, et al. Base editing with a Cpf1-cytidine deaminase fusion[J]. Nat Biotechnol, 2018, 36(4): 324-327. DOI:10.1038/nbt.4102 |
| [32] |
Abudayyeh O O, Gootenberg J S, Konermann S, et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector[J]. Science, 2016, 353(6299). DOI:10.1126/science.aaf5573 |
| [33] |
Abudayyeh O O, Gootenberg J S, Essletzbichler P, et al. RNA targeting with CRISPR-Cas13[J]. Nature, 2017, 550(7675): 280-284. DOI:10.1038/nature24049 |
| [34] |
Gootenberg J S, Abudayyeh O O, Lee J W, et al. Nucleic acid detection with CRISPR-Cas13a/C2c2[J]. Science, 2017, 356(6336): 438-442. DOI:10.1126/science.aam9321 |
| [35] |
Smargon A A, Cox D B T, Pyzocha N K, et al. Cas13bis a Type Ⅵ-B CRISPR-associated RNA-guided RNase differentially regulated by accessory proteins Csx27 and Csx28[J]. Mol Cell, 2017, 65(4): 618-630. DOI:10.1016/j.molcel.2016.12.023 |
| [36] |
Yan W X, Chong S R, Zhang H B, et al. Cas13d is a compact RNA-targeting Type Ⅵ CRISPR effector positively modulated by a WYL-domain-containing accessory protein[J]. Mol Cell, 2018, 70(2): 327-339. DOI:10.1016/j.molcel.2018.02.028 |
| [37] |
Xu S, Cao S S, Zou B J, et al. An alternative novel tool for DNA editing without target sequence limitation:the structure-guided nuclease[J]. Genome Biol, 2016, 17(1): 186. DOI:10.1186/s13059-016-1038-5 |
| [38] |
Dymond J S, Richardson S M, Coombes C E, et al. Synthetic chromosome arms function in yeast and generate phenotypic diversity by design[J]. Nature, 2011, 477(7365): 471-476. DOI:10.1038/nature10403 |
| [39] |
Jones S. SCRaMbLE does the yeast genome shuffle[J]. Nat Biotechnol, 2018, 36(6): 503-503. DOI:10.1038/nbt.4164 |
| [40] |
Blount B A, Gowers G O F, Ho J C H, et al. Rapid host strain improvement by in vivo rearrangement of a synthetic yeast chromosome[J]. Nat Commun, 2018, 9(1): 1932. |
| [41] |
Jia B, Wu Y, Li B Z, et al. Precise control of SCRaMbLE in synthetic haploid and diploid yeast[J]. Nat Commun, 2018, 9(1): 1933. DOI:10.1038/s41467-018-03084-4 |
| [42] |
Shen M J, Wu Y, Yang K, et al. Heterozygous diploid and interspecies SCRaMbLEing[J]. Nat Commun, 2018, 9(1): 1934. DOI:10.1038/s41467-018-04157-0 |
| [43] |
Wu Y, Zhu R Y, Mitchell L A, et al. In vitro DNA SCRaMbLE[J]. Nat Commun, 2018, 9(1): 1935. DOI:10.1038/s41467-018-03743-6 |
| [44] |
Liu W, Luo Z Q, Wang Y, et al. Rapid pathway prototyping and engineering using in vitro and in vivo synthetic genome SCRaMbLE-in methods[J]. Nat Commun, 2018, 9(1): 1936. |
| [45] |
Luo Z Q, Wang L H, Wang Y, et al. Identifying and characterizing SCRaMbLEd synthetic yeast using ReSCuES[J]. Nat Commun, 2018, 9(1): 1930. DOI:10.1038/s41467-017-00806-y |
| [46] |
Hochrein L, Mitchell L A, Schulz K, et al. L-SCRaMbLE as a tool for light-controlled Cre-mediated recombination in yeast[J]. Nat Commun, 2018, 9(1): 1931. DOI:10.1038/s41467-017-02208-6 |
| [47] |
Boeke J D, Church G, Hessel A, et al. The Genome Project-Write[J]. Science, 2016, 353(6295): 126-127. DOI:10.1126/science.aaf6850 |




