生命的起源是什么?最原始的生命是怎样形成的?人类能否创造生命?从古至今,科学家和哲学家们一直在不断探索并希望解决这些困惑。大量的微生物基因组序列的信息,以及各种组学的研究,包括基因组、转录组、蛋白质组、代谢组等,并由此构建出的调控网络和代谢网络模型,促进了我们对生命的理解,也让我们意识到生命系统的复杂性。然而,在这些复杂的调控网络和代谢途径中,哪些是生存必需的呢?怎样设计和构建一个简约的生命体系呢?设计并构建简约基因组将为合成生物学和人造生命的研究提供一个重要的基础。
2010年美国科学家Venter和他的科研团队发表了世界上首个由化学合成基因组控制的原核生物支原体[1]。在此基础上,他们通过大量的设计与测试,构建了目前已知的基因组最小的支原体[2]。与国际合成生物学的迅猛发展相比,我国的合成生物学研究起步较晚。2011年,我国启动了10项合成生物学相关的“973”项目,极大促进我国在人工生物体系的设计和合成,细胞生长功能群的模块化及可控运行,发展重构、编辑简小基因组,以及在底盘细胞上“即插即用”型地高效组装生物学模块的合成生物学使能技术等方面的研究,并取得了较好的进展。我国科学家(主要是天津大学、清华大学和深圳华大基因研究所的科学家)与美国等国家的科研机构共同推动了“酵母基因组合成国际计划”,并于2017年完成了其中4条染色体的从头设计与全合成[3-6],这标志着中国的基因组设计合成能力达到了国际水平。2018年,中国科学院合成生物学重点实验室覃重军研究团队与合作者通过大胆假设驱动与合成生物学工程化的方法,将酿酒酵母的16条染色体融合为1条,创建了国际上首例单条染色体的真核细胞[7]。这一成果完全由中国科学家独立完成,是合成生物学具有里程碑意义的重大突破。
近年来技术的发展,使得DNA合成成本降低,尤其是关键的DNA大片段拼接以及基因组转移技术的进步,使得从头全合成和拼接完整的微生物基因组获得了成功,为人工合成生命细胞打破了技术屏障。人工合成生命细胞对于理解生命的本质和发展改造生命具有重要的意义。
1 人工设计与合成小基因组原核细胞Venter研究组于2010年报道了人工全合成和组装1.08 Mbp的丝状支原体JCVI-syn1.0基因组,并将其移植到受体山羊支原体细胞,创造出新的细胞具有丝状支原体细胞的信息和特征[1]。这是一个庞大的工程,参与该项目有20多人,历经了15年,花费了4 000万美元,从而最终完成了这一重大的人工合成基因组的任务。在全基因组合成和拼接中,需要多级的质量监控,以保证合成出来的基因组序列的精确性。例如,Venter团队曾经因为一个碱基的错误而使得细胞不能存活——在调控染色体复制的生存必需基因dnaA中的一个碱基缺失造成基因的读码框移位,一个小的错误曾经耽误了他们不少的时间。这个例子表明细胞的存活是需要精确调控的。Venter等人合成的基因组几乎跟天然的基因组一样,要理解、设计和改造生命系统,仅仅拷贝并合成一个天然的小基因组是不够的。
那么如何设计并合成一个比天然更小的基因组呢?Venter团队从全合成的生长比较快的1.08 Mbp的丝状支原体JCVI-syn1.0基因组出发,通过多轮的设计—合成—测试,最后成功合成出了531 kbp且可存活的最小基因组的JCVI-syn3.0[2]。这个3.0版本比天然最小的583 kbp的生殖道支原体基因组更小,生长却是更快,代时仅有后者的1/5。这被认为是接近于最小基因组的版本了。
这个成功的背后是超大量的设计、合成和测试工作。首先,他们发现基于已有知识设计最小基因组是不可行的。通过整合现有的转座子突变和敲除数据,以及分子生物学累积的知识,从1.08 Mbp的丝状支原体基因组JCVI-syn1.0中找到了可敲除的440个生存非必需基因,使得基因组减小到了483 kbp。他们将这个预设的483 kbp的最小基因组分成8个大段,分别合成和测试功能。结果8个大段中仅1个大段是有功能的,但是该段的设计改造使得细菌生长很不好。导致前期的设计失败的一个重要原因是在生存必需基因(e)及非必需基因(n)之间,还存在对生长重要的准-生存必需基因。根据准-生存必需基因对生长的影响情况,他们还分了非常影响生长的基因(ie)、影响生长的基因(i),以及轻微影响生长的基因(in)。他们认为应该保留生存必需的基因e以及影响生长的ie和i,只把基本不影响生长的in和n作为敲除的备选基因。除了准-生存必需基因之外,还存在执行生存必需功能的冗余基因。比如,基因A和B都能执行生存必需功能E,单敲除A或者B都不影响生存必需功能E,那么我们一般会认为A和B都是生存非必需的。但是A和B的同时缺失将导致生存必需功能E的缺失而使得细菌不能存活。因此,需要大量的测试工作来寻找这些准-生存必需基因,以及执行生存必需功能的冗余基因。
值得注意的是,在基因组减小的过程中,基因组大小和生长速度之间有一个平衡。随着基因组的减小,生长速度急速减慢。人工设计及合成出来的531 kbp支原体最小基因组JCVI-syn3.0代时为180 min,比1.08 Mbp的支原体基因组JCVI-syn1.0的60 min的代时要慢很多。并且,它不像出发菌株那样可以在液体培养基中形成均一的混悬液,而是容易沉淀。显微镜和电镜下可以看到JCVI-syn3.0细胞容易形成长的细丝状的网络结构,也会形成大的泡囊体。因此,这个接近于最小基因组的细胞的生长还是有缺陷的,不那么完美。但是,无论如何,这是一个成功设计与合成最小基因组的范例。
2 人工设计与合成原核模式生物大肠杆菌基因组大肠杆菌是被最广泛研究的原核模式生物。它生长快速,遗传操作相对简单——很多的遗传操作技术都是首先在大肠杆菌中建立的。对大肠杆菌在组学水平的大量研究技术及生物信息学分析和建模促进了我们对大肠杆菌生物系统的理解,使我们能对大肠杆菌进行更有理性的改造,从而使它成为我们想要的超级菌株,以用于工业应用生产所需要的产物。基因组减小化可以降低基因组的复杂度,也减少了非必需的代谢途径,有利于整合异源的高效生产元件以获得高产的优化菌株。大肠杆菌的基因组有4—5 Mbp,远比1 Mbp的支原体基因组大。因此通过类似的“自下而上”从头合成的方法来构建大肠杆菌简约基因组并进行测试会很困难。因此,大部分与大肠杆菌减小基因组相关的研究报道都是通过“自上而下”的基因组删减途径来进行的。由于工业应用需求导向,我们希望构建的是具有快速生长、基因组稳定的大肠杆菌菌株。基因组的减小化与生长速度之间存在一定的平衡。因此,我们对大肠杆菌简小基因组的研究并不是一味追求基因组的最小化,而是小而强大。
“自上而下”的基因组删减策略是首先通过生物信息学方法分析生存必需基因[8],比如,分析天然进化的含小基因组的微生物,或者用比较基因组学分析等。第二步是用高通量基因敲除方法鉴定生存必需基因。有了这些数据,我们可以确定基因组哪些区域是生存必需的,哪些区域是生存非必需的、可敲除的。通过对基因组中的生存非必需的区域进行大段删减的测试,测试可敲除的区域再合并到一个菌株中,这样可以逐步构建一系列基因组减小化的菌株。
目前主要有3类基因组减小的大肠杆菌菌株的构建[9]:① Delta系列是通过大段敲除生存非必需区域,迅速将大肠杆菌基因组减小。但是由于没有很理性的设计,最后构建出来的菌株存在生长缺陷。②相比而言,MGF系列只累积不影响生长的较小片段的敲除,因此最后构建出来的菌株生长状况良好。③ MDS系列着重敲除插入序列、原噬菌体等不影响生长且有助于基因组稳定的基因。因此,最后构建出来的菌株被认为是含有clean genome(干净的基因组)的,且菌株生长状况良好。
2016年,美国科学家Church研究组最近报道了全合成的密码子简约化的大肠杆菌基因组[9]。我们知道,1个遗传密码子是由3个碱基组成,共有64个密码子,除了3个终止密码子外,其他61个密码子对应于20种氨基酸。1种氨基酸可以对应1到多个密码子。全基因组范围的同义密码子置换可以构建具有遗传隔离并具有新的生物功能的生命体。遗传隔离是指由于新合成出来的生命体少了一些密码子,使得外源DNA,包括来源于病毒、质粒或其他细胞的DNA,进入生物体内后可能无法正常表达,从而使生物体对感染和基因水平转移不敏感。此外,可以利用置换出来的密码子来表达20个氨基酸以外的非标准氨基酸,从而合成具有新化学活性的蛋白。Church研究组通过重编程大肠杆菌基因组,将大肠杆菌基因组的7个密码子,包括丝氨酸、亮氨酸、精氨酸的各2个密码子以及1个终止子,用同义密码子置换,从而将基因组使用的密码子从64个降为57个[9]。这是个很大胆的举动,因为这7个密码子的置换一共涉及了6万多处改动。他们将大肠杆菌基因组分成87个约50 kbp的片段,并对其中的55个片段进行合成与分别测试。他们发现91%的生存必需基因在这样的改动之后仍能保持功能且不影响生长。这表明了基因组的可塑性还是很大的。
3 人工设计与合成真核生物酿酒酵母基因组酿酒酵母是最简单的单细胞真核模式生物,对其基因组的重新设计及人工合成并进行功能研究将极大地推动人类对生命理解及改造能力。“酿酒酵母基因组合成计划”(Sc2.0项目)是人类首次尝试改造和从头合成真核生物。Sc2.0项目是由美国科学院院士Jef Boeke发起,由多国研究机构参与并分工协作,对真核模式生物酿酒酵母16条染色体进行人工重新设计和化学再造,是继原核支原体基因组合成项目之后,合成生物学领域的又一重大标志性国际合作项目。最先完成的是3号染色体的全合成与替换,2014年在Science杂志报道[10]。2017年,Science杂志以封面、专刊的形式同时发表了介绍“人工合成酵母基因组计划”已完成2号、5号、6号、10号和12号这5条染色体的从头设计与全合成的研究,研究人员把经过设计的人工合成染色体导入酿酒酵母中,并保证带有人工合成染色体的酵母菌仍能够正常存活。我国天津大学、清华大学、华大基因研究院和Boeke院士一起合作,完成了其中4条染色体的全合成及功能验证[3-6],进一步证明了人工合成生命体系的可行性。对酵母16条染色体的人工设计内容,包括终止密码TAG被TAA代替,引入了loxP重组位点可以允许基因组的快速改变,引入PCR检测标记及限制性内切酶识别位点,以及敲除tRNA及重复序列等。这些设计及改变不会对酵母的生长有明显的影响等。前期人工合成和构建的酿酒酵母染色体引入了很多loxP重组位点,诱导Cre切割loxP位点,任意两个loxP重组位点之间的序列可以发生倒转、缺失或重复。这个技术被称为SCRaMbLE,可以用于染色体快速重排。通过定向筛选,可以很快找到对微生物细胞工厂性能提升、生命快速进化和人类染色体异常疾病等研究有用的菌株。
2018年,以中国科学院覃重军研究组为主的研究团队通过15轮的染色体融合将酿酒酵母天然的16条染色体逐一融合,人工创建了只含有单条线型染色体的酵母细胞(SY14)。在染色体的人工逐一融合过程中,构建了从SY0到SY13的染色体数目不断减少的一系列中间菌株,最终构建的SY14菌株删除了15个着丝粒、30个端粒、19个长的重复序列[7](图 1)。单条染色体在三维结构上有极大的改变,但是单染色体的酵母具有与野生型细胞相似的转录组和表型组。不仅如此,单染色体的酵母还保持了减数分裂的能力,虽然在产孢和孢子存活率上略有降低。这些都表明单染色体的酿酒酵母可以有正常的细胞功能。
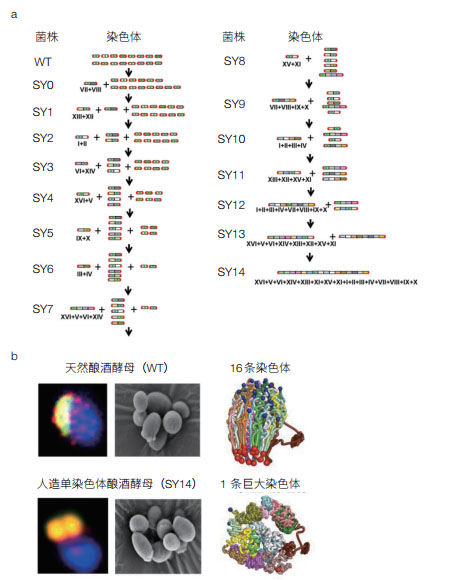
|
| 图 1 人工创建的单染色体真核酵母 a)单染色体真核酵母的创建过程。通过15轮的染色体融合将酿酒酵母天然的16条染色体逐一融合,人工创建了只含有单条线型染色体的酵母细胞。(b)人工创建的单染色体酵母细胞虽然在染色体三维结构上有巨大改变,但在细胞功能上与天然酵母几乎一致 |
生命系统的复杂性使其难以分析和操作,对基因组的简约化设计和工程化构建不仅有利于对生命起源、进化与代谢调控的研究,也为生物技术的应用提供了合适的表达宿主。从基因组简约化的角度来看,对基因组生存非必需的基因的删减可能并非人类想象的那么简单。尽管对于大部分细菌和真菌基因组来说,有80%— 90%的基因是生存非必需的。但是,这些生存非必需基因,如执行生存必需功能的冗余基因等,在不同程度的简约化基因组背景下可能是生存必需的。对天然小基因组原核细胞支原体的“自下而上”的人工重构及简约化设计与功能测试表明,即使是天然已经很紧凑的基因组,仍然可以继续删减一些基因,得到更简约的基因组。但是,目前对简约基因组的人工设计并没有很好的策略,更多需要依赖大量的“设计—合成—测试”的工作。对于原核模式生物“自上而下”的基因组的删减工作也是进展较慢,尤其是大肠杆菌的基因组简约化工程已进行了近20年的研究,不同的课题组从不同的菌株出发进行基因组连续删减获得的菌株(基因组仅为约3 Mbp),均远远无法接近理论上的最小基因组(约300个基因,大小约0.3 Mbp)。这表明人类的理性设计简约化基因组人造生命还处于比较初期的阶段。
然而,从其他方面进行基因组的简约化的成功尝试,比如对酿酒酵母多条染色体的端粒和着丝粒的删减,并将天然的16条染色体融合为1条,使得染色体间及染色体内的相互作用都大大简约化。这提示我们,天然复杂生命系统是可以通过人工设计和改造进行简约化并以全新的生命形式表现出来的。
| [1] |
Gibson D G, Glass J I, Lartigue C, et al. Creation of a bacterial cell controlled by a chemically synthesized genome[J]. Science, 329, 5987: 52-56. |
| [2] |
Hutchison 3rd C A, Chuang R Y, Noskow V N, et al. Design and synthesis of a minimal bacterial genome[J]. Science, 2016, 351: aad6253. DOI:10.1126/science.aad6253 |
| [3] |
Zhang W, Zhao G, Luo Z, et al. Engineering the ribosomal DNA in a megabase synthetic chromosome[J]. Science, 2017, 355: aaf3981. DOI:10.1126/science.aaf3981 |
| [4] |
Xie Z X, Li B Z, Mitchell L A, et al. "Perfect" designer chromosome V and behavior of a ring derivative[J]. Science, 2017, 355: aaf4704. DOI:10.1126/science.aaf4704 |
| [5] |
Wu Y, Li B Z, Zhao M, et al. Bug mapping and fitness testing of chemically synthesized chromosome Ⅹ[J]. Science, 2017, 355: aaf4706. DOI:10.1126/science.aaf4706 |
| [6] |
Shen Y, Wang Y, Chen T, et al. Deep functional analysis of synⅡ, a 770-kilobase synthetic yeast chromosome[J]. Science, 2017, 355: aaf4791. DOI:10.1126/science.aaf4791 |
| [7] |
Shao Y, Lu N, Wu Z, et al. Creating a functional singlechromosome yeast[J]. Nature, 2018, 560: 331-335. DOI:10.1038/s41586-018-0382-x |
| [8] |
薛小莉, 覃重军. 大肠杆菌最小基因组分析和删减进展[J]. 生命科学, 2013, 10: 978-982. |
| [9] |
Ostrov N, Landon M, Guell M, et al. Design, synthesis, and testing toward a 57-codon genome. Science, 353(6301): 819-822. http://www.ncbi.nlm.nih.gov/pubmed/27540174
|
| [10] |
Annaluru N, Muller H, Mitchell L A, et al. Total synthesis of a functional designer eukaryotic chromosome. Science, 344(6179): 55-58. http://www.ncbi.nlm.nih.gov/pubmed/24674868
|




