2. 中国科学院 重大科技任务局 北京 100864;
3. 中国科学院遗传与发育生物学研究所 北京 100101;
4. 中国科学院上海生命科学研究院 植物生理生态研究所 上海 200032;
5. 中国农业大学 生物学院 北京 100083
2. Bureau of Major R & D Programs, Chinese Academy of Sciences, Beijing 100864, China;
3. Institute of Genetics and Developmental Biology, Chinese Academy of Sciences, Beijing 100101, China;
4. Institute of Plant Physiology & Ecology, Shanghai Institutes for Biological Sciences, Chinese Academy of Sciences, Shanghai 200032, China;
5. College of Biological Sciences, China Agricultural University, Beijing 100083, China
JING Haichun Principal Investigator, Assistant Director, and a member of the Academic Committee of Institute of Botany, Chinese Academy of Sciences.He obtained Ph.D.degree from the University of Groningen in 2005, and was selected into CAS Hundred Talents Program in 2010.His recent research interests mainly focus on the molecular genetic research on the biomass energy traits of sweet sorghum and maize. E-mail:hcjing@ibcas.ac.cn
玉米,属C4高产作物,是世界上主要的粮食作物,也是“饲料之王”和重要的工业及能源原料。随着社会的进步、人民生活水平的提高和畜牧业的不断壮大,玉米在食品、饲料、能源及医疗等方面的实用价值正在被不断发掘。然而,在世界人口不断增长、人均耕地面积和水资源逐渐减少、极端气候越来越频繁的情况下,目前玉米的生产情况已无法满足人们的需求。同时,随着生活水平的提高,人们对于粮食营养品质的需求加剧,联合国粮农组织的战略口号也从“粮食安全”转变成为“粮食和营养安全”。因此,如何保证玉米产量和品质的提高就成为当前玉米育种的关键问题。
我国是世界上主要的玉米生产国之一,玉米总产量和单位面积产量仅次于水稻,在我国人民生活和国民经济中有着举足轻重的作用。早期通过科学家们的不断努力,我国在玉米育种方面取得了非常显著的成绩,极大促进了国民经济的发展。然而,随着国外种业不断进入我国市场,我国玉米的市场竞争力明显下降,国内玉米原有种质资源狭窄、育种手段落后等问题也日趋突出;近年来,在一些玉米主产区,干旱、低温、土壤贫瘠和病虫害等各种生物和非生物胁迫频发,导致玉米大面积减产。2016年,原国家农业部对玉米种植面积进行了结构性调整,大幅度调减“镰刀弯”等非优势产区的面积,计划到2020年将继续调减5 000万亩,使玉米种植面积稳定在5亿亩。随着国家对种植业结构的调整,我国玉米种植面积增长的空间十分有限,如何利用有限的资源保障国家粮食安全供给是今后我国玉米育种研究所面临的巨大挑战。为了不断提高玉米的产量,改善玉米品质以确保国家粮食安全、满足人民更高质量的需求,并且为新能源发展提供支撑,必须通过遗传改良来选育高产、高抗、优质玉米新品种,其将成为解决这些问题的关键,同时也是未来玉米育种的重点发展方向。
2 玉米育种技术的主要发展历程 2.1 杂交育种技术——玉米育种的“基石”杂交育种技术是在有性杂交基础上,通过前期的表型选择和后期的遗传重组,利用杂种优势来进行育种。该方法无法直接检测材料的基因型,而主要是通过对玉米材料的表型性状来推测基因型。虽然早期的科研工作者利用杂交育种技术培育了一系列高产、优质、抗逆的品种,然而随着社会的发展,传统育种技术的缺陷逐渐显现出来:育种周期太长,如果要定向改良一个农艺性状,从选择目标表型性状进行杂交到培育出新品种一般需要7—8年的时间;育种效率较低,选育1个新品种往往需要配制上千个杂交组合;对杂交后代的表型难预测,传统常规育种工作在很大程度上主要依赖于育种工作者的经验,对育种材料的遗传基础、配合力、环境影响了解较少,因此很难预测杂交后代的表现。此外,玉米自交系材料遗传基础狭窄、种间生殖隔离的存在等,在一定程度上限制了人们对特定材料的改良[1-3]。
2.2 分子育种技术——玉米育种的“加速器”分子育种技术是将生物遗传学理论与常规育种相结合,通过利用基因型对目标材料进行性状改良,从而培育符合人们所需的新品种。早期的分子育种技术主要包括分子标记辅助育种和转基因育种[4-8]。分子标记辅助育种主要以DNA多态性为基础,通过标记目的基因进行性状改良,为实现对基因型的直接选择提供了可能。转基因技术则是通过分子生物学手段将目的基因转到受体材料中,在短时间内获得所需的生物性状。目前转基因技术在抗除草剂及抗病虫害等方面发挥了非常重要的作用,其中抗除草剂基因包括草胺膦乙酰转移酶(BAR)基因和5-烯醇丙酮莽草酸-3-磷酸合成酶(EPSPS)基因,而抗虫基因主要是Bt基因。转基因技术从1987年开始到现在已获得了极大的成功,有些抗虫、抗除草剂品种已进入商业化推广或生产阶段[9]。与传统杂交育种技术相比,分子标记辅助育种和转基因育种被认为是定向选择、改善目标性状的高效、精准的方法,可以缩短育种时间,在一定程度上提升了我国的玉米育种水平;缺点是涉及到的基因通常为单个或少数基因,只能针对简单的农艺性状进行遗传改良。
2.3 分子模块设计育种——玉米育种的新突破随着生物技术的不断发展,分子设计育种逐步成为了玉米育种中主要的育种方式。该育种理念最早由Peleman[10]于2003年提出,我国科学家也先后开展了针对作物重要农艺性状的分子设计及新品种培育的推广等重大项目。然而,由于作物大多数重要的农艺性状都是由多基因控制的复杂性状,更为重要的是控制这些复杂性状的基因调控网络通常呈现“模块化”的特性[2, 11],而现有的分子育种技术无法解决复杂农艺性状的遗传改良这一难题。因此,分子模块设计育种这一新型育种理念由此被提出。分子模块设计育种实际上是分子育种的一个新的类型,主要以基因组学、分子生物学、系统生物学、合成生物学及计算生物学为基础,将多学科的发展与玉米育种相结合,以培育具有目标性状的新品种。分子模块设计育种主要包括3个方面:①发掘和解析分子模块,对控制复杂性状的重要基因或QTL(quantitative trait locus)进行功能研究;②对发掘的模块进行有机耦合,并进行理论模拟和功能预测,提出最佳选配策略;③在全基因组水平上进行多模块优化组装,实现复杂性状的定向改良[12]。无论是从理论上还是技术上都可以说分子模块设计育种是我国玉米育种事业的一个新突破,是实现玉米复杂性状分子改良的基础,也将在今后的实践过程中成为新品种创制的主力军。
3 分子模块设计育种在玉米育种中的实际应用及重要成果目前采用分子设计育种技术已经成为了国际上玉米育种的大趋势。世界玉米种业巨头“拜耳-孟山都” “陶氏杜邦”及利马格兰等均在通过生物技术手段,将分子设计育种广泛应用到玉米育种中,有效加快育种进程,其研发投入约占营业额的15%。早在2007年,先锋良种国际有限公司(以下简称“先锋公司”)借助玉米全基因组测序技术测定了600多个优良玉米自交系的10 000个基因组;目前,先锋公司和孟山都公司已拥有上万个SNPs(single nucleotide polymorphisms)标记[13]。同时,这些种业公司还积极开发先进的表型检测设备,对玉米的田间表型进行精准收集和分析,挖掘切实可用的分子模块,为分子设计育种提供信息支撑。与此同时,我国科学家对玉米分子设计育种也进行了一系列深入探索,在骨干自交系的全基因组测序和SNP挖掘[14]、控制玉米籽粒油分[15]、抗病虫害[16]关键分子模块的功能解析及新品种创制等方面取得了重要进展。
近年来,水资源不足已成为世界各国农业产区存在的现实问题。2012年,干旱曾导致美国中西部地区玉米产量下降20%。玉米作为旱地作物,整个生育期的需水量较大;而玉米的抗旱性是受多基因控制的数量性状,遗传机制非常复杂。我国是世界上最缺水的国家之一,特别是在我国主要的玉米生产区,水分不足严重影响了玉米的生长和产量。因此,挖掘、解析玉米中水分胁迫调控的分子模块,培育水分利用率高的品种,对我国农业生产过程中玉米的增产稳产具有十分重要的意义。针对这一目标,2013年中国科学院战略性先导科技专项(A类)“分子模块设计育种创新体系”(以下简称“分子模块先导专项”)专门开展了“玉米水分高效利用分子模块解析”方面的工作,院内多家单位从苗期、开花期、根系、叶夹角以及干旱-盐碱互作等多个方面进行了深入研究,挖掘到一批具有重要生产应用价值的分子模块,并将部分关键模块成功导入底盘品种中,主要进展如下。
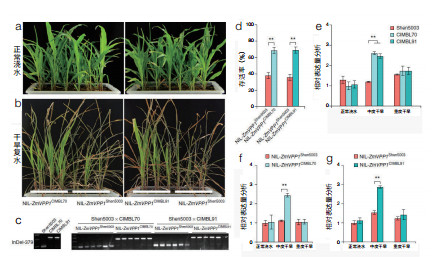
3.1 苗期水分高效利用分子模块得到挖掘和解析秦峰研究组对来自全球的368份玉米自交系的苗期耐旱性进行统计,结合全基因组关联分析发现了83个遗传变异位点(解析至42个候选基因)与玉米苗期抗旱性显著相关,并对挖掘到的2个分子模块的功能变异位点和遗传效应进行了深度解析。研究发现,在干旱敏感的材料中,ZmNAC111基因启动子区域携带1个82 bp的微型转座子插入,该转座子可通过小RNA介导的DNA和组蛋白甲基化抑制ZmNAC111的表达(图 1)。提高ZmNAC111的表达量可促进干旱胁迫下气孔的关闭,气孔导度和蒸腾速率显著降低,诱导一些重要的水分胁迫应答基因表达上调,从而提高水分利用率,增强玉米的耐旱性[17]。而ZmVPP1基因,编码一个定位于液泡膜上的焦磷酸水解酶。在抗旱玉米自交系中,ZmVPP1基因启动子前端插入了366个碱基的DNA片段(InDel-379)。将抗旱材料的ZmVPP1基因导入干旱敏感的材料中可有效提高玉米苗期的抗旱性[18]。目前该课题组对于其他候选基因的分析工作还在进行中,这些研究成果将为玉米耐旱性的遗传改良及培育玉米耐旱新品种提供极其重要的基因资源。
谢旗研究组通过对PH4CV(耐旱品种)与F9721(旱敏感品种)构建的181个重组自交系(RILs)的耐旱处理实验,选取极端耐旱株系RIL70和极端敏感株系RIL93的叶片进行了比较转录组学分析,利用SIMM(simultaneously identification of multiple mutations)方法计算每个株系特异的渐渗区间,最终确定了2个耐旱分子模块[19]。目前,已将耐旱相关分子模块导入骨干自交系“郑58”和“昌7-2”中。利用苗期耐盐碱鉴定技术标准,王柏臣研究组对69个玉米自交系进行了苗期耐盐碱性的鉴定,通过关联分析确定了一个耐盐碱的分子模块ZmTLP,能解释表型变异的15%。同时,还组配了“郑58”和“昌7-2”的重组自交系以进行耐盐碱性的筛选,相关研究工作还在进行中。
3.2 开花期耐旱分子模块的挖掘及应用研究表明,玉米开花前后对干旱胁迫最为敏感,田间持水量轻微程度的降低都可能严重影响玉米的各种生理特性,最终造成玉米产量的损失[20]。为了探寻玉米开花期耐旱的分子机制,解析玉米生殖生育期水分胁迫调控的分子模块,我们通过对前期收集的1 100份玉米自交系材料在甘肃、新疆和内蒙古等地的多年多点干旱处理实验,对叶片持绿性、雌雄穗开花间隔时间以及产量相关性状进行了深入调查,并对243份核心种质资源进行芯片检测和GWAS分析,挖掘到了44个与花期耐旱显著相关的遗传位点。同时,结合转录组测序分析,确定了1个核心分子模块。将其导入东北底盘品种“吉单27”后,叶片的光合速率显著提高、持绿耐旱性及稳产性明显增强。
3.3 根系耐旱分子模块的挖掘根系作为植物感知、吸收水分的主要器官,与耐旱性密切相关。一般来说,发达的根系更有利于作物在干旱条件下吸收土壤深层的水分,增强其抵抗干旱胁迫的能力。为了深入了解成株期根系构型的变化在玉米耐旱中的作用机制,寻找与玉米水分高效利用密切相关的分子模块,谢旗研究组从150多份自交系中筛选出根系构型差异较大的23个自交系(12个根少的自交系和11个根多的自交系)进行多年多点的干旱实验,最终确定2号染色体上~6.66 Mb的区间对根系表型具有最大贡献率。与此同时还构建了回交群体,目前正在对包含目标区间的回交个体进行鉴定,并挖掘可能的候选分子模块。
3.4 叶夹角分子模块的解析及初步应用叶夹角是决定玉米株型的一个主要因素,与产量及耐旱性密切相关。刘宏涛研究组发现玉米叶夹角突变体WF01和WF02均是由茎尖分生组织特异表达基因RS1的异位表达所致。通过启动子序列分析,发现WF01和WF02中RS1基因的−758 bp处插入了一个1089 bp的反转录转座子Flourf长末端重复序列。此外,将这2个分子模块分别导入到“昌7-2”中,以组配改良的“郑单958”。初步测产结果表明WF01回交改育的材料与“郑单958”的产量相当;下一步将继续通过回交以纯化背景并提高种植密度,对改良系在正常及水分胁迫条件下的表现与“郑单958”进行比较分析。
通过分子模块先导专项的开展,我国科研人员在玉米分子模块设计育种方面取得了一定的进展,挖掘到多个具有重要应用价值的玉米水分高效利用分子模块,并对部分模块的作用机理、调控网络及功能进行了分析。下一步仍需要继续深入探索玉米水分胁迫调控的遗传网络,鉴定关键分子模块,挖掘优异等位变异,建立“分子模块—水分胁迫调控性状”的关联;同时通过多模块计算模拟和定向设计育种技术将这些关键分子模块进行优化设计、耦合和组装,定向改良底盘品种,缩短育种周期,实现玉米水分高效利用新品系的创制。
4 我国玉米分子模块设计育种的前景展望玉米是我国最重要的禾谷类作物之一,在我国农业资源不断减少,而人们对优质玉米需求不断增加的情况下,依靠科技创新大力发展玉米育种事业刻不容缓。由于玉米的重要农艺性状包括抗逆性、产量及品质等都是多基因控制的复杂性状,针对这些复杂农艺性状,分子标记辅助育种以及转基因技术由于仅局限于单个或几个基因的遗传改良,已不能满足当前玉米生产对优良新品种的需求;而新型的育种模式——分子模块设计育种将现代信息技术和育种工作相结合,能够将控制玉米复杂农艺性状的多个有效模块聚合,提高改良育种效率,为保障我国粮食安全提供有力的理论和技术支撑。可以预见,随着测序技术和大数据的发展,在未来的玉米遗传育种中,分子模块设计育种必将持续发挥领军作用,其中亟待解决的问题主要包括3个方面。
4.1 分子模块的挖掘及调控网络的解析在过去的几十年里,人们已挖掘并鉴定了一大批与玉米重要农艺性状密切相关的功能基因和QTL位点,包括持绿耐旱性、籽粒品质、株型以及抗病性等各个方面[21-27]。但是关于这些重要农艺性状的分子模块网络并不完全清楚,这极大阻碍了基于分子模块调控的玉米遗传改良。随着全基因组测序技术的不断发展和更新,较低的测序成本和高通量的测序数据使得大规模挖掘控制玉米复杂性状的基因信息成为了可能——很多的玉米骨干自交系已完成测序,有关玉米的生物信息学数据库的数据大幅增加[28-30];同时,转录组测序、蛋白质组测序和基因芯片等功能基因组研究平台和玉米突变体库的构建,为深入研究玉米相关功能基因的调控网络打下了良好的基础[31-35]。今后还需要借助分子生物学、基因组学以及表观修饰等技术,深入探索玉米复杂农艺性状的遗传基础及调控网络。
4.2 高通量表型的精准检测和利用表型是基因型和环境型相互作用的结果,高效、准确的表型检测和分析是玉米生产育种中十分关键的环节,也是优良品种筛选、精准育种的先决条件。随着测序技术的提高,玉米基因组学、蛋白质组学及代谢组学等快速发展,而由于玉米表型的高度复杂性和传统表型鉴定技术的限制,导致玉米表型组学研究相对滞后,也使得相当一部分基因组信息无法被充分利用。因此,发展先进的表型鉴定技术、搭建高通量表型检测平台成为了必然。国内外大型企业及科研机构先后在人工气候或大田等不同环境条件下建立了各种重要农艺性状的表型鉴定平台,可以自动化收集表型数据[36-38]。在未来的农业生产中,如何利用先进的高通量表型检测技术和平台精准收集表型数据信息,并与基因组学等多学科技术相结合,深入挖掘与玉米重要农艺性状密切相关的分子模块,以促进玉米高通量遗传育种将是今后人们关注的重点,也是实现玉米复杂农艺性状改良的理论基础。
4.3 多学科的交叉与融合玉米分子模块设计育种作为一个多学科的集合与交叉,是一项高度综合的系统工程,需要将基因组学、表型组学、计算生物学、系统生物学和合成生物学相结合,对控制玉米复杂农艺性状的分子模块进行解析、组装、耦合,并进行新型品种的分子设计和培育。但值得注意的是,目前我国玉米育种队伍比较分散,研究缺乏持续性和系统性。因此,在今后的育种工作中需要高度整合资源,加强多学科交叉和共性技术集成以加强玉米分子育种平台的建设,完善和促进玉米分子模块设计育种体系。利用分子模块设计育种技术无疑能够全面解析控制玉米复杂农艺性状的重要分子模块,通过多模块的耦合和组装模拟,实现复杂性状的定向改良,培育高产优质的玉米新品系。
5 结语我国是农业大国,玉米的持续稳定生产对于保障我国农业可持续发展和国家粮食安全具有重大的现实和战略意义。在过去的几十年里,我国玉米育种取得了举世瞩目的成绩,总产不断上升,但是单产相对较低。将分子模块设计育种充分应用到玉米育种工作中,是将分子生物学前沿知识与生产育种实践的紧密结合,对于玉米育种的发展,特别是对于玉米复杂性状的遗传改良具有重要的引领作用。玉米分子模块设计育种创新体系的建立,将有助于提升我国农作物育种的技术水平,实现从“经验育种”向“精准育种”的转化,加速我国玉米新品种的选育进程。
| [1] |
万建民. 作物分子设计育种[J]. 作物学报, 2006, 32: 455-462. DOI:10.3321/j.issn:0496-3490.2006.03.023 |
| [2] |
薛勇彪, 段子渊, 种康, 等. 面向未来的新一代生物育种技术——分子模块设计育种[J]. 中国科学院院刊, 2013, 28(3): 308-314. DOI:10.3969/j.issn.1000-3045.2013.03.003 |
| [3] |
戴景瑞, 鄂立柱. 百年玉米, 再铸辉煌——中国玉米产业百年回顾与展望[J]. 农学学报, 2018, 8(1): 74-79. |
| [4] |
黎裕, 王建康, 邱丽娟, 等. 中国作物分子育种现状与发展前景[J]. 作物学报, 2010, 36: 1425-1430. |
| [5] |
Yang D E, Jin D M, Wang B, et al. Characterization and mapping of Rpi1, a gene that confers dominant resistance to stalk rot in maize[J]. Molecular Genetics & Genomics, 2005, 274(3): 229-234. |
| [6] |
Camuskulandaivelu L, Veyrieras J B, Madur D, et al. Maize adaptation to temperate climate:Relationship between population structure and polymorphism in the Dwarf8 gene[J]. Genetics, 2006, 172(4): 2449-2463. |
| [7] |
Wang R, Yu Y, Zhao J, et al. Population structure and linkage disequilibrium of a mini core set of maize inbred lines in China[J]. Theoretical Applied Genetics, 2008, 117(7): 1141-1153. DOI:10.1007/s00122-008-0852-x |
| [8] |
Xu G J, Zhang S L, Lu R Q. Application of genetically modified technology in maize breeding[J]. Agricultural Science & Technology, 2015, 16(6): 1177-1179. |
| [9] |
杨双. 转基因作物发展状况及趋势[J]. 中国种业, 2018. DOI:10.19462/j.cnki.1671-895x.20180404.016 |
| [10] |
Peleman J D, van der Voort J R. Breeding by design[J]. Trends in Plant Science, 2003, 8: 330-334. DOI:10.1016/S1360-1385(03)00134-1 |
| [11] |
杨艳萍, 袁建霞, 董瑜. 分子模块设计育种科技将创造新一代农业生物品种[J]. 中国科学院院刊, 2013, 28(5): 585-587. |
| [12] |
薛勇彪, 王道文, 段子渊. 分子设计育种研究进展[J]. 中国科学院院刊, 2007, 22(6): 486-490. DOI:10.3969/j.issn.1000-3045.2007.06.009 |
| [13] |
赵久然, 王荣焕, 刘新香. 我国玉米产业现状及生物育种发展趋势[J]. 生物产业技术, 2016(3): 45-52. |
| [14] |
Jiao Y P, Zhao H N, Ren L H, et al. Genome-wide genetic changes during modern breeding of maize[J]. Nature Genetics, 2012, 44: 812-815. DOI:10.1038/ng.2312 |
| [15] |
Li H, Peng Z, Yang X, et al. Genome-wide association study dissects the genetic architecture of oil biosynthesis in maize kernels[J]. Nature Genetics, 2013, 45: 43-50. DOI:10.1038/ng.2484 |
| [16] |
Zuo W, Chao Q, Zhang N, et al. A maize wall-associated kinase confers quantitative resistance to head smut[J]. Nature Genetics, 2015, 47(2): 151-157. DOI:10.1038/ng.3170 |
| [17] |
Mao H, Wang H, Liu S, et al. A transposable element in a NAC gene isassociated with drought tolerance in maize seedlings[J]. Nature Communication, 2015, 6: 8326. DOI:10.1038/ncomms9326 |
| [18] |
Wang X, Wang H, Liu S, et al. Genetic variation in ZmVPP1 contributes to drought tolerance in maize seedlings[J]. Nature Genetics, 2016, 48: 1233-1241. DOI:10.1038/ng.3636 |
| [19] |
Min H, Chen C, Wei S, et al. Identification of drought tolerant mechanisms in maize seedlings based on transcriptome analysis of recombination inbred lines[J]. Frontier in Plant Science, 2016, 7: 1080-1090. |
| [20] |
Lopes M S, Araus J L, van Heerden P D, et al. Enhancing drought tolerance in C(4) crops[J]. Journal of Experimental Botany, 2011, 62(9): 3135-53. DOI:10.1093/jxb/err105 |
| [21] |
Zheng H J, Wu A Z, Zheng C C, et al. QTL mapping of maize (Zea mays) stay-green traits and their relationship to yield[J]. Plant Breeding, 2009, 128(1): 54-62. DOI:10.1111/pbr.2009.128.issue-1 |
| [22] |
Zhang Z, Yang J, Wu Y. Transcriptional regulation of zein gene expression in maize through theadditive and synergistic action of opaque2, prolamine-box binding factor, and O2 heterodimerizing proteins[J]. Plant Cell, 2015, 27: 1162-1172. DOI:10.1105/tpc.15.00035 |
| [23] |
Zhang ZY, Zheng X X, Yang J, et al. Maize endosperm-specific transcription factors O2 and PBF network the regulation of protein and starch synthesis[J]. PNAS, 2016, 113(39): 10842-10847. DOI:10.1073/pnas.1613721113 |
| [24] |
Yang Y Z, Ding S, Wang Y, et al. Small kernel2 Encodes a Glutaminase in Vitamin B6 Biosynthesis Essential for Maize Seed Development[J]. Plant Physiology, 2017, 174(2): 1127-1138. DOI:10.1104/pp.16.01295 |
| [25] |
Teng F, Zhai L H, Liu R X, et al. ZmGA3ox2, a candidate gene for a major QTL, qPH3.1, for plant height in maize[J]. Plant Journal, 2013, 73: 405-416. DOI:10.1111/tpj.12038 |
| [26] |
Li Q, Li L, Yang X, et al. Relationship. Evolutionary fate and function of two maize co-orthologs of rice GW2 associated with kernel size and weight[J]. BMC Plant Biology, 2010, 10: 143. DOI:10.1186/1471-2229-10-143 |
| [27] |
Strable J, Wallace J G, Unger-Wallace E, et al. Maize YABBY Genes drooping leaf1 and drooping leaf2 Regulate Plant Architecture[J]. Plant Cell, 2017, 29(7): 1622-1641. DOI:10.1105/tpc.16.00477 |
| [28] |
Pan Q, Li L, Yang X, et al. Genome-wide recombination dynamics are associated with phenotypic variation in maize[J]. New Phytologist, 2016, 210: 1083-1094. DOI:10.1111/nph.13810 |
| [29] |
Yang N, Xu X W, Wang R R. Contributions of Zea mays subspecies mexicana haplotypes to modern maize[J]. Nature Communications, 2017, 8(1): 1874. DOI:10.1038/s41467-017-02063-5 |
| [30] |
Yang H, Liu X, Xin M, et al. Genome-wide mapping of targets of maize histone deacetylase HDA101 reveals its function and regulatory mechanism during seed development[J]. Plant Cell, 2016, 28: 629-645. DOI:10.1105/tpc.15.00691 |
| [31] |
Lu X, Chen D, Shu D, et al. The differential transcription network between embryo and endosperm in the early developing maize seed[J]. Plant Physiology, 2013, 162: 440-455. DOI:10.1104/pp.113.214874 |
| [32] |
Shi J, Yan B, Lou X, et al. Comparative transcriptome analysis reveals the transcriptional alterations in heat-resistant and heatsensitive sweet maize (Zea mays L.) varieties under heat[J]. BMC Plant Biology, 2017, 17(1): 26. DOI:10.1186/s12870-017-0973-y |
| [33] |
Yu T, Li G, Dong S, et al. Proteomic analysis of maize grain development using iTRAQ reveals temporal programs of diverse metabolic processes[J]. BMC Plant Biology, 2016, 16(1): 241. DOI:10.1186/s12870-016-0878-1 |
| [34] |
Xu C, Ren Y, Jian Y, et al. Development of a maize 55 K SNP array with improved genome coverage for molecular breeding[J]. Molecular Breeding, 2017, 37(3): 20. DOI:10.1007/s11032-017-0622-z |
| [35] |
Lu X, Liu J, Ren W, Yang Q. Gene-indexed mutations in maize (Zea mays)[J]. Molecular Plant, 2017, 11(3): 496-504. |
| [36] |
Yang W, Duan L, Chen G, et al. Plant phenomics and high-throughput phenotyping:accelerating rice functional genomics using multidisciplinary technologies[J]. Current Opinion in Plant Biology, 2013, 16(2): 180-187. DOI:10.1016/j.pbi.2013.03.005 |
| [37] |
Langridge P. High-throuput phenotyping of nitrogen response and use in wheat with lemnatec scanalyzer 3D. Okamoto, Mamoru:International Plant and Animal Genome XXII Conference[J]. Plant and Animal Genome, 2014. |
| [38] |
郭庆华, 吴芳芳, 庞树鑫, 等. Crop 3D——基于激光雷达的作物高通量表型监测平台[J]. 中国科学:生命科学, 46(10): 1210-1221. |