2. 中国科学院大学 经济与管理学院图书情报与档案管理系 北京 100190;
3. 中国科学院遗传与发育生物学研究所 北京 100101;
4. 中国科学院种子创新研究院 北京 100101
2. Department of Library, Information and Archives Management, School of Economics and Management, University of Chinese Academy of Sciences, Beijing 100190, China;
3. Institute of Genetics and Developmental Biology, Chinese Academy of Sciences, Beijing 100101, China;
4. Institute of Seed Innovation, Chinese Academy of Sciences, Beijing 100101, China
党的十八大以来,党中央和国务院将知识产权工作提到了前所未有的高度,加强知识产权保护,是完善产权保护制度最重要的内容,也是提高中国经济竞争力最大的激励。信息领域和生命科学领域是推动知识产权发展的两大重要方向,每一次的技术突破和创新都推动着生命科学的重大飞跃。20世纪50年代DNA双螺旋结构的解析,揭开了人类对于生命体遗传信息的认知。20世纪70年代出现的重组DNA技术、特异DNA序列的体外扩增技术、基因克隆技术已然成为当今实验室的常规技术,极大地推动了现代生命科学的快速发展,使之进入到现代分子生物学时代;伴随着测序技术的不断进步,在人类基因组计划带动下,多个物种的基因组图谱得以解析,各种组学得以兴起,基因芯片、生物云计算、个性化分子诊断等各项生物产业蓬勃发展。进而,生命科学进入到后基因组时代,人类对各种复杂的生命现象有了进一步的认知。
基因编辑(gene editing),又称基因组编辑(gene editing)或基因组工程(genome engineering),是对生物体基因组特定目标基因进行修饰的一种基因工程技术。锌指核酸酶(ZFN)、转录激活因子样效应物核酸酶(TALEN)、规律间隔成簇短回文重复序列(CRISPR)等基因组编辑工具层出不穷,为基因组编辑技术的突破带来了新的革命浪潮[1-5]。CRISPR的相关序列(CRISPR/CAS)技术在这些技术的基础上,又向前推进了一大步,可以实现对DNA序列的任意切割和改造[6-11]。随着研究的深入,基因编辑技术在基因研究、疾病防治和遗传改良等方面展示出了巨大的潜力。政府资助和基因组学应用领域的增长是推动基因组编辑市场增长的关键因素。基因组编辑与基因组工程技术的进步,以及传染病和癌症的日益高发,进一步支持了这一市场的发展。
本研究通过对基因编辑相关专利和期刊文献的分析,揭示了基因编辑技术的发展现状与趋势,并基于对CRISPR技术的专利分析及其相关重要研发机构的专利布局分析,对我国基因编辑技术的发展提出了对策与建议,以期为相关领域的研究人员及决策者提供参考。
1 数据与方法 1.1 数据来源基因编辑相关专利检索数据库为德温特专利数据库(Derwent Innovation Index,DII)(专利文献范围为1963—2019年)①;相关期刊文献检索数据库为科学引文索引(Web of Science,WoS)核心合集(期刊文献范围为1900—2019年)①;相关基金资助检索自美国国立卫生研究院(NIH)。
① https://apps.webofknowledge.com.
1.2 数据集合在DII中,以ZFN、TALEN、CRISPR、gene、genomic、base edit等词及基因编辑相关的德温特手工代码组合检索(检索时间为2019年11月22日)。检索结果经过数据清洗,去除不相关信息后,得到专利6012项;其中,涉及CRISPR专利4041项,涉及TALEN专利794项,涉及ZFN专利250项。得到期刊文献22278篇;其中,涉及CRISPR 17404篇,涉及TALEN 2151篇,涉及ZFN 2717篇。
1.3 分析工具利用VOSviewer和incoPat对相关数据进行分析,包括作者/发明人合作、机构合作及国家合作等分析。incoPat深度整合全球发明智慧,完整收录全球120个国家和地区1亿多条基础专利数据,对22个主要国家的专利数据进行收录和加工处理,字段多样、数据完整。
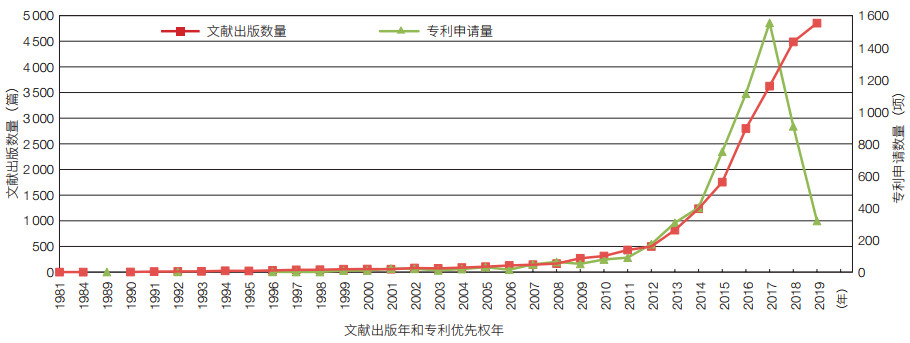
2 基因编辑技术的全球产出趋势分析通过对基因编辑相关专利的年度申请情况与相关期刊的年度发文数量进行分析(图 1),以了解基因编辑的年度产出趋势、全球重要的研究机构与国家,以及国内主要的研究机构。
|
| 图 1 基因编辑的年度专利申请与文献出版数量 |
最早关于基因编辑的专利出现于1989年。1997年专利(US781559),即“在基因组中对靶DNA序列进行修饰的培养细胞用于生产转基因动物或调控植物细胞,包括用正负选择载体转化细胞”,其最早优先权可以追溯到1989年(US397707)[12]。随后10年,每年的相关专利申请量都少于10项;2003年开始,专利申请数量逐年增加。2012年Doudna Jennifer和Emmanuelle Charpentier将CRISPR/Cas9改造成为可高效实现精准修饰靶基因特定DNA序列的低成本工具。CRISPR/Cas9的问世标志着新一代的生物核心技术问世,并使得精准调控生命过程成为可能。基因组编辑的热潮席卷了整个生命科学研究领域,生命科学研究开启了基因组编辑时代。2012年以后,每年专利申请数量超过100项,2017年达到最高峰为2 032项②。
② 由于专利申请在18个月的公开期后才给予授权;因此,2018年和2019年的数据不能代表当年全部数据。
最早与基因编辑相关的文献,出现于1981年,为Routman [13]撰写的关于大学和工业界对DNA重组所有权的争论。1990年Blum等[14]撰写关于在Kinetoplastid线粒体中RNA分子介导的RNA编辑模型,这是第一次明确在文献中提出基因编辑的概念。基因编辑相关期刊文献出版趋势与专利申请趋势基本相似,并在2019年达到最高峰为4 854篇③。
③ 由于WoS核心合集收录及数据加工的问题,2019年和2020年的数据不能覆盖当年的所有数据。
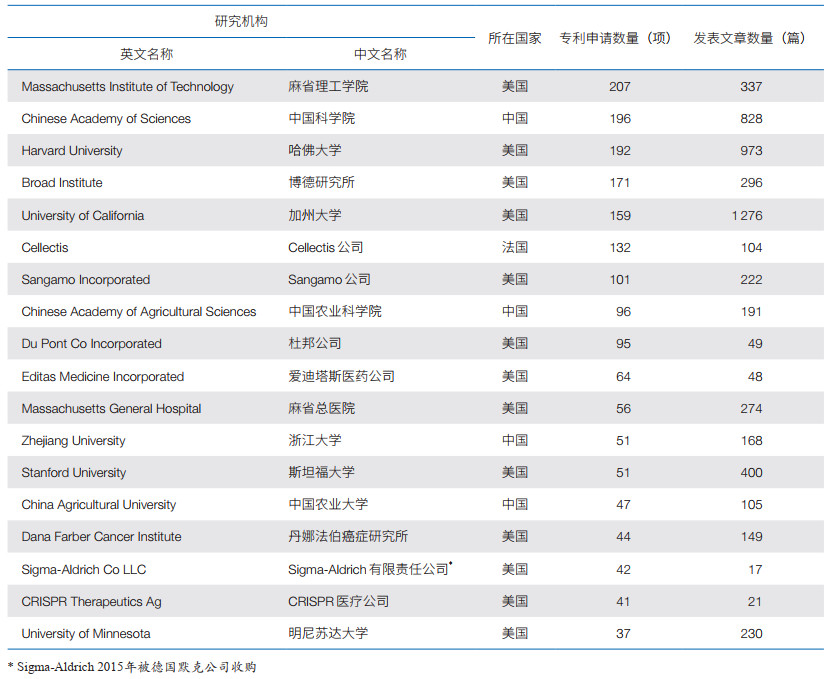
2.2 主要机构基因编辑相关产出情况美国麻省理工学院的基因编辑相关专利数量最多,为207项;其相关期刊论文为337篇,列研究机构第7。中国科学院相关专利数量为196项,列第2;相关期刊论文数量为828篇。美国哈佛大学相关专利数量列第3,为192项;相关期刊论文数量为973篇(表 1)。
图 2和3显示基因编辑全球主要机构发文合作情况。通过机构的合作发文情况,可以看出美国哈佛大学是目前基因编辑研究的核心机构,跟全球很多的高校、研究所或企业有相关合作研究;同时,法国Cellectis公司、韩国生物科技公司和美国Sangamo公司的合作发文对象相对较少,这可能是由于商业公司的主要力量在于专利技术的研发与转移转化。中国科学院是中国基因编辑研究的主导力量,跟国内多个高校和研究院所及欧美等多个国家的研究机构有紧密的合作关系。
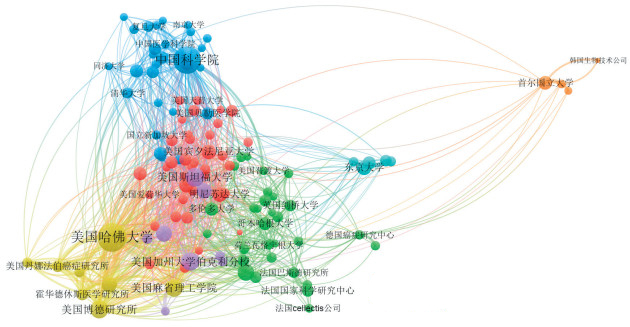
|
| 图 2 基因编辑领域全球机构发文合作的情况 |
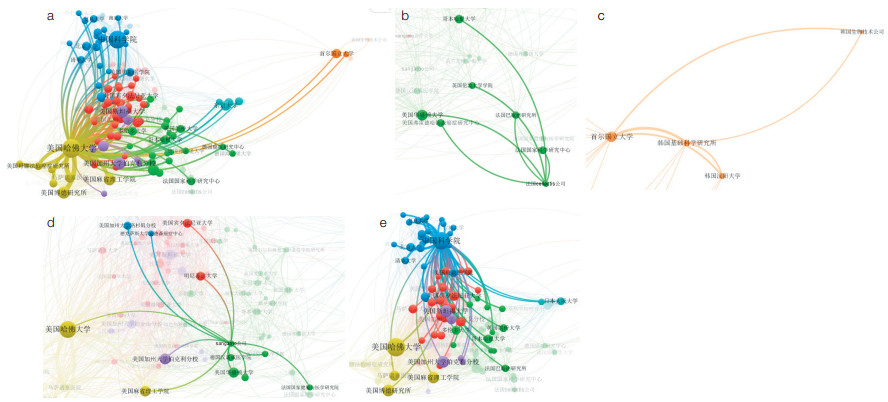
|
| 图 3 基因编辑领域主要机构合作情况 a)美国哈佛大学;(b)法国Cellectis公司;(c)韩国生物技术公司;(d)美国Sangamo公司;(e)中国科学院 |
通过对发表文章的国家进行分析,一共有129个国家参与了基因编辑的相关研究。美国、中国、德国和日本,占据了全球发文数量的前4位。在“一带一路”沿线国家中,也有不少已经参与了基因编辑的相关研究;其中,印度、俄罗斯和以色列发表的文章数量位列“一带一路”沿线国家前3名。
从论文发表国家的合作情况来看,美国仍处于全球合作的中心——美国几乎跟所有的国家都有合作,而中国是与美国合作数量最多的国家(图 4)。
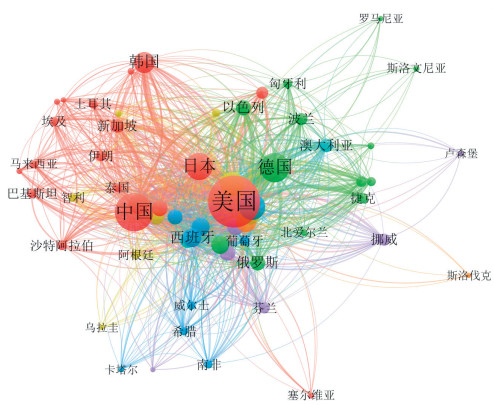
|
| 图 4 基因编辑领域国家/地区发文合作分析 |
国内基因编辑研究领域的主要机构以高校和科研院所为主;其中,中国科学院遗传与发育生物学研究所、中国科学院上海生命科学研究院、浙江大学、中国农业大学、北京大学和中山大学的专利数量最多(图 5)。

|
| 图 5 基因编辑领域中国主要机构拥有专利情况 |
中国科学院是国内主要从事基因编辑研究的单位之一,拥有基因编辑专利较多的研究所有:中国科学院遗传与发育生物学研究所60项,中国科学院上海生命科学院56项。国内其他单位:中国农业科学院,专利96项,论文191篇;北京大学,专利35项,论文190篇;清华大学,专利24项,论文127篇。
3 CRISPR基因编辑技术分析通过对CRISPR技术的专利布局情况分析,可了解目前全球的技术竞争市场和先进技术的集中国家与地区及研究人员;通过对其专利年度申请趋势和申请人年度趋势的分析,可了解CRISPR技术的发展历程。
3.1 CRISPR相关专利全球布局分析经检索,CRISPR技术相关专利族共4 041项。对4 041项专利族中的所有专利(共10 470件专利)的受理国进行分析可知,中国、美国为专利布局最多的国家(图 6)。由于专利具备地域性,在同一个地域申请的专利量越多,则覆盖的技术点越多,技术保护范围越大;相应的,对产品的保护越全面,在地域内产品的技术竞争力和市场竞争力越强大。因此,根据在中国、美国的CRISPR相关专利布局量可推知中国、美国为关注度较高的技术争夺市场。
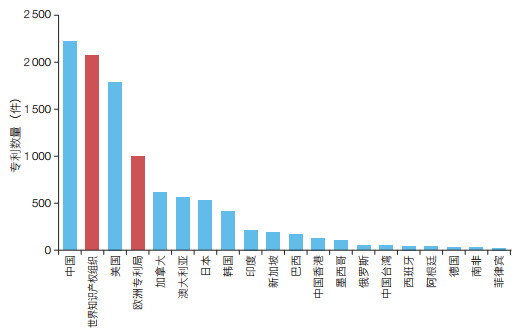
|
| 图 6 CRISPR技术专利受理数量排名前20的国家(地区)/组织 |
此外,世界知识产权组织(WO)和欧洲专利局(EP)的专利受理量也较多。由于世界知识产权组织拥有近150个成员国,一项技术在国际阶段向其提交申请后,在国家阶段可进入所有成员国;而欧洲专利局拥有近40个欧洲成员国,通过其审查的专利,均可以在其所有成员国中获得保护。因此,世界知识产权组织和欧洲专利局都是专利申请人向全球专利布局的有效途径。通过这2个组织的受理量可推知CRISPR技术实施全球专利布局的趋势。
3.2 CRISPR相关专利申请人分析在CRISPR相关专利的申请人中,美国的麻省理工学院、哈佛大学、博德研究所为主要申请人(图 7),且多为共同申请,三者合作紧密度极高。中国科学院也具备较强的研究实力,申请量位列全球第4;其中,中国科学院的遗传与发育生物学研究所、上海生命科学研究院、北京基因组研究所、生物物理研究所、植物研究所、重庆绿色智能研究院、微生物研究所、武汉病毒研究所、武汉植物园、动物研究所均有申请CRISPR技术相关专利。不过,中国科学院的各研究单元多属于单兵作战,合作关系为弱相关。
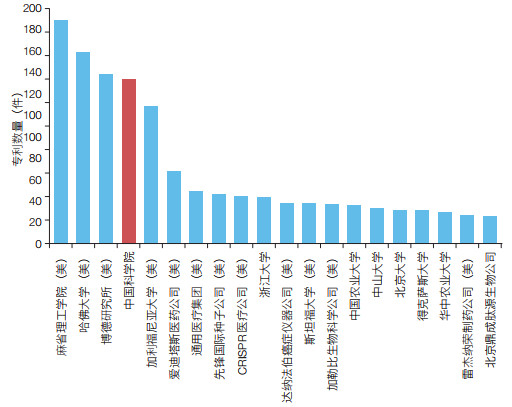
|
| 图 7 全球CRISPR技术相关专利申请量排名前20的专利申请人 |
从4 041项专利的发明人分析来看,美国麻省理工学院张锋的CRISPR相关专利申请量远高于其他发明人。美国的刘如谦(Liu D R)、Church G M、Donohoue P D和Doudna J A等研究者的专利申请量也位于前列,这表明美国已成为CRISPR技术的研究聚集地。
为通过重点国家专利的发明人分布探究研发团队实力,以及集中优势力量合作研发的潜力,分别对中国、美国、世界知识产权组织、欧洲专利局、澳大利亚、韩国和日本的CRISPR相关专利的发明人合作关系进行分析。从图 8a可以看出,中国的CRISPR相关专利发明人出现频次较为平均,在CRISPR方向的研发团队呈散乱状态,研发团队界限不清晰,未出现领军型的核心研究者或研究团队,国内合作研发和国际合作研发均呈弱相关。
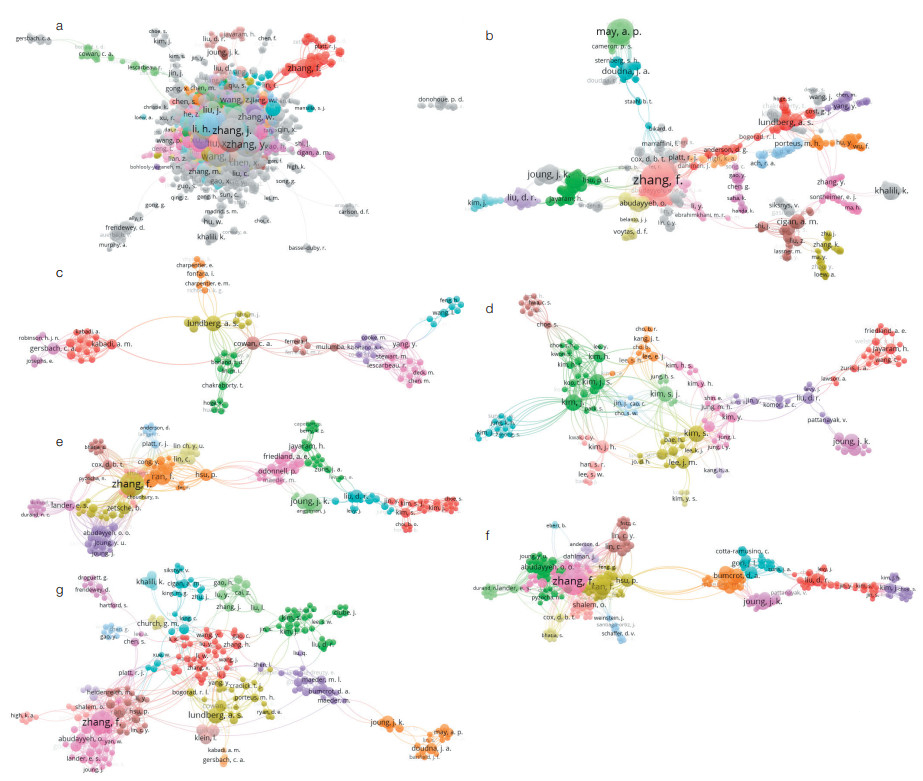
|
| 图 8 各国(地区)/组织中CRISPR技术相关专利的发明人合作关系 (a)中国;(b)美国;(c)日本;(d)韩国;(e)澳大利亚;(f)世界知识产权组织;(g)欧洲专利局 |
美国、世界知识产权组织、欧洲专利局、韩国、澳大利亚、日本的CRISPR技术相关专利的发明人呈现的状态较为相似:均由多个研发团体构成,且研发团体边界清晰,易分辨;每个研发团体存在1—2位核心人物,研发团体内部围绕核心人物又构建了多个相互关联的小研发团队(图 8b—g)。这些国家(地区)/组织的研发团体之间构成交叉网络合作关系,合作关联性强。世界知识产权组织和欧洲专利局符合全球性或区域性布局,拥有核心的强关联发明人,发明人合作关系呈现网状或集簇状,多个研发团体间构成复杂的合作网络。
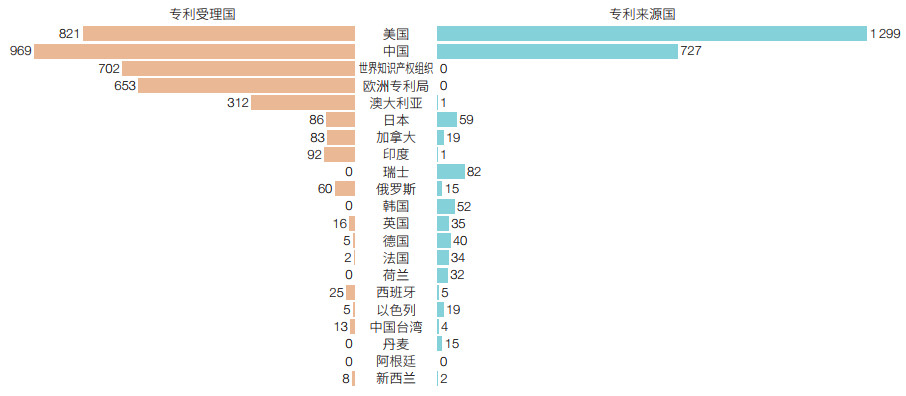
3.4 CRISPR相关专利技术流向分析从专利受理国的专利量和专利来源国的专利量2个维度④进行分析,了解各国技术的输出和输入情况(图 9)。
④ 专利受理国的专利量即为全球技术在该国的技术输入量;专利来源国的专利量即为该国技术向全球的技术输出量。
|
| 图 9 全球各国(地区)/组织CRISPR技术相关专利技术输出量和技术输入量分析 分析中合并了同族专利,图中数值均为专利族量;单位:项 |
美国是最大的专利来源国,在其他国家的专利布局量大,整体呈现技术对外输出;中国是最大的专利受理国,其他国家在中国的专利布局量较大,中国是全球技术的输入国。中国受理的专利主要来自国内的申请人,中国的大部分技术并未“走出去”,集中在本土化保护,尚未形成有效的国际布局,未申请专利的区域,一旦出现侵权行为,则无法维权。
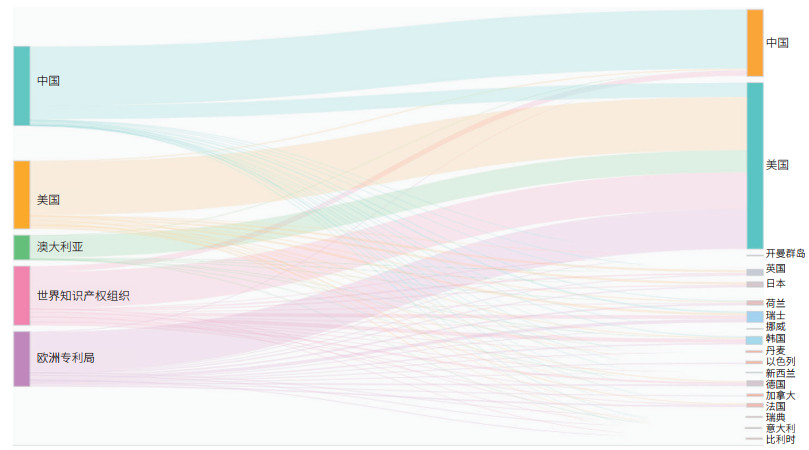
CRISPR技术相关专利的全球布局如图 10:美国近12%的技术申请世界知识产权组织或欧洲专利局,进而达到多国保护目的;相反,中国近90%的技术均为本土化申请,少于10%的技术申请世界知识产权合作组织或欧洲专利局的专利,极少的技术申请了美国专利或澳大利亚专利。
|
| 图 10 各国(地区)/组织CRISPR技术相关专利布局 左侧为受理国(地区)/组织,右侧为专利来源国(地区)/组织;每一个国家(地区)/组织对应一个条形框,条形框越长,数据量越大,占总量的比例也越大 |
CRISPR相关专利技术应用领域主要集中于医学、农业和工业领域;其中,医学是最大的应用领域,其专利产出量约为工业和农业领域专利数量的总和。美国的CRISPR相关专利近1/2应用于医学领域,在工业领域和农业上各占有1/4。在中国,CRISPR相关专利技术应用领域主要为医学,应用专利高达中国相关专利总量的2/3,农业专利占比略大于1/6,工业领域专业占比不足1/6。
(1)CRISPR主要研究方向分析。通过分析可知,CRISPR主要的技术方向包括:①乳腺癌、肺癌治疗;②抗原受体T细胞表达,干细胞诱导;③蛋白载体,PCR(聚合酶链式反应),敲除结构[15];④双链DNA的选择;⑤植物位点,核酸内切酶,多核苷酸修饰;⑥互补修饰合成杂交核苷酸;⑦染色体序列表达;⑧修饰靶向核酸;⑨核苷酸序列;⑩靶向DNA的RNA定向转录;⑪离体基因特异性。
(2)CRISPR相关专利技术点汇总。对4 041项CRISPR专利按技术点粗标引,用CRISPR的技术步骤演进图呈现相关技术点(图 11)。准确度(如脱靶、识别度低等)和适应性(如毒性、特异性和耐药性等)成为靶向基因治疗研究的关键问题[16, 17]。为此,研究者在探索不同的酶、不同的蛋白序列和精确的修复方式等,以及寻找新的替代方式[18-20]。同时,开始研发体细胞基因组编辑,直接治疗遗传疾病[21]。

|
| 图 11 CRISPR技术方向演进图 |
(3)CRISPR“行业大牛”张锋的研究主题分析。为了解CRISPR相关专利最多的发明人——美国麻省理工学院张锋的研究方向及专利保护情况,特对张锋的专利进行了初步标引,其标引点集中于脱靶、识别、修复和序列表达等。笔者团队还对标引的专利进行了数量的统计分析(图 12)。针对CRISPR技术在脱靶、识别、运输、分选、修复、减少毒性及序列表达等方向均存在非常多的技术薄弱点,且技术之间具备较强的相关性,张锋团队专利技术并未集中于某一些点,且单个专利技术通常涉及多个技术点并跨越多个方向。
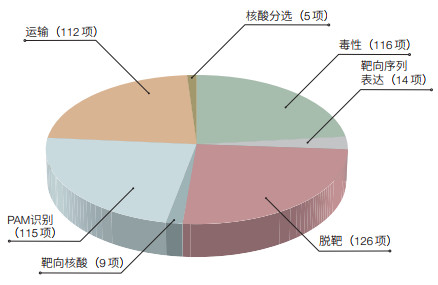
|
| 图 12 张锋的CRISPR相关专利的研究主题 |
随着研究的深入,基因编辑在基因研究、基因治疗和遗传改良等方面展示出了巨大的潜力。整体来看,北美地区占据了基因编辑市场的主导地位,美国把基因编辑在疾病治疗中的应用视作地区市场增长的主要动力。欧洲和韩国在基因编辑领域的研究也较为活跃。
4.1 基因编辑专利纷争美国麻省理工学院张锋也是美国博德研究所的重要骨干力量。多年来,张锋团队与美国加州大学伯克利分校杜德纳(Doudna J A)团队之间一直有着CRISPR相关专利争夺:杜德纳团队申请了第一个CRISPR相关专利,张锋团队则通过快速通道先一步得到CRISPR在真核领域的应用专利。随着加州大学伯克利分校的相关专利在2019年4月被授权,双方的专利纷争将会持续。双方对于CRISPR/Cas9系统开发商品化产品的权利纷争,成为CRISPR技术产业化过程中的主要竞争点。
4.2 重要研发机构专利布局(1)美国博德研究所专利布局分析。该研究所基因编辑相关专利171件(图 13a),其中明确用于疾病治疗65项。美国博德研究所早在2010年就开始布局基因编辑的相关专利。布局重点在于Cas酶,包括Cas13酶、Cas9酶、Cpf1酶和Cas酶的通用技术,其中:Cas9酶是其长期的布局重点,Cpf1酶则是近年的布局重点。此外,CRISPR系统是博德研究所最重视的基因编辑技术主题,总共布局了59项专利。gRNA技术也是其布局重点,共布局了23项专利申请。此外,其在前间隔序列邻近基序(PAM)、核酸检测系统、基因测序技术、计算机算法、载体、ZFN、TALEN和CRISPR的技术方面也布局了一定数量的专利。值得关注的是,2017—2018年,其布局了较多数量的其他技术主题的专利,这可能意味着其专利布局转向了新的研究热点。综合来看,美国博德研究所的布局重点在于CRISPR及其相关技术,在CRISPR技术方面,布局较为全面,涉及了CRISPR系统、Cas酶、gRNA、PAM等。
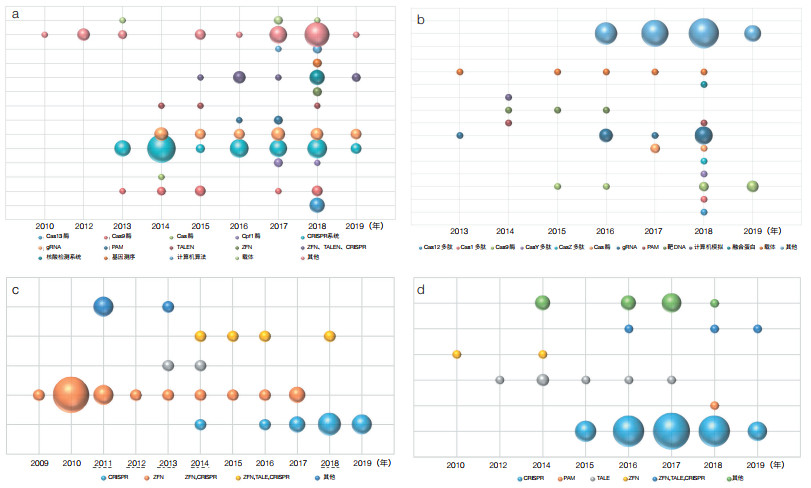
|
| 图 13 重要研发机构基因编辑专利布局趋势 (a)美国博德研究所;(b)美国加州大学;(c)德国默克公司;(d)韩国生物技术公司;图中节点大小表示申请专利数量的多少,节点颜色表示不同的技术领域 |
(2)美国加州大学专利布局分析。该大学基因编辑相关专利159件(图 13b)。加州大学自2008年开始布局基因编辑相关专利,在ZFN、TALE和CRISPR技术方面有较多专利申请;自2013年以来,持续在CRISPR技术领域进行布局,共申请92项。在专利用途方面,64项用于生命健康领域,7项用于农作物领域。加州大学在Cas酶方面专利布局较多,尤其是自2018年以来,在各种Cas酶方面布局较多,其中布局量最大的是Cas9酶。此外,在gRNA和靶DNA方面专利申请量也较多。
(3)德国默克公司专利布局分析。该公司于2015年收购了Sigma-Aldrich公司,使其成为默克公司的子公司。经检索,共检索到德国默克公司基因编辑相关专利42件(图 13c)。德国默克公司前期主要在ZFN技术方面进行专利布局,从2014年开始专利布局重点转向CRISPR领域;截至目前,其拥有11项CRIPSR技术相关专利,布局重点技术在于Cas酶、gRNA、CRISPR、ZFN和TALEN等,这显示近年来欧洲大型企业也开始注重CRISPR技术的研发,并积极参与专利布局。
(4)韩国生物技术公司专利布局分析。由图 13d可以看出,早在2010年韩国生物技术公司开始在ZFN技术领域进行专利布局,2015年开始重视在CRISPR领域的专利布局。2015—2019年,韩国共布局CRISPR技术相关专利55项,是近年来在CRISPR技术领域的专利申请大户,其CRISPR技术的布局重点在于Cas酶和gRNA。
5 对策与建议通过上述主要机构发文情况、全球专利概况、各国专利发明人的合作关系、技术流向分析、技术构成分析、主要创新主体现状等方面的数据和分析,提出4条对策与建议。
5.1 基因编辑是关乎生命科学未来发展的重要研究领域,应注重原始性技术的创新利用基因编辑技术,可以实现对基因组DNA的精准修饰,从而加强基因功能的解析、了解生长发育机制、揭示生命的本质等。①在农业生产中,基因组编辑技术可以用来改良动植物品种。基因组编辑品种仅针对靶基因序列进行精准的改造,不再受制于外源基因随机整合带来的生物安全和伦理风险的制约,可为人类提供高产、优质、安全的食品。②在疾病防治和精准医疗方面,基因组编辑不仅可以建立各种动物模型了解致病机制,还可用于疾病治疗,解决艾滋病、血液病、癌症、神经性退行性疾病等其他多种遗传性疾病的基因治疗难题。
我国科学家在基因组编辑领域取得了一系列具有原创性的突破,仍存在着一些问题:①在技术方面仍需要不断升级和完善;②在基础研究方面,应加强DNA修复机制、精准基因定点替换与插入的研究,支持各种模式生物(特别是灵长类动物)的研究。全面加速CRISPR基因组编辑技术的成熟和广泛应用,利用与表观遗传学、细胞图像学、合成生物学等多学科交叉来研发新的基因组编辑衍生技术。在改进现有技术的同时,重视对基因组编辑原始创新的投入,加速向源头创新转移,开发新工具、新技术,解决原创性技术匮乏、研发重点不够突出、国家专项经费支持缺乏、产业化方向不明确、相关的法律法规制订不完善等问题。
5.2 基因编辑技术的发展日新月异,必须加快我国基因编辑产品的产业化进程基因编辑新技术带来的最大的冲击莫过于其不可估量的应用价值。美国近1/2基因编辑相关专利应用于医学领域,其余专利应用在工业领域和农业领域。中国的基因编辑研究主要集中在医学领域,约有2/3相关专利用于医学,农业领域相关专利占比略大于1/6,工业领域专利量最少,占比不足1/6。全球其他国家也均将基因编辑研究重点落在医学领域,较少的专利关注于农业和工业领域。建议中国进一步关注基因编辑技术在农业和工业领域的应用,以抢占这2个领域的领先地位。
建议现阶段:①可重点推进农作物基因组编辑育种的生产应用,从非粮食作物、工业原料作物做起,逐渐推进到主要粮食作物,带动农业生产的绿色转型;②加大临床疾病防治、精准医学新模式及药物治疗的应用步伐,谨慎推进人类胚胎等有争议方向的研究,稳步推进有中国特色的基因组编辑产业化之路;③选择自主研发为主,联合研发、专利运营与并购手段为辅的策略,从提高基因编辑技术的便利性、适用性和安全性出发,充分利用基因编辑技术进行二次创新,培育高价值的专利,进而活跃市场的比较竞争优势。
5.3 全球竞争异常激烈,研发中要特别注重高水平的国际合作在注重原创性技术的创新,以及研发具有自主知识产权的基因组编辑新技术、新方法、新产品的基础上,应该加强互补性合作,把专利的渠道拓宽,把技能本身往前推——系统化、规模化,在速度和成本方面做一个改变,这可能对我们来说更具有价值。
合作关系展示了高水平科研合作的重要性和有效性。哈佛大学目前是基因编辑研究的核心机构,跟全球很多的高校、研究所或企业有相关合作研究。中国科学院是中国基因编辑研究的主导力量之一,建议研发中要特别注重合作;除了同国际领先的实验室合作外,更应该注重国内优势单位的强强联手;同时,要加强产学研结合,通过研究机构、高校与企业的合作,加速研究成果的转移转化。
5.4 从国家层面来说,应进一步增加专利全球布局的支持力度技术竞争的实现在很大程度上已经演变为知识产权之争,知识产权制度正是发挥市场机制决定性作用,鼓励创新、激发创造、促进运用的关键性制度保障。①在专利布局方面,应合理利用优先权、分案申请及全球化布局。例如,使用《专利合作条约》(PCT)和《欧洲专利公约》的方式进入世界各国,抢占更多技术市场,为后续技术竞争做好准备。②重点关注国外主要申请人的专利布局情况,并结合自身技术规划,构建我国应对竞争对手的围堵式专利布局,以有效回避未来的侵权风险。③定期搜集并整理外来专利的申请策略、保护范围和技术方向等,及时准确地掌握外来专利的知识产权战略;充分利用《中华人民共和国专利法》中规定的授权条款和无效条款,对外来专利进行防范,主动进行专利风险处置。④专利申请过程中的申请方式和公开方式,需要结合技术成果的先进性和市场竞争力来选择,合理的方式方法有助于推动知识产权的保护。
全球的专利布局将有利于在全球构建有规模、有效力的专利保护范围,从而占据全球的竞争优势。通过世界知识产权组织和欧洲专利局开展全球专利布局则是较受关注的申请方式。对较为优秀的技术成果,应加大全球的布局,国家应增加全球布局的支持力度,通过政策、资金等方式对我国的专利申请人提供帮助和支持,提高我国在全球的竞争优势和话语权地位。同时,应提高专利保护意识,密切跟踪重要申请人和重要发明人的中国专利进度,寻找其未来可能面临的困难,善用司法制度积极维护自身合法权利;还要注重运用世界贸易组织(WTO)中的争端解决机制及4种救济途径,以应对贸易摩擦引发的争端。
| [1] |
Gaj T, Gersbach C A, Barbas C F III. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology, 2013, 31(7): 397-405. DOI:10.1016/j.tibtech.2013.04.004 |
| [2] |
Gupta R M, Musunuru K. Expanding the genetic editing tool kit:ZFNs, TALENs, and CRISPR-Cas9. Journal of Clinical Investigation, 2014, 124(10): 4154-4161. DOI:10.1172/JCI72992 |
| [3] |
LaFountaine J S, Fathe K, Smyth H D C. Delivery and therapeutic applications of gene editing technologies ZFNs, TALENs, and CRISPR/Cas9. International Journal of Pharmaceutics, 2015, 494(1): 180-194. DOI:10.1016/j.ijpharm.2015.08.029 |
| [4] |
Liu H, Liu C, Zhao Y H, et al. Comparing successful gene knock-in efficiencies of CRISPR/Cas9 with ZFNs and TALENs gene editing systems in bovine and dairy goat fetal fibroblasts. Journal of Integrative Agriculture, 2018, 17(2): 406-414. DOI:10.1016/S2095-3119(17)61748-9 |
| [5] |
Shan Q W, Baltes N J, Atkins P, et al. ZFN, TALEN and CRISPR-Cas9 mediated homology directed gene insertion in Arabidopsis:A disconnect between somatic and germinal cells. Journal of Genetics and Genomics, 2018, 45(12): 681-684. DOI:10.1016/j.jgg.2018.07.011 |
| [6] |
Jaworski A, Dobrowolska A. Organization of the bacterial interference RNAi-like system (CRISPR-CAS) and its role against bacteriophages. Postepy Mikrobiologii, 2009, 48(1): 23-30. |
| [7] |
Carte J, Pfister N T, Compton M M, et al. Binding and cleavage of CRISPR RNA by Cas6. RNA, 2010, 16(11): 2181-2188. DOI:10.1261/rna.2230110 |
| [8] |
Babu M H, Beloglazova N, Flick R, et al. A dual function of the CRISPR-Cas system in bacterial antivirus immunity and DNA repair. Molecular Microbiology, 2011, 79(2): 484-502. DOI:10.1111/j.1365-2958.2010.07465.x |
| [9] |
Makarova K S, Haft D H, Barrangou R, et al. Evolution and classification of the CRISPR-Cas systems. Nature Reviews Microbiology, 2011, 9(6): 467-477. DOI:10.1038/nrmicro2577 |
| [10] |
Burley K M, Sedgley C M. CRISPR-Cas, a prokaryotic adaptive immune system, in endodontic, oral, and multidrugresistant hospital-acquired Enterococcus faecalis. Journal of Endodontics, 2012, 38(11): 1511-1515. DOI:10.1016/j.joen.2012.07.004 |
| [11] |
Weinberger A D, Gilmore M S. CRISPR-Cas:To take up DNA or not-That is the question. Cell Host & Microbe, 2012, 12(2): 125-126. |
| [12] |
Capecchi M R. Culturing cells having a modification of a target DNA sequence in its genome, useful for producing transgenic animals or for manipulating plant cells, comprises transforming cells with a positive-negative selector vector: US, 781559.1997-01-09.
|
| [13] |
Routman K D. Universities and industry square off over recombinant-DNA ownership rights and research talent. Drug & Cosmetic Industry, 1981, 128(3): 40. |
| [14] |
Blum B, Bakalara N, Simpson L. A model for RNA editing in kinetoplastid mitochondria:"Guide" RNA molecules transcribed from maxicircle DNA provide the edited information. Cell, 1990, 60(2): 189-198. DOI:10.1016/0092-8674(90)90735-W |
| [15] |
Zhang B B, Xia Q, Wang Q, et al. Detecting and typing target DNA with a novel CRISPR-typing PCR (ctPCR) technique. Analytical Biochemistry, 2018, 561/562: 37-46. DOI:10.1016/j.ab.2018.09.012 |
| [16] |
Alkan F, Wenzel A, Anthon C, et al. CRISPR-Cas9 offtargeting assessment with nucleic acid duplex energy parameters. Genome Biology, 2018, 19(1): 177-189. DOI:10.1186/s13059-018-1534-x |
| [17] |
Lee S H, Kim S, Hur J K. CRISPR and target-specific DNA endonucleases for efficient DNA knock-in in Eukaryotic genomes. Molecules and Cells, 2018, 41(11): 943-952. |
| [18] |
Bewg W P, Ci D, Tsai C J. Genome editing in trees:From multiple repair pathways to long-term stability. Frontiers in Plant Science, 2018, 9: 1732. DOI:10.3389/fpls.2018.01732 |
| [19] |
Otsuka K, Tomita M. Concurrent live imaging of DNA doublestrand break repair and cell-cycle progression by CRISPR/Cas9-mediated knock-in of a tricistronic vector. Scientific Reports, 2018, 8(1): 17309. DOI:10.1038/s41598-018-35642-7 |
| [20] |
Slyskova J, Sabatella M, Ribeiro-Silva C, et al. Base and nucleotide excision repair facilitate resolution of platinum drugs-induced transcription blockage. Nucleic Acids Research, 2018, 46(18): 9537-9549. DOI:10.1093/nar/gky764 |
| [21] |
Zhu W, Saw D, Weiss M, et al. Induction of brain arteriovenous malformation through CRISPR/Cas9-mediated somatic Alk1 gene mutations in adult mice. Translational Stroke Research, 2019, 10(5): 557-565. DOI:10.1007/s12975-018-0676-1 |