2. 中国科学院青藏高原研究所 北京 100101
2. Institute of Tibetan Plateau Research, Chinese Academy of Sciences, Beijing 100101, China
微生物是地球生物圈的重要组成部分。作为肉眼看不见的大多数,微生物受生态环境和人类活动的影响,同时也反作用于生态环境和人类健康。多样化的微生物参与生态系统物质循环,维持人体、动物、植物的健康稳态,并在农业、工业等领域发挥着重要作用。从山川湖海到空气土壤,从宏观世界到微观世界,微生物无处不在。在地表和地球深部极端环境中,微生物是少数甚至唯一能够存活的生命形式。其中,冰冻圈微生物是极端环境微生物研究中极具代表性的研究对象。
冰冻圈(cryosphere)是指地球表层连续分布且具有一定厚度的负温圈层,主要分布在地球两极和高山地区,组成要素包括冰川、冰山、积雪、河冰、湖冰、海冰和冻土等[1]。冰冻圈环境寒冷、缺氧、黑暗,人类活动微弱,这种独特而极端的环境蕴藏着丰富的微生物资源,部分微生物可以存活数百万年[2]。证据表明,冰冻圈事实上构成了微生物的巨大存储池,保存着古微生物多样性和进化历史的珍贵记录[3]。同时,冰冻圈与其下游生态环境、人类生产生活具有直接或间接的联系。随着生态环境的变迁和人类活动的拓展,冰冻圈微生物自身及其与人类世界的关系处于不断演变中。
气候变化对微生物的影响及微生物的反馈机制近年来逐渐受到关注。尤其值得注意的是,病原微生物对人类的不利影响可能在加剧:媒介传播传染病、海洋生物感染、农作物感染、抗生素耐受微生物的风险增加均与气候变化及人类活动存在相关性[4]。全球变暖背景下,冰冻圈正在持续且加速融化、缩减,冻存其中的微生物被“解封”,一些微生物适应新的环境条件从而复苏存活[5],而它们对当代生物圈的影响值得评估。本文主要从冰冻圈微生物多样性和生物安全的角度,剖析气候变化条件下休眠微生物释放、复苏对生态环境和人类健康的影响,并提出相应的风险防控建议。
1 冰冻圈微生物多样性根据以积雪、海冰、冰川、冻土等为对象的调查研究估计,冰冻圈中细胞密度范围为10—108 cells·mL−1,细胞总数可达1025—1028个[6]。随着测序技术和生物信息学的发展及调查范围的扩大,冰冻圈蕴藏的复杂微生物群落正在被更为系统、完整地记录和揭示。
冰冻圈是受气候变化影响显著的地区,微生物作为生态环境中碳、氮和各种营养物质降解与循环的参与者,对全球生物多样性的动态变化至关重要。气候变化引起冰川、冻土等的融化改变了冰冻圈生态环境,对当地动、植物等生物的组成、数量、分布、生理状态等造成影响,直接或间接地改变着栖息其中的微生物群落的数量、组成和生态功能。同时,微生物群落的变化也会对生态环境变化产生反馈机制,从而对生态环境的演化施加进一步影响。在此过程中,随着冰川、冻土等融化而释放的微生物离开原有栖息环境,其中复苏微生物的增殖和代谢活动,可对周边乃至下游生态环境的微生物多样性[7]及其生态功能造成深远影响。
1.1 细菌、真菌和古菌针对北极和高山地区等典型冰冻圈环境的调查研究证实了冰冻圈中的微生物呈现多样性。据估计,冰川环境中微生物的生物量约为102—104 cells · mL−1 [8]。例如,北极斯瓦尔巴(Svalbard)群岛冰川底部冰和冰川融水中检测到了丰富的细菌和真菌群落[9],细菌中放线菌门和变形菌门占主导,包括Flavobacterium、Cryobacterium、Hymenobacter等13个属;真菌中担子菌占比最高,分离得到分属19个属的30种丝状真菌和10个属的11种酵母,微生物组成依不同采样位置而呈现显著差异。喀喇昆仑山脉巴托拉(Batura)冰川冰中分离得到了Penicillium、Cladosporium、Geomyces等14个属的真菌[10]。对钻取自青藏高原西北古里雅冰帽的50 m冰芯进行分析[11],得到254个细菌属,其中118种可归入已知细菌分类,主要包括Janthinobacterium、Polaromonas、Flavobacterium等32种细菌。微生物组成依据冰层深度而呈现差异,指示了不同时期的环境变化和条件差异。
冻土也是微生物的重要栖息地,保存着不同年代的古微生物种群。冻土中的细胞密度可达105—108 cells · mL−1[6]。例如,瑞典北部斯托达林(Stordalen)沼泽的非连续多年冻土被认为是一处模式北极泥炭地生态系统;利用宏基因组测序从该沼泽的大量冻土样本中分析获得了1 529个基因组,其中细菌基因组1 434个,古菌基因组95个,揭示了该冻土中存在跨越30个门的微生物类群[12]。
1.2 病毒研究者已在全世界多个冰冻圈环境中发现了丰富的病毒资源。例如,南极林诺博拉尔(Limnopolar)湖中检出大量DNA病毒和RNA病毒[13, 14],大多具有不同于已知病毒的基因组特点。南极土壤中发现了大量噬菌体、藻类病毒和感染变形虫的巨型病毒;病毒分布依采样点呈现显著不同,共涉及38个科,感染宿主涉及细菌、古菌、单细胞真核生物、昆虫、脊椎动物和植物[15]。瑞典北部斯托达林沼泽冻土中发现了大量DNA病毒,分析获得1 907个病毒基因组,有1 106个在宏转录组中检出,提示许多病毒处于活跃状态,且其中仅有17% 的病毒可在现有分类学中归类[16]。在美国阿拉斯加乌特恰维克镇(Alaska Utqiagvik)采集的冻土样本中病毒密度为108 mL−1,附近海冰样本中病毒密度为105 mL−1,分析得到476个病毒种群,其中仅有12% 可归入已知病毒种类[17]。高海拔地区冰冻圈环境同样发现了丰富的病毒种类。我国青藏高原古里雅冰芯不同深度的冰芯样本(约520—15 000年冰龄)中发现了33种病毒,其中4种可归入已知病毒种类,其余29种为新病毒种类[11]。大量新病毒类型的发现表明了冰冻圈在生态资源多样性方面的独特价值。
病毒通过“病毒-宿主”感染关系参与微生物群落结构的调控和元素的生物化学循环。一些病毒具有非同寻常的策略和基因来控制其复制及维持与宿主的长期关系。例如,有溶原性噬菌体的基因以卫星噬菌体质粒的形式存在于细菌当中;有噬菌体编码一种CRISPR/Cas自适应细菌免疫系统,在提升自身竞争力的同时赋予其宿主免于其他噬菌体感染的免疫力[18]。冰冻圈极端的环境条件维持着简单的食物链,丰富多样的病毒通过裂解宿主细胞、介导基因传递、参与宿主细胞代谢等方式调控微生物群落的数量规模,驱动微生物进化,并且影响着生态系统中碳和营养物质的释放和再循环。
2 冰冻圈微生物的生物安全风险在冰冻条件下,微生物处于休眠状态或低代谢水平,该生理状态抑制基因变异而有利于基因修复,支持着微生物的稳定保存。同时,古微生物凋亡所分解释放的生物分子(如基因残余物),也因降解酶活性受到抑制而得以保存。冰冻圈内冰川、冻土的融化或人类在冰冻圈活动的拓展,导致其中封存的微生物或基因残余物离开原有栖息环境,直接或间接地与周围乃至下游自然环境产生关联。有研究估计每年约有1017—1021个微生物细胞从冰冻环境中释放[19],且证实休眠的微生物在百万年后仍可复苏[20]。当复苏的古微生物进入当今地球生物圈,其中的病原体和抗生素抗性基因将对人类健康构成潜在威胁。
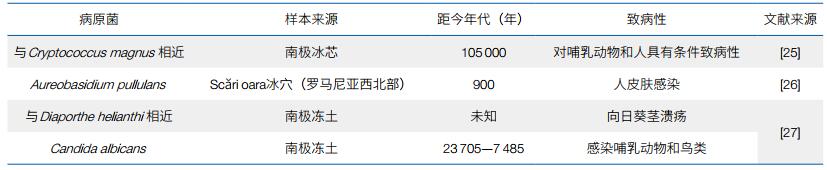
2.1 致病菌研究者已在世界多处冰冻圈环境中分离得到病原微生物。例如,可感染免疫缺陷人群的条件致病菌Cryptococcus[21],具有多种极端环境适应性的条件致病菌黑酵母菌(Aureobasidium pullulans)[22, 23],以及植物病原菌Pleosporales、Helotiales等[24]。研究者还从冰芯、冻土等环境中发现了距今百年甚至上万年的病原微生物(表 1),包括短梗霉菌、隐球菌、白念珠菌等,这些古老的真菌以其特有的规避机制在极端寒冷和营养缺乏的环境下保存生命力。
病原微生物可通过复苏感染、基因传播等方式导致生物安全风险,对人类、动物、植物造成影响。 2016年俄罗斯出现炭疽疫情,调查者推断病原体很可能源于埋藏于冻土中70余年的受感染驯鹿尸体——冻土融化导致炭疽杆菌释放,通过驯鹿、牛等牲畜感染继而传播至人类[3]。埋藏于冰冻环境的致病菌可随冰雪融化而扩散,经由水、动物等媒介传播至人类[28],从而对人类健康构成致病风险。古老病原体中的致病基因还可通过水平基因转移进入当代微生物圈。
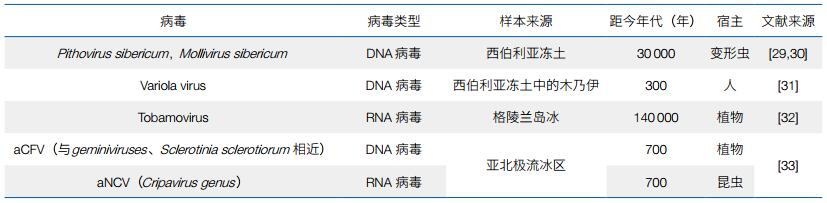
2.2 病毒冰冻圈储存着年代久远的病毒,冰冻条件使大多数古病毒都保持了完整性和多样性,对人类具有极大的生物安全风险。研究者从西伯利亚冻土、亚北极地区流冰、格陵兰岛冰等冰冻圈环境中提取到了距今几百年乃至上万年的DNA和RNA病毒(表 2)。一些古病毒的基因组结构和复制循环与可感染人类和动物的病毒具有相似性[29]。古病毒的发现及复苏病毒所展示出的感染宿主的能力[29, 30]预示着冰冻圈缩减所导致的极大生物安全风险。全球冰冻圈的加速消融缩减和人类在极寒地带不断增强的工业开采等活动可“解封”冻存的微生物资源,从而开启“潘多拉之盒”,“激活”过去全球传染病的病原体。由于现代生物的免疫系统对古病毒并不熟悉,古病原体可能对现代生物构成更大的威胁,从而引发广泛的传染病流行。
冰冻圈环境中病毒的演化及其与流行病发生的关系尚待深入研究,然而研究者已经发现了一些潜在相关的病毒感染机制。在西伯利亚湖冰中检测到了流感病毒[34],表明这种鸟类传播的RNA病毒可以在高纬度冰或水环境中储存。冰雪冻融储存/释放病毒和鸟类迁徙引入/感染病毒共同促进了冰冻圈病毒基因的动态变化和流动传播。在亚北极地区流冰区冻存的驯鹿粪便中检测到2种病毒[33],病毒通过驯鹿摄食植物或昆虫传播进入胃肠道,病毒壳体保护其在肠道中完成复制,直至通过粪便排出,病毒仍具有传染性。冰冻条件下,病毒壳体保护病毒在数百年间不被降解。这些研究揭示了古病毒长期保存的稳定性,以及病毒通过动物肠道等媒介保存、扩散的传播机制。深入解析冰冻圈病毒跨越原有时空环境进入当代生物圈后的演化机理,及其与宿主的互作关系,将有助于进一步评价古病毒的生物安全风险。
2.3 抗生素抗性基因抗生素抗性基因是冰冻圈生物安全风险的重大威胁之一。抗生素抗性已经构成当今世界公共卫生事业的一大威胁,特别是多种抗生素耐受细菌严重挑战公共健康。抗生素抗性过去普遍归因于抗生素滥用。然而,研究者已在冰冻圈环境中发现了大量携带抗生素抗性基因的细菌和抗性基因组,这些抗性基因共涉及50余种抗生素,地理分布跨越地球多数冰冻圈环境[5]。许多抗性基因与当代致病细菌中的相关基因具有可比性,这表明抗生素抗性具有古老且世界性的起源,早于人类使用抗生素[35, 36]。
冰冻圈中的抗性基因可能随着雪冰的融化而释放,继而随细菌增殖扩散,通过水平基因转移被冰冻圈以外的微生物所获得。有研究表明历史上的冰冻圈融化很可能曾经导致显著的水平基因转移[37],已经对微生物进化造成了影响。冰冻圈微生物构成了一个抗生素抗性基因的存储池,可通过基因转移影响抗生素耐药菌的进化过程[38]。例如,复苏的携带抗生素抗性基因的非致病菌,可能从环境中获得致病基因,或将抗生素抗性基因传递给其他微生物。当冰冻圈微生物不适应新环境而分解,其所释放的基因残余物中的抗生素抗性基因,也可能传递给周围环境中的微生物。迁徙鸟类等动物、空气传播细菌均可在抗性基因的扩散传播中充当媒介[39]。
3 对策建议 3.1 建立冰冻圈微生物战略资源库冰冻圈微生物的多样性使其构成了一个微生物资源的存储池,其中保存着大量新颖的原核、真核微生物和病毒类型。一些微生物具有特殊的生物功能,如极端条件适应性、抑菌性、生物催化或合成能力等,这些基因资源未来可用于生物技术研发及生物化工、生物制药等领域。建议在充分调查和掌握冰冻圈微生物多样性的基础上,开展冰冻圈微生物资源建库工作。同时,鉴于冰冻圈古微生物存在潜在的病原菌和抗生素耐受菌,这与生物安全防控息息相关,是研究传染病传播、抗生素抗性基因及其进化机制的珍贵材料,建议建立专门的微生物样本实体库和基因组数据库,并将其纳入国家生物安全战略资源。
3.2 开展冰冻圈病毒学普查,建立监测和预警机制冰川冻土等冰冻圈要素保存了不同时期的病毒样本,通过宏基因组学和病毒分离鉴定等手段,可以分析冰冻圈病毒组成和多样性的时空变化,反映极端环境中和受气候变化影响下病毒圈的演变,揭示历史不同时期病毒的特点及其与传染病流行的时空关联。
中国作为全球中低纬度地区冰冻圈最发育的国家,冰川、冻土和积雪分布广、数量大。其中,青藏高原以其深居内陆、低纬度、高海拔的独特地理特点而被称为“第三极” [40],具有十分重要的生态学价值。有必要聚焦青藏高原等代表性冰冻圈中的微生物多样性,开展病毒学普查工作。针对特殊生境,包括冰川和冻土及其中埋藏的动植物干尸,以及其他可能保存古老病毒的环境,进行广泛的病毒学普查、病毒多样性和形态学分析,提前了解可能会释放到环境里的病毒种类,回溯分析古病毒与传染病流行的关系。在此基础上,建立冰冻圈消融释放病毒种类、通量及对生态环境影响的长期监测网络,综合长期监测数据和模型模拟,设置预警机制。
3.3 多学科视角开展冰冻圈病原微生物研究冰冻圈微生物中含有的致病菌、病毒和抗生素抗性基因提示了潜在的生物安全风险,这些风险因素与生态环境、人类健康的关系有待深入解析。为充分评估冰冻圈微生物的生物安全风险,需结合基因组分析、病原菌致病机理研究、病毒致病机理研究、病毒与宿主的互作分析等一系列手段,对特殊生境里的古老微生物进行前瞻性研究。同时,人类健康和疾病防控是一项涉及多学科、跨时空尺度的研究课题,需要将古微生物致病机理研究、病原体及其传播机制等研究纳入多学科研究框架,充分吸纳病原学、流行病学、生态学、社会学、人类学等学科知识和大数据分析方法,从更为全面的视角探讨病原菌、病毒等微生物的致病机理、扩散传播机制,以及潜在的生物安全风险。
| [1] |
秦大河, 姚檀栋, 丁永建, 等. 冰冻圈科学体系的建立及意义. 中国科学院院刊, 2020, 35(4): 394-406. |
| [2] |
Charlier P, Claverie J M, Sansonetti P, et al. Re-emerging infectious diseases from the past: Hysteria or real risk?. European Journal of Internal Medicine, 2017, 44: 28-30. DOI:10.1016/j.ejim.2017.06.018 |
| [3] |
Houwenhuyse S, Macke E, Reyserhove L, et al. Back to the future in a petri dish: Origin and impact of resurrected microbes in natural populations. Evolutionary Applications, 2018, 11(1): 29-41. DOI:10.1111/eva.12538 |
| [4] |
Cavicchioli R, Ripple W, Timmis K, et al. Scientists' warning to humanity: Microorganisms and climate change. Nature Reviews Microbiology, 2019, 17(9): 569-586. DOI:10.1038/s41579-019-0222-5 |
| [5] |
Sajjad W, Rafiq M, Din G, et al. Resurrection of inactive microbes and resistome present in the natural frozen world: Reality or myth?. Science of the Total Environment, 2020, 735: 139275. DOI:10.1016/j.scitotenv.2020.139275 |
| [6] |
Boetius A, Aneesio A M, Deming J W, et al. Microbial ecology of the cryosphere: Sea ice and glacial habitats. Nature Reviews Microbiology, 2015, 13(11): 677-690. DOI:10.1038/nrmicro3522 |
| [7] |
Cameron K A, Müller O, Stibal M, et al. Glacial microbiota are hydrologically connected and temporally variable. Environmental Microbiology, 2020, 22(8): 3172-3187. DOI:10.1111/1462-2920.15059 |
| [8] |
Miteva V. Psychrophiles: From Biodiversity to Biotechnology. Berlin, Heidelberg: Springer, 2008, 31-50. |
| [9] |
Perini L, Gostinčar C, Gunde-Cimerman N. Fungal and bacterial diversity of Svalbard subglacial ice. Scientific Reports, 2019, 9(1): 20230. DOI:10.1038/s41598-019-56290-5 |
| [10] |
Hassan N, Hasan F, Nadeem S, et al. Community analysis and characterization of fungi from Batura glacier, Karakoram mountain range, Pakistan. Applied Ecology and Environmental Research, 2018, 16(5): 5323-5341. DOI:10.15666/aeer/1605_53235341 |
| [11] |
Zhong Z P, Solonenko N E, Li Y F, et al. Glacier ice archives fifteen-thousand-year-old viruses. bioRxiv, 2020. DOI:10.1101/2020.01.03.894675 |
| [12] |
Woodcroft B J, Singleton C M, Boyd J A, et al. Genomecentric view of carbon processing in thawing permafrost. Nature, 2018, 560: 49-54. DOI:10.1038/s41586-018-0338-1 |
| [13] |
Cavicchioli R, Erdmann S. The discovery of Antarctic RNA viruses: A new game changer. Molecular Ecology, 2015, 24(19): 4809-4811. DOI:10.1111/mec.13387 |
| [14] |
López-Bueno A, Rastrojo A, Peiró R, et al. Ecological connectivity shapes quasispecies structure of RNA viruses in an Antarctic lake. Molecular Ecology, 2015, 24(19): 4812-4825. DOI:10.1111/mec.13321 |
| [15] |
Adriaenssens E M, Kramer R, van Goethem M W, et al. Environmental drivers of viral community composition in Antarctic soils identified by viromics. Microbiome, 2017, 5(1): 83. DOI:10.1186/s40168-017-0301-7 |
| [16] |
Emerson J B, Roux S, Brum J R, et al. Host-linked soil viral ecology along a permafrost thaw gradient. Nature Microbiology, 2018, 3(8): 870-880. DOI:10.1038/s41564-018-0190-y |
| [17] |
Zhong Z P, Rapp J Z, Wainaina J M, et al. Viral ecogenomics of arctic cryopeg brine and sea ice. mSystems, 2020, 16, 5(3): e00246-20.
|
| [18] |
Bellas C M, Anesio A M, Barker G. Analysis of virus genomes from glacial environments reveals novel virus groups with unusual host interactions. Frontiers in Microbiology, 2015, 6: 656. |
| [19] |
Rogers S O, Starmer W T, Castello J D. Recycling of pathogenic microbes through survival in ice. Medical Hypotheses, 2004, 63(5): 773-777. DOI:10.1016/j.mehy.2004.04.004 |
| [20] |
Barras C. Zombie creatures that have been resurrected after millions of years can help us understand the very nature of life. New Scientist, 2017, 234: 34-37. |
| [21] |
Turchetti B, Selbmann L, Blanchette R, et al. Cryptococcus vaughanmartiniae sp. nov and Cryptococcus onoforri sp. nov: Two new species isolated from worldwide cold environments. Extremophiles, 2015, 19: 149-159. DOI:10.1007/s00792-014-0692-3 |
| [22] |
Babič M N, Zupančič J, Gunde-Cimerman N, et al. Ecology of the human opportunistic black yeast exophiala dermatitidis indicates preference for human-made habitats. Mycopathologia, 2018, 183(1): 201-212. DOI:10.1007/s11046-017-0134-8 |
| [23] |
Gostinčar G, Grube M, Gunde-Cimerman N. Evolution of fungal pathogens in domestic environments. Fungal Biology, 2011, 115(10): 1008-1018. DOI:10.1016/j.funbio.2011.03.004 |
| [24] |
Edwards A, Douglas B, Anesio A M, et al. A distinctive fungal community inhabiting cryoconite holes on glaciers in Svalbard. Fungal Ecology, 2013, 6(2): 168-176. DOI:10.1016/j.funeco.2012.11.001 |
| [25] |
Knowlton C, Veerapaneni R, D'Elia T, et al. Microbial analyses of ancient ice core sections from Greenland and Antarctica. Biology, 2013, 2(1): 206-232. DOI:10.3390/biology2010206 |
| [26] |
Brad T, Itcus C, Pascu M, et al. Fungi in perennial ice from Scări?oara Ice Cave (Romania). Scientific Reports, 2018, 8: 10096. DOI:10.1038/s41598-018-28401-1 |
| [27] |
Kochkina G, Ivanushkina N, Ozerskaya S, et al. Ancient fungi in Antarctic permafrost environments. FEMS Microbiology Ecology, 2012, 82(2): 501-509. DOI:10.1111/j.1574-6941.2012.01442.x |
| [28] |
Goodwin K, Loso M, Braun M. Glacial transport of human waste and survival of fecal bacteria on mt. mckinley's kahiltna glacier, Denali National Park, Alaska. Arctic, Antarctic, and Alpine Research, 2012, 44(4): 432-445.
|
| [29] |
Legendre M, Bartoli J, Shmakova L, et al. Thirty-thousandyear-old distant relative of giant icosahedral DNA viruses with a pandoravirus morphology. PNAS, 2014, 111(11): 4274-4279. DOI:10.1073/pnas.1320670111 |
| [30] |
Legendre M, Lartigue A, Bertaux L, et al. In-depth study of Mollivirus sibericum, a new 30, 000-y-old giant virus infecting Acanthamoeba. PNAS, 2015, 112(38): 5327-5335. DOI:10.1073/pnas.1510795112 |
| [31] |
Biagini P, Theves C, Balaresque P, et al. Variola virus in a 300-year-old Siberian mummy. The New England Journal of Medicine, 2012, 367: 2057-2059. DOI:10.1056/NEJMc1208124 |
| [32] |
Castello J D, Rogers S O, Starmer W T, et al. Detection of tomato mosaic Tobamovirus RNA in ancient glacial ice. Polar Biology, 1999, 22(3): 207-212. DOI:10.1007/s003000050411 |
| [33] |
Ng T F F, Chen L F, Zhou Y C, et al. Preservation of viral genomes in 700-year-old caribou feces from a subarctic ice patch. PNAS, 2014, 111(47): 16842-16847. DOI:10.1073/pnas.1410429111 |
| [34] |
Zhang G, Shoham D, Gilichinsky D, et al. Evidence of influenza a virus RNA in Siberian lake ice. Journal of Virology, 2006, 80(24): 12229-12235. DOI:10.1128/JVI.00986-06 |
| [35] |
Petrova M, Kurakov A, Shcherbatova N, et al. Genetic structure and biological properties of the first ancient multiresistance plasmid pKLH80 isolated from a permafrost bacterium. Microbiology, 2014, 160(10): 2253-2263. DOI:10.1099/mic.0.079335-0 |
| [36] |
Perron G G, Whyte L, Turnbaugh P J, et al. Functional characterization of bacteria isolated from ancient arctic soil exposes diverse resistance mechanisms to modern antibiotics. PLoS One, 2015, 10(3): e0069533. DOI:10.1371/journal.pone.0069533 |
| [37] |
Bidle K, Lee S, Marchant D, et al. Fossil genes and microbes in the oldest ice on Earth. PNAS, 2007, 104(33): 13455-13460. DOI:10.1073/pnas.0702196104 |
| [38] |
Bhullar K, Waglechner N, Pawlowski A, et al. Antibiotic resistance is prevalent in an isolated cave microbiome. PLoS One, 2012, 7(4): e34953. DOI:10.1371/journal.pone.0034953 |
| [39] |
Segawa T, Takeuchi N, Rivera A, et al. Distribution of antibiotic resistance genes in glacier environments. Environmental Microbiology Reports, 2013, 5(1): 127-134. DOI:10.1111/1758-2229.12011 |
| [40] |
姚檀栋, 陈发虎, 崔鹏, 等. 从青藏高原到第三极和泛第三极. 中国科学院院刊, 2017, 32(9): 924-931. |