2. 中国科学院深圳理工大学(筹)* 合成生物学院 深圳 518055
2. Faculty of Synthetic Biology, Shenzhen Institute of Advanced Technology, Chinese Academy of Sciences, Shenzhen 518055, China
生命科学是研究生命本质及运行规律的自然科学,且与人类健康息息相关。本文试图从宏观层面,概略生命科学发展历程,归纳当代生命科学发展特征和前沿热点方向,分析中国在世界生命科学格局中的地位,举例介绍近10年来的代表性成果,讨论未来发展中需要关注和解决的问题。
1 生命科学百年发展趋势概略:3个“50年”生命科学近150多年发展史中大约每半个世纪出现1次地标性突破(图 1),并形成3个发展阶段。19世纪中叶,孟德尔通过种植豌豆实验发现遗传学基本规律并提出遗传因子假说(1865年),以后,摩尔根通过果蝇研究将遗传因子定位在染色体(1910年),他们共同奠基了经典遗传学,为第1个阶段。20世纪中叶,沃森和克里克发现了DNA双螺旋结构(1953年),开启了分子遗传学和分子生物学时代,为第2个阶段。此间,发现了生命遗传学密码和DNA-RNA-蛋白质生命科学中心法则,并催生了惠及人类的基因生物技术。20世纪和21世纪之交,随着“人类基因组计划”的实施和完成,生命科学进入组学和系统生物学时代,开启了第3个阶段。大量复杂的生命过程和疾病机制被阐明,基因组测序、合成与编辑,以及与人工智能的结合,正在谱写基因组的“读-编-写”新篇章,科学家开始书写合成生命体、精准调控生命过程。

|
| 图 1 生命科学里程碑事件与3个“50年” Figure 1 Landmark events and three 50-year stages in life sciences |
上述3个阶段也是环环相扣的3轮生命科学革命,其标志是生命科学研究范式发生深刻变化,从生物表观性状及遗传的观察描述到生命过程的分子生物学表征与关联,再到以组学为特征的系统生物学运用,广泛影响生命科学研究各领域,并引领医学、农业生物学等领域及其技术的全面进步,极大地贡献了人类健康和经济社会发展。
2 当代生命科学特征在新科技革命驱动下,生命科学呈现以下5个特征。
(1)原始发现层出不穷,底层创新呈现井喷。分子细胞生物学已经渗入生命科学的整个学科体系,成为生命科学各基础学科和应用学科的基础和支柱,促进层出不穷的原始性发现和底层创新。例如,强大的基因编辑技术[1, 2]源于微生物和古菌进化出对付病毒(噬菌体)感染的适应性免疫分子机制CRISPR的发现[3, 4];改变分子生物学面貌的聚合酶链式反应(PCR)技术源于极端微生物细胞中耐热DNA多聚酶的发现[5],免疫检查点(CTLA-4和PD-1)的发现[6, 7]和细胞免疫学研究导致了如火如荼的肿瘤靶向免疫治疗[8-10],正在颠覆传统的癌症治疗模式;RNA干扰(RNAi)机制的发现[11],启动了遗传病基因沉默的治疗路线;细胞凋亡(apoptosis)[12]、细胞焦亡(pyroptosis)[13]、细胞程序性坏死(necroptosis)[14]、细胞自噬(authophagy)[15]、细胞铁死亡(ferroptosis)[16]、细胞组分相变(transition)[17]等现象被发现,叙述着细胞在生理、病理过程中种种巧妙的自调节机制,并衍生出重大疾病治疗的新策略。
(2)系统论与还原论并重,层层揭秘复杂生命过程。分子生物学成功地注释了大量功能基因,并将许多生命过程和疾病发生机制与相关功能基因及转录和表达产物联系起来,若将此称为“还原论”,以基因组学、转录组学、蛋白组学和代谢组学等构成的生命组学,则是对复杂生命网络体系的系统性认识。“自下而上”与“自上而下”的结合,极大地提升了生命科学中发现的机会,催生了一个又一个新的研究方向和前沿热点。例如:整合基因组学、疾病分子生物学基础和临床数据,将精准医学与个体化治疗结合起来[18-20];人体微生物组及代谢组被发现与健康和许多疾病息息相关[21, 22],“肠道微生物组及代谢组也为解释中医药原理提供了一个新的视角” ①;通过基因组学和转录组研究及基因功能注释,发现仅有2% 基因组编码蛋白质,其余98% 功能未知部分被类比为基因组中的“暗物质” [23]。其中,大量非编码RNA被发现在细胞网络时空调控中发挥关键作用[24],这“为生物学开辟了一个全新的领域,……在未来的后续研究中具有无限的潜力” [25]。
① 仝小林,个人通信。
(3)学科汇聚融合,生命科学研究从定性描述开始实现动态、精准和定量解读。生命系统的遗传变异、代谢及调控的时空动态变化和生命物质的柔性等决定了生命过程的复杂性,目前所积累的生命科学系统知识多是大量定性片段结果的集成。超分辨显微成像、冷冻电镜、流式质谱、质谱成像、磁共振成像、增强拉曼光谱、膜片钳、光镊、纳米孔测序、纳米与分子生物传感、全微分析系统(µTAS)及器官芯片(organ-on-a-chip)、3D生物打印等各种物理和化学方法及技术平台的创建和应用,为生命科学研究提供了越来越强有力的工具,从而实现单细胞、可视化、高通量、高时空分辨分析和操纵。已经获得高分辨脑图谱[26, 27]、单细胞转录组[28, 29]、单细胞蛋白组[30]、胚胎细胞谱系[31]、活细胞中蛋白质3D结构测定[32]、单粒子病毒在活细胞中的示踪[33, 34]、芯片上多器官互作及类器官创建[35, 36]等。生命系统得以在微观层面被精准、定量、可视化表征,甚至被成功模拟。
(4)科学数据共享,成为生命科学界普遍遵循的规则。以基因数据库和蛋白质结构数据库为核心的各类生命科学数据库在现代生命科学研究中发挥巨大作用。数据库建设者与科学共同体形成一个准则:研究人员在使用数据库的同时,将自己研究发现的(基因序列或蛋白质结构)数据存放到数据库中,因而同时成为数据库的使用者和贡献者。如今,数据库已经成为生命史书最可靠的纪录载体和强大的数据分析平台,为整个生命科学研究所依赖。例如,新冠肺炎疫情全球大流行至今,新冠病毒基因组序列数据已经超过1 000万条②。这些数据通过全球共享流感数据倡议组织(GISAID)、中国国家基因组科学数据中心(CNCB)、美国国家生物技术信息中心(NCBI)和欧洲生物信息研究所(EBI)等共同实时发布,为新冠病毒病原生物学和分子流行病学研究、检测技术建立、药物和疫苗的研发提供依据,在全球科技抗疫中发挥了重大作用。
② https://ngdc.cncb.ac.cn/ncov/release_genome.
(5)合成生物学与人工智能(AI)兴起,为生命科学研究提供了新的范式。①合成生物学兴起与21世纪同步[37, 38],它汇聚生命科学、物理学、化学、材料科学、计算机与信息科学,并结合工程学理念和自动化技术,对生物体进行再设计与合成[39]。其“自下而上”的模式,从表征天然生物大分子,使之成为标准化“元件”,到创建“模块”和“线路”等生物部件和细胞“底盘”,来构建预期的人造生命系统,研究生命的底层规律。这一理念,将我们习以为常的“格物致知”研究策略,推进到了“建物致知”的新高度[40]。然而,鉴于生物体系的复杂性,目前生物体系的理性设计还依赖于高通量的“试错”实验,由此出现了“生物铸造工厂”(Biofoundary),即生物设计与合成自动化设施。也基于此,合成生物学的另一理念“造物致用”,正在催生未来生物技术。② AI基于大数据、算法和机器学习,应用于生命科学中最典型的例子是对蛋白质3D结构的预测。长期以来,蛋白质结构预测进展十分缓慢。对于一个未知结构的蛋白质,若没有其同源蛋白质的结构,则需要通过实验来测定其结构信息。谷歌公司DeepMind团队的AlphaFold在两年一届的“蛋白质结构预测技术的关键测试”(CASP)中脱颖而出之后,该团队于2021年在Nature上分享了AlphaFold2开源代码[41]。与此同时,美国华盛顿大学等团队也在Science上公布了新的深度学习工具RoseTTAFold[42]。接着,AlphaFold2又对人类98.5% 的蛋白质3D结构进行了高精度预测[43]。进而,DeepMind团队公布了AlphaFold蛋白质结构数据库,其使已知蛋白质序列空间的结构覆盖范围空前扩大;该数据库初始版本包含了21个模式生物蛋白质组中超过36万个预测结构,很快将扩展到涵盖UniRef90数据集(已验证的蛋白质序列)的大多数(超过1亿个)代表性序列[44]。这些进展对结构生物学技术是颠覆性的,体现在2个方面:①蛋白质3D结构数据将指数性增长,从而为机器学习提供更好的数据基础,将使AI结构预测目前还存在的质量缺陷逐一被解决;②由于蛋白质结构与功能是分子细胞生物学的基本科学问题,相关进展必将对生命科学产生深远影响。
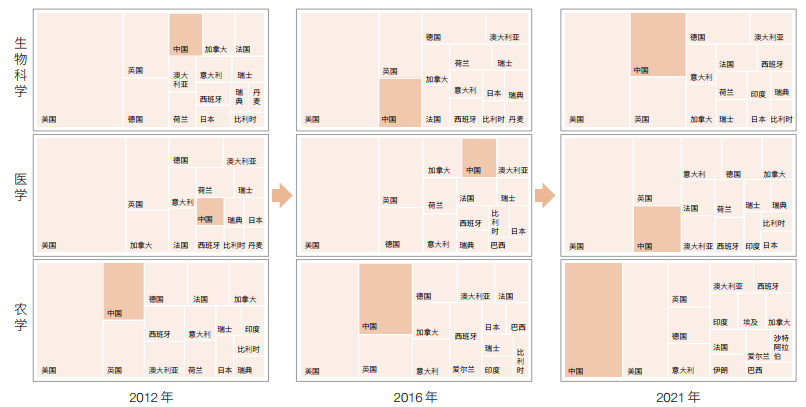
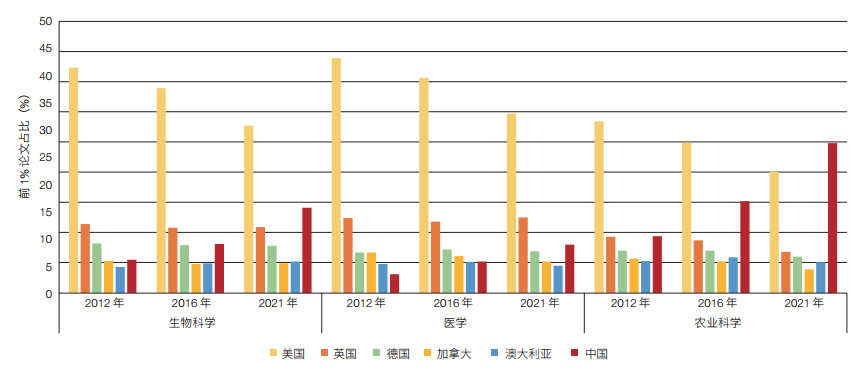
3 中国生命科学高影响力研究贡献 3.1 10年来高影响力论文情况分析用科学文献数据库数据构建可视化图谱,可以宏观地分析一个国家科学研究的贡献和发展水平。对一个快速发展中的科技大国而言,用论文总数和篇均被引用数来评价都显然偏颇,论文总引用数相对合理[45]。然而,如今中国研究与试验发展(R&D)全时人员当量已逾500万人年③,远超欧美各国,因此聚焦分析高影响力研究活动更有意义。高影响力论文包括高被引论文和热点论文。本文利用科睿唯安(Clarivate Analytics)公司的InCite科研评估分析平台仅对被引用次数在同领域方向前1% 以内的高被引论文(以下简称“前1% 论文”)进行分析。虽然可能会有少数例外(即其学术影响力并非真正高),而且科学论文不代表全部科技实力,但前1% 论文总体上反映各方向上有实质性贡献、出类拔萃的研究。图 2为全球生命科学前1% 论文数量产出最多的前15个国家(以下简称“15强”)构成的版图,展现近10年来高影响力研究产出在主要科技大国中的分布和相对位次及变化。中国生命科学研究在底子极薄、“跟踪发展” 20年后,于21世纪第1个10年开始进入视野,以后呈现持续、强劲的增长势头。在2012年、2016年和2021年的10年跨越中,中国学者发表的前1% 论文在15强总数中的占比分别为:生物科学(biological sciences),5.5%、8.1% 和14.1%;医学(medical sciences),3.1%、5.2% 和8.0%;农学(agriculture sciences),9.4%、15.2% 和24.8%(图 3)。本分析主要基于各领域大的学科方向,部分分支学科或收集未尽,或在三大领域中有交叉,但不影响统计结果所展现的总体趋势;另,中国港澳台地区学者的贡献暂未统计。
③ 中华人民共和国科学技术部. 中国科技人才发展报告(2020). 北京: 科学技术文献出版社, 2021.
|
| 图 2 全球生命科学格局中的中国:10年来进步显著 Figure 2 Changes in global life sciences landscape show that China has made remarkable progress over past 10 yearss 图中各方格面积大小表示某一国前1% 论文产出量及其在世界前15强中的位次;根据科睿唯安InCite分析平台原始数据绘制 Area of each square in figures represents number of high impact (top 1% highly cited) papers produced by country and its ranking in world top 15. Drawn from Clarivate Analytics's InCite/Web of Science data |
|
| 图 3 10年来中国及年生命科学5强的高影响力论文在15强中的占比变迁 Figure 3 Ten years' changes of the proportion of high influential papers in life sciences published by selected countries in total number of high influential papers published by top 15 countries in life sciences. 中国及生命科学5强国发表的高被引论文分别在15强(图 2)发表的总高被引论文中的占比及10年来的变化。数据采集时间间隔为5年,即,2012年、2016年和2021年;根据科睿唯安InCite/Web of Science原始数据绘制 For top 1% high influential papers in life sciences, the calculated is output proportion of top 5 countries in total output of top 15 countries and changes over past 10 years. Data collection is from 2012, 2016 and 2021. See Figure 2 for top 15 countries in life sciences. Drawn from Clarivate Analytics's InCite/Web of Science data |
中国生命科学高影响力研究显著性增多,代表整个研究水平实质性提升,这可归于4点:①中国对科技的重视,全社会R&D投入经费持续近20年的2位数增长,为生命科学研发提供了保障性资金。②科研队伍不断壮大,2013年以来中国R&D人员总数保持世界第一,包括学成回国和本土成长人才,形成了人才高地的基础。③国家围绕科学前沿领域设立了各类科技专项计划,并大幅改善科研条件和实验平台,国家自然科学基金的资助力度也不断加强。④广泛的国际合作。例如,近20年中,中国的国际合作论文为23% 以上,其中,高影响力合作论文占比2.9%,是中国发表的全部论文中高影响力论文占比比例的2倍。生命科学领域情况也类似。
3.2 近10年中国生命科学领域的重要研究成果举例如果说高被引用论文经过时间考验,属于回顾性的,那么每年由科技界评选出来的年度科学进展则属于科学热点。中国有2项生命科学年度进展相关评选活动。①科学技术部高技术研究发展中心(基础研究管理中心)组织的“中国十大科学进展”,始于21世纪初,由两院院士、国家重点实验室主任群和“973”计划项目首席科学家群投票产生;②年度“中国生命科学领域十大进展”,创办于2015年,由中国科学技术协会生命科学学会联合体选出。这2项评选活动所产生的结果通常高度吻合。前者历经20年,笔者过去因工作关系常常应邀对评选结果作整体点评,印象比较深。这里,对其近10年评选结果进行梳理,这些研究大多有明确的问题导向,或在科学上产生重要影响,或在医学和粮食安全等方面具有重要价值。
(1)分子遗传学和细胞生物学领域。发现精子RNA可作为记忆载体将获得性性状跨代遗传[46];揭示了一种精细的DNA复制起始位点的识别调控[47],Tet双加氧酶在哺乳动物表观遗传调控中的作用[48-50],以及人类原始生殖细胞基因表达与表观遗传调控特征等分子机制[51];发现营养匮乏引发细胞自噬的分子机制[52, 53]等。表观遗传学成果连续上榜,反映出分子遗传学的发展趋势;其中,中国学者贡献了全球15.5% 的高引论文(2011—2021年)。
(2)结构生物学领域。破解藻类水下光合作用的蛋白结构和功能[54];揭示非洲猪瘟病毒结构及其组装机制[55];揭示RNA剪接的关键分子机制[56, 57];解析人源葡萄糖转运蛋白结构[58]、阿尔茨海默病致病蛋白三维结构[59]、TAL效应蛋白特异性识别DNA的结构基础[60]。这些重要蛋白质复合物结构的解析,对理解其分子机器机理的意义重大。自从装备了冷冻电镜以后,中国学者解析了大批重要的蛋白质三维结构,包括一批新冠病毒重要蛋白质结构。然而,如前所述,AI预测蛋白质3D结构的能力越来越强大,结构生物学家已经在思考如何乘潮流而动、更加深入开展蛋白质结构与功能研究④。
④ 香山会议S63:人工智能与结构生物学。
(3)干细胞与再生医学、生殖与发育领域。中国科学家在早期率先用iPS干细胞克隆出小鼠以后[61, 62],逐渐在细胞和动物克隆领域形成强大阵容。近期代表成果包括:深度解析多器官衰老的标记物和干预靶标[63];证实单倍体孤雄干细胞具有可替代精子和快速传递基因修饰的能力[64, 65];基于体细胞核移植技术成功克隆出猕猴[66]。近5年,中国学者在该领域高被引论文中的贡献率为20.3%。中国科学院战略性先导科技专项“器官重建与制造”从器官原位再生、体外制造和异体再造等方面系统部署,挑战高等哺乳类复杂器官难以再生的根本问题;目前,该专项已经取得一批重要的原始性发现和技术创新,部分已经进入临床研究。
(4)重大疾病机理与诊疗方面。提出基于DNA检测酶调控的自身免疫疾病治疗方案[67];构建出世界上首个非人灵长类自闭症模型[68];揭示抑郁发生及氯胺酮快速抗抑郁[69];建立胆固醇代谢调控的肿瘤免疫治疗新方法[70]。两种天然产物靶向特异蛋白治疗白血病[71, 72]是中国科学家在率先成功治愈急性早幼粒白血病基础上的系统性贡献。中国学者首次揭示细胞炎性坏死(焦亡)的关键分子机制,为多种自身炎症性疾病提供了一个全新的药物靶点,重新定义了细胞焦亡的概念,开辟了一个新的程序性细胞坏死的研究方向[13]。
(5)病原生物学与传染病领域。虽然,新冠病毒变异株还在困扰人类,但必须看到,中国学者在应对新冠肺炎疫情中作出了系统性贡献,从病原生物学[73, 74]、结构生物学[75-77]、分子流行病学[78]、免疫学[79]、检测技术[80]、药物[81]、疫苗研发和应用[82],到临床诊治[83]获得大量重大成果,高被引用论文1 450余篇,热点论文200余篇,占比均为世界总数的20%。其中,“揭示新冠病毒(SARS-CoV-2)逃逸抗病毒药物机制” [75]被评选为“2021年度中国十大科学进展”。其他重大进展包括:揭示了埃博拉病毒演化及遗传多样性特征[84];在H7N9禽流感病毒溯源和H5N1禽流感跨种间传播机制研究中获突破[85];乙型肝炎病毒(HBV)受体肝脏牛磺胆酸钠共转运多肽(NTCP)的发现为解决实验中HBV感染细胞难题和发展抗乙肝药物奠定了重要基础[86];通过揭示非洲猪瘟病毒结构及其组装机制[55],为解决抗非洲猪瘟病毒疫苗这个世界难题提供了基于结构生物学的解决方案。
(6)农业生物学领域。调控植物生长—代谢平衡实现可持续农业发展[87];揭示水稻产量性状杂种优势的分子遗传机制[88];揭示蝗虫聚群成灾的奥秘——4-乙烯基苯甲醚是蝗虫的群聚信息素[89];生态学试验证实Bt转基因棉花种植可促进对害虫的生物控制[90]。尤其重要的是,继半矮秆形状基因为代表的“绿色革命”之后,以袁隆平为代表的中国科学家成功实现了水稻杂交育种理论与技术的突破,是第二次飞跃。后来袁隆平倾其毕生精力培育的超级稻,亩产首破1 000 kg(2014年中国十大科技进展新闻)。2017年,李家洋团队的“水稻高产优质性状形成的分子机理及品种设计”获国家自然科学奖一等奖,被杂交小麦之父李振声誉为“继‘绿色革命’和杂交水稻后的第三次重大突破,标志着‘新绿色革命’的起点” ⑤。中国学者的贡献,为全球粮食安全提供了重要技术路径,是对人类的重大贡献。
⑤ 为祖国种好一棵水稻——记荣获国家自然科学奖一等奖的李家洋团队. 中国科学报, 2018-01-10(01).
(7)合成生物学领域。实现酵母长染色体的精准定制合成[91-93]、创建出首例人造单染色体真核细胞[94],从而打开了染色体工程新的研究窗口;通过拓展密码子将病毒直接转化为活疫苗,安全且有效[95];设计了用于肿瘤治疗的智能型DNA纳米机器人[96]。近5年,中国学者在高引用论文和热点论文中的贡献均超过20%。自从2021年创建了从二氧化碳到淀粉的无细胞人工合成途径[97]以来,2022年又相继报道了最简酶促合成碳固定循环[98]和从二氧化碳还原合成葡萄糖和脂肪酸[99]。这些开创性研究试图将二氧化碳资源化,为工业生物制造提供多种路径,并有可能服务于“双碳”目标。
(8)生物起源与演化。中国学者曾在澄江动物化石群取得了一系列举世瞩目的成果,被誉为“20世纪最惊人的科学发现之一”。近期中国学者采用古基因组研究揭示了近万年来中国人群的演化与迁徙历史[100],用大数据刻画出迄今最高精度的地球3亿年生物多样性演变历史[101],足见现代技术对经典传统学科的影响。
(9)模式动物和实验工具。基于体细胞核移植技术成功克隆出猕猴[66],创建了非人灵长类自闭症模型[68],这些最接近人类的模式动物将为医学研究提供重要的支撑。中国物理学家、化学家和信息技术专家创造出多种科学工具,对生命科学的贡献不胜枚举,如可实现自由状态脑成像的微型显微成像系统[102]、单个蛋白质分子的磁共振探测[103]等。
限于篇幅,不能对这些成果展开论述,更多的开创性学术成果可参见年度“中国生命科学领域十大进展”等;它们每一项都是科学珍珠,其中部分应用潜力无限。
4 中国生命科学研究未来发展趋势研判和建议 4.1 发展趋势中国生命科学厚积薄发,10年进步显著,悄然改写世界生命科学版图。科技抗击新冠肺炎疫情是一个缩影。其间,中国科学家经受严峻考验,从基础研究到临床,表现可圈可点。发表前1% 论文数量仅次于美国,单篇引用达成千上万次的前20篇论文大部分来自中国。其中,率先鉴定出病原并给出基因组序列、明确细胞ACE2是病毒的受体、提供临床治疗方案和经验等,对全球新冠肺炎疫情防控作出重大基础性共享。
中国生命科学未来发展,已具备3个优势:①研究水平普遍提高。这已经被图 2和3的宏观数据证明,而且几乎在生命科学各个领域都有卓越的研究成果;从趋势来看,继续扩大影响力属于必然。②队伍不断发展壮大。在各个领域都有数量可观的团队在活跃着,形成金字塔人才结构,且仍然有青年人才红利。一个令人鼓舞的例子是,中国每年有100多支学生团队参加美国麻省理工学院创办的国际遗传工程机器(合成生物学,iGEM)大赛;一届接一届,保持了30%—40% 的金牌纪录,这说明中国在生命科学及其交叉学科领域有优秀的青年后备军。③国家高度重视生命科学。已经设立了脑计划、干细胞与器官修复、合成生物学、生物大分子机器、微生物组学、前沿生物技术、IT(信息技术)-BT(生物技术)、诊疗装备、中医药、慢病防治、生殖健康等一系列重点计划和专项,并有明确的战略发展目标。国家自然科学基金则强调鼓励探索、突出原创等指导方针,加上中国科学院战略性先导科技专项等,共同协调支持中国生命科学研究。
4.2 问题分析和措施建议中国生命科学在取得长足进步的同时,也有亟待要解决的问题及新的挑战。以下是相关思考及建议。
(1)理顺各科技创新模块的定位分工和资源配置模式。经过40多年发展和持续改革,中国生命科学研究力量分布已经形成几大模块。由于模块之间并无明确分工,且资源配置模式几无差别,在大家奋力前行的同时,也形成同质化和无序竞争。例如,一个生命科学基础研究国家机构,70% 以上的年度经费需要通过与其他模块同行竞争获得,这不同于国际上的通行做法,难以形成“铁打的营盘”和“百年老店”(即国际知名研究机构),也增加了整个科研体系的管理成本。希望在新的改革中予以解决。
(2)处理好渐进式研究和颠覆性创新的关系。由于历史的原因,生命科学理论知识体系和现代重大科学发现及创新主要源于欧美科学家的贡献。数十年来,中国科技逐渐告别跟踪性研究模式,历史性地站到了科学发展前沿,做更多“0到1”的创新是众望所归。笔者曾经与澳大利亚前政府首席科学顾问Alan Finkel谈及渐进式科研和颠覆性科研,得到的共识是,量变到质变是真实地存在。例如,正是由于在SARS病毒溯源方向长期的“冷板凳”研究,中国病毒学家在新冠肺炎疫情发生初期就能及时担当。在挑战重大科学命题和应用目标的同时,坚持广泛支持探索性研究,也同样重要。“从0到1”的创新常常刷新我们的三观,这本身就说明颠覆性创新难以“预测”和“设计”,它们源自创新沃土、科学海洋。
(3)加快解决生物技术转化的难题。生物技术对人类健康和粮食安全作出了巨大贡献。中国农业科技进步贡献率已经超过60%⑥,但与发达经济体的现代化农业相比还有不少差距。医药技术和生物医学工程的差距更明显,此不赘述。这里有3个原因:①竞争性的技术源于原始性创新,原始性创新需要大量非急功近利的研究积累——积累,是绕不过去的坎。②以论文为唯一导向的评价活动还普遍存在,不少研究活动是为了论文而做论文。结果是,单位的论文指标上去了,转化研究人员却常常被边缘化,这也导致科研成果供给不足。③产学研机制在许多地区并没有实质性建立,从实验室到产业之间的鸿沟难以逾越。科技开发园区有不少成功的经验值得推广。例如,深圳市光明区政府与中国科学院深圳先进技术研究院共同创造的“楼上楼下创新创业综合体”是一个很好的模式,“将研究板块与初创企业的空间距离压缩为零” ⑦。
⑥ 《“十三五”中国农业农村科技发展报告》发布. (2022-11-19). http://www.gov.cn/xinwen/2021-11/22/content_5652387.htm.
⑦ 光明科学城:楼上创新楼下创业“沿途下蛋”融合发展. (2021-08-19)[2022-05-06]. https://baijiahao.baidu.com/s?id=1708478916127079948&wfr=spider&for=pc.
(4)营造先进的科研文化和氛围。笔者曾与英国学者共同主持国家自然科学基金-牛顿基金支持的“促进新生物科技的公众理解”研讨班⑧,分析和讨论新兴科研议题的公众参与及沟通模式、生命科学研究科研范式、科研管理、互信互助、知识共享、科学伦理与法律等。参与者的热情和智慧、前瞻性理念、乐于分享与相互启发的精神,让笔者想起许多校训、院训及其对莘莘学子的精神陶冶,先进的科研文化和环境氛围孕育先进的科学思想。反观,争“帽子”和“打招呼”风气一直以来深深困扰一线研究人员,侵蚀着科学精神,其危害不可小觑,需要根治。
⑧ “中英促进新生物科技的公众理解双边研讨会”. (2020-05-29). http://school.freekaoyan.com/bj/ibp/2020/05-29/15907559101192792.shtml.
(5)坚持开放性研究和国际合作。生命科学是人类命运共同体的联系纽带,具有长期的国际合作传统。但近些年,世界政治经济格局发生深刻变化,国际合作的传统被践踏。对InCtie/Web of Science数据库分析可见,中美学者合作发表的科学论文数量持续增长数十年并相互成为最大的合作伙伴,2019年双方合作论文达66 600多篇,为历史最高。然而,2020年首度下降,减少1.5%,2021年继续下降5.9%(生命科学合作论文减少9%),2022年可能出现断崖式下降。此形势下,中国生命科学可充分利用世界卫生组织(WHO)和国际粮食及农业组织(FAO)等联合国和政府间组织机制、生命科学各类学术社团的平台和网络多媒体等形式,坚持开展合作研究,世界终将回归理性。
6 结语中国生命科学整体上已经转向高质量发展阶段。在继续扩大优势的同时,解决好存在的问题,用新的辉煌,建设健康中国、平安中国,打造科技强国,贡献人类。
致谢 感谢科睿唯安公司提供的Web of Science文献数据库和InCite分析平台的支持。| [1] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337: 816-821. DOI:10.1126/science.1225829 |
| [2] |
Cong L, Ran F A, Cox D., et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339: 819-823. DOI:10.1126/science.1231143 |
| [3] |
Mojica F J M, Diez-Villaseñor C, García-Martinez J, et al. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. Journal of Molecular Evolution, 2005, 60(2): 174-182. DOI:10.1007/s00239-004-0046-3 |
| [4] |
Pourcel C, Salvignol G, Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiolog, 2005, 151: 653-663. |
| [5] |
Saiki R K, Gelfand D H, Stoffel S, et al. Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science, 1988, 239: 487-491. DOI:10.1126/science.2448875 |
| [6] |
Leach D R, Krummel M F, Allison J P. Enhancement of antitumor immunity by CTLA-4 blockade. Science, 1996, 271: 1734-1736. DOI:10.1126/science.271.5256.1734 |
| [7] |
Freeman G J, Long A J, Iwai Y, et al. Engagement of the PD-1 immunoinhibitory receptor by a novel B7 family member leads to negative regulation of lymphocyte activation. The Journal of Experimental Medicine, 2000, 192(7): 1027-1034. DOI:10.1084/jem.192.7.1027 |
| [8] |
Pardoll D M. The blockade of immune checkpoints in cancer immunotherapy. Nature Reviews Cancer, 2012, 12(4): 252-264. DOI:10.1038/nrc3239 |
| [9] |
Waldman A D, Fritz J M, Lenardo M J. A guide to cancer immunotherapy: From T cell basic science to clinical practice. Nature Reviews Immunology, 2020, 20(11): 651-668. DOI:10.1038/s41577-020-0306-5 |
| [10] |
Hegde P S, Chen D S. Top 10 challenges in cancer immunotherapy. Immunity, 2020, 52(1): 17-35. DOI:10.1016/j.immuni.2019.12.011 |
| [11] |
Fire A, Xu S Q, Montgomery M K, et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature, 1998, 391: 806-811. DOI:10.1038/35888 |
| [12] |
Elmore S. Apoptosis: A review of programmed cell death. Toxicologic Pathology, 2007, 35(4): 495-516. DOI:10.1080/01926230701320337 |
| [13] |
Shi J J, Zhao Y, Wang K, et al. Cleavage of GSDMD by inflammatory caspases determines pyroptotic cell death. Nature, 2015, 526: 660-665. DOI:10.1038/nature15514 |
| [14] |
Vandenabeele P, Galluzzi L, Vanden Berghe T, et al. Molecular mechanisms of necroptosis: An ordered cellular explosion. Nature Reviews Molecular Cell Biology, 2010, 11(10): 700-714. DOI:10.1038/nrm2970 |
| [15] |
Levine B, Kroemer G. Autophagy in the pathogenesis of disease. Cell, 2008, 132(1): 27-42. DOI:10.1016/j.cell.2007.12.018 |
| [16] |
Chen X, Kang R, Kroemer G, et al. Broadening horizons: The role of ferroptosis in cancer. Nature Reviews Clinical Oncology, 2021, 18(5): 280-296. DOI:10.1038/s41571-020-00462-0 |
| [17] |
Dolgin E. What lava lamps and vinaigrette can teach us about cell biology. Nature, 2018, 555: 300-302. DOI:10.1038/d41586-018-03070-2 |
| [18] |
Jameson J L, Longo D L. Precision medicine—PPersonalized, problematic and promising. The New England Journal of Medicine, 2015, 372(23): 2229-2234. DOI:10.1056/NEJMsb1503104 |
| [19] |
Collins F S, Varmus H. A new initiative on precision medicine. The New England Journal of Medicine, 2015, 372(9): 793-795. DOI:10.1056/NEJMp1500523 |
| [20] |
Ashley E A. Towards precision medicine. Nature Reviews Genetics, 2016, 17(9): 507-522. DOI:10.1038/nrg.2016.86 |
| [21] |
Turnbaugh P J, Ley R E, Mahowald M A, et al. An obesity associated gut microbiome with increased capacity for energy harvest. Nature, 2006, 444: 1027-1031. DOI:10.1038/nature05414 |
| [22] |
Fan Y, Pedersen O. Gut microbiota in human metabolic health and disease. Nature Reviews Microbiology, 2021, 19(1): 55-71. DOI:10.1038/s41579-020-0433-9 |
| [23] |
Blaxter M. R Genetics. evealing the dark matter of the genome. Science, 2010, 330: 1758-1759. DOI:10.1126/science.1200700 |
| [24] |
Xue Y C, Chen R S, Qu L H, et al. Noncoding RNA: From dark matter to bright star. Science China-Life Sciences, 2020, 63(4): 463-468. DOI:10.1007/s11427-020-1676-5 |
| [25] |
Everett L J, Huang W, Zhou S S, et al. Gene expression networks in the Drosophila Genetic Reference Panel. Genome Research, 2020, 30(3): 485-496. DOI:10.1101/gr.257592.119 |
| [26] |
Feng Z, Li A A, Gong H, et al. Constructing the rodent stereotaxic brain atlas: A survey. Science China-Life Sciences, 2022, 65(1): 93-106. DOI:10.1007/s11427-020-1911-9 |
| [27] |
Fan L Z, Li H, Zhuo J J, et al. The human brainnetome atlas: a new brain atlas based on connectional architecture. Cerebral Cortex, 2016, 26(8): 3508-3526. DOI:10.1093/cercor/bhw157 |
| [28] |
Tang F C, Barbacioru C, Wang Y Z, et al. mRNA-Seq whole transcriptome analysis of a single cell. Nature Methods, 2009, 6(5): 377-382. DOI:10.1038/nmeth.1315 |
| [29] |
Ren X W, Wen W, Fan X Y, et al. COVID-19 immune features revealed by a large-scale single-cell transcriptome atlas. Cell, 2021, 184(7): 1895-1913. DOI:10.1016/j.cell.2021.01.053 |
| [30] |
Cong Y Z, Motamedchaboki K, Misal S A, et al. Ultrasensitive single-cell proteomics wokflow identifies>1000 protein groups per mammalian cell. Chemical Science, 2020, 12(3): 1001-1006. |
| [31] |
Peng G D, Suo S B, Cui G Z, et al. Molecular architecture of lineage allocation and tissue organization in early mouse embryo. Nature, 2019, 572: 528-532. DOI:10.1038/s41586-019-1469-8 |
| [32] |
Tanaka T, Ikeya T, Kamoshida H, et al. High-resolution protein 3D structure determination in living eukaryotic cells. Angewandte Chemie International Edition, 2009, 131(22): 7362-7366. |
| [33] |
Qin C, Li W, Li Q, et al. Real-time dissection of dynamic uncoating of individual influenza viruses. PNAS, 2019, 116(7): 2577-2582. DOI:10.1073/pnas.1812632116 |
| [34] |
Li X, Wang D B, Cui Z Q, et al. HIV-1 viral cores enter the nucleus collectively through the nuclear endocytosis-like pathway. Science China-Life Science, 2021, 64(1): 66-76. DOI:10.1007/s11427-020-1716-x |
| [35] |
Skardal A, Murphy S V, Devarasetty M, et al. Multi-tissue interactions in an integrated three-tissue organ-on-a-chip platform. Scientific Reports, 2017, 7: 88347. |
| [36] |
Tao T T, Wang Y Q, Chen W W, et al. Engineering human islet organoids from iPSCs using an organ-on-chip platform. Lab on a Chip, 2019, 19(6): 948-958. DOI:10.1039/C8LC01298A |
| [37] |
Cameron D E, Bashor C J, Collins J J. A brief history of synthetic biology. Nature Reviews Microbiology, 2014, 12(5): 381-390. DOI:10.1038/nrmicro3239 |
| [38] |
张先恩. 中国合成生物学发展回顾与展望. 中国科学: 生命科学, 2019, 49(12): 1543-1572. Zhang X E. Synthetic biology in China: Review and prospects. Scientia Sinica (Vitae), 2019, 49(12): 1543-1572. (in Chinese) |
| [39] |
Royal Society of Engineering. Synthetic Biology: scope, applications and implications, 2009.
|
| [40] |
赵国屏. 合成生物学: 生命科学的"利器". 人民日报, 2020-11-17(20). Zhao G P. Synthetic biology: The "edge tool" of life sciences. People's Daily, 2020-11-17(20). (in Chinese) |
| [41] |
Jumper J, Evans R, Pritzel A, et al. Highly accurate protein structure prediction with AlphaFold. Nature, 2021, 596: 583-589. DOI:10.1038/s41586-021-03819-2 |
| [42] |
Baek M, DiMaio F, Anishchenko I, et al. Accurate prediction of protein structures and interactions using a three-track neural network. Science, 2021, 373: 871-876. DOI:10.1126/science.abj8754 |
| [43] |
Tunyasuvunakool K, Adler J, Wu Z, et al. Highly accurate protein structure prediction for the human proteome. Nature, 2021, 596: 590-596. DOI:10.1038/s41586-021-03828-1 |
| [44] |
Varadi M, Anyango S, Deshpande M, et al. AlphaFold Protein Structure Database: Massively expanding the structural coverage of protein-sequence space with highaccuracy models. Nucleic Acids Research, 2021, 50(D1): D439-D444. |
| [45] |
Zhang X E. Forty years of highly synchronous development in China's scientific research and economy. National Science Review, 2018, 5(6): 796-798. DOI:10.1093/nsr/nwy064 |
| [46] |
Chen Q, Yan M H, Cao Z H, et al. Sperm tsRNAs contribute to intergenerational inheritance of an acquired metabolic disorder. Science, 2016, 351: 397-400. DOI:10.1126/science.aad7977 |
| [47] |
Long H Z, Zhang L W, Lv M J, et al. H2A.Z facilitates licensing and activation of early replication origins. Nature, 2020, 577: 576-581. DOI:10.1038/s41586-019-1877-9 |
| [48] |
He Y F, Li B Z, Li Z, et al. Tet-mediated formation of 5-carboxylcytosine and its excision by TDG in mammalian DNA. Science, 2011, 333: 1303-1307. DOI:10.1126/science.1210944 |
| [49] |
Gu T P, Guo F, Yang H, et al. The role of Tet3 DNA dioxygenase in epigenetic reprogramming by oocytes. Nature, 2011, 477: 606-610. DOI:10.1038/nature10443 |
| [50] |
Dai H Q, Wang B A, Yang L, et al. TET-mediated DNA demethylation controls gastrulation by regulating Lefty Nodal signalling. Nature, 2016, 538: 528-532. DOI:10.1038/nature20095 |
| [51] |
Guo F, Yan L Y, Guo H S, et al. The transcriptome and DNA methylome landscapes of human primordial germ cells. Cell, 2015, 161(6): 1437-1452. DOI:10.1016/j.cell.2015.05.015 |
| [52] |
Lin S Y, Li T Y, Liu Q, et al. GSK3-TIP60-ULK1 signaling pathway links growth factor deprivation to autophagy. Science, 2012, 336: 477-481. DOI:10.1126/science.1217032 |
| [53] |
Yi C, Ma M S, Ran L L, et al. Function and Molecular mechanism of acetylation in autophagy regulation. Science, 2012, 336: 474-477. DOI:10.1126/science.1216990 |
| [54] |
Pi X, Zhao S H, Wang W D, et al. The pigment-protein network of a diatom photosystem Ⅱ-light-harvesting antenna super complex. Science, 2019, 365: eaax4406. DOI:10.1126/science.aax4406 |
| [55] |
Wang N, Zhao D M, Wang J L, et al. Architecture of African swine fever virus and implications for viral assembly. Science, 2019, 366: 640-644. DOI:10.1126/science.aaz1439 |
| [56] |
Yan C Y, Wan R X, Bai R, et al. Structure of a yeast activated spliceosome at 3.5 Å resolution. Science, 2016, 353: 904-911. DOI:10.1126/science.aag0291 |
| [57] |
Wan R X, Yan C Y, Bai R, et al. Structure of a yeast catalytic step I spliceosome at 3.4 Å resolution. Science, 2016, 353: 895-904. DOI:10.1126/science.aag2235 |
| [58] |
Deng D, Yan C Y, Pan X J, et al. Structural basis for sequence-specific recognition of DNA by TAL effectors. Science, 2012, 335: 720-723. DOI:10.1126/science.1215670 |
| [59] |
Lu P L, Bai X C, Ma D, et al. Three-dimensional structure of human γ-secretase. Nature, 2014, 512: 166-170. DOI:10.1038/nature13567 |
| [60] |
Deng D, Yan C Y, Pan X J, et al. Structural Basis for Sequence-Specific Recognition of DNA by TAL Effectors. Science, 2012, 335: 720-723. DOI:10.1126/science.1215670 |
| [61] |
Zhao X Y, Li W, Lv Z, et al. iPS cells produce viable mice through tetraploid complementation. Nature, 2009, 461: 86-90. DOI:10.1038/nature08267 |
| [62] |
Kang L, Wang J L, Zhang Y, et al. iPS cells can support fullterm development of tetraploid blastocyst-complemented embryos. Cell Stem Cell, 2009, 5(2): 135-138. DOI:10.1016/j.stem.2009.07.001 |
| [63] |
Wang S, Zheng Y X, Li J Y, et al. Single-cell transcriptomicatlas of primate ovarian aging. Cell, 2020, 180(3): 585-600. DOI:10.1016/j.cell.2020.01.009 |
| [64] |
Yang H, Shi L Y, Wang B A, et al. Generation of genetically modified mice by oocyte injection of androgenetic haploid embryonic stem cells. Cell, 2012, 149(3): 605-617. DOI:10.1016/j.cell.2012.04.002 |
| [65] |
Li W, Shuai L, Wan H F, et al. Androgenetic haploid embryonic stem cells produce live transgenic mice. Nature, 2012, 490: 407-411. DOI:10.1038/nature11435 |
| [66] |
Liu Z, Cai Y J, Wang Y, et al. Cloning of macaque monkeys by somatic cell nuclear transfer. Cell, 2018, 172(4): 881-887. DOI:10.1016/j.cell.2018.01.020 |
| [67] |
Dai J, Huang Y J, He X H, et al. Acetylation blocks cGAS activity and inhibits self-DNA-induced autoimmunity. Cell, 2019, 176(6): 1447-1460. DOI:10.1016/j.cell.2019.01.016 |
| [68] |
Liu Z, Li X, Zhang J T, et al. Autism-like behaviours and germline transmission in transgenic monkeys overexpressing MeCP2. Nature, 2016, 530: 98-102. DOI:10.1038/nature16533 |
| [69] |
Zhang Y Y, Ye F, Zhang T T, et al. Structural basis of ketamine action on human NMDA receptors. Nature, 2021, 596: 301-305. DOI:10.1038/s41586-021-03769-9 |
| [70] |
Yang W, Bai Y B, Xiong Y, et al. Potentiating the antitumour response of CD8+ T cells by modulating cholesterol metabolism. Nature, 2016, 531: 651-655. DOI:10.1038/nature17412 |
| [71] |
Liu C X, Yin Q Q, Zhou H C, et al. Adenanthin targets peroxiredoxin Ⅰ and Ⅱ to induce differentiation of leukemic cells. Nature Chemical Biology, 2012, 8(5): 486-493. DOI:10.1038/nchembio.935 |
| [72] |
Zhen T, Wu C F, Liu P, et al. Targeting of AML1-ETO int(8;21) leukemia by oridonin generates a tumor suppressor like protein. Science Translational Medicine, 2012, 4: 127ra38. |
| [73] |
Zhou P, Yang X L, Wang X G, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature, 2020, 579: 270-273. DOI:10.1038/s41586-020-2012-7 |
| [74] |
Wu F, Zhao S, Yu B, et al. A new coronavirus associated with human respiratory disease in China. Nature, 2020, 579: 265-269. DOI:10.1038/s41586-020-2008-3 |
| [75] |
Yan L M, Ge J, Zheng L T, et al. Cryo-EM structure of an extended SARS-CoV-2 replication and transcription complex reveals an intermediate state in cap synthesis. Cell, 2021, 184(1): 184-193. DOI:10.1016/j.cell.2020.11.016 |
| [76] |
Jin Z M, Du X Y, Xu Y C, et al. Structure of M-pro from SARS-CoV-2 and discovery of its inhibitors. Nature, 2020, 582: 289-293. DOI:10.1038/s41586-020-2223-y |
| [77] |
Yan R H, Zhang Y Y, Li Y N, et al. Structural basis for the recognition of SARS-CoV-2 by full-length human ACE2. Science, 2020, 367: 1444-1448. DOI:10.1126/science.abb2762 |
| [78] |
Lu R J, Zhao X, Li J, et al. Genomic characterisation and epidemiology of 2019 novel coronavirus: Implications for virus origins and receptor binding. The Lancet, 2020, 395: 565-574. DOI:10.1016/S0140-6736(20)30251-8 |
| [79] |
Zhao J J, Yuan Q, Wang H Y, et al. Antibody responses to SARS-CoV-2 in patients with novel coronavirus disease 2019. Clinical Infectious Diseases, 2020, 71(16): 2027-2034. DOI:10.1093/cid/ciaa344 |
| [80] |
Wang W L, Xu Y L, Gao R Q, et al. Detection of SARS-CoV-2 in different types of clinical specimens. JAMA, 2020, 323(18): 1843-1844. |
| [81] |
Wang M L, Cao R Y, Zhang L K, et al. Remdesivir and chloroquine effectively inhibit the recently emerged novel coronavirus (2019-nCoV) in vitro. Cell Research, 2020, 30(3): 269-271. DOI:10.1038/s41422-020-0282-0 |
| [82] |
Xia S L, Zhang Y T, Wang Y X, et al. Safety and immunogenicity of an inactivated SARS-CoV-2 vaccine, BBIBP-CorV: A randomised, double-blind, placebo controlled, phase 1/2 trial. The Lancet Infectious Diseases, 2021, 21(1): 39-51. DOI:10.1016/S1473-3099(20)30831-8 |
| [83] |
Zhou F, Yu T, Du R H, et al. Clinical course and risk factors for mortality of adult inpatients with COVID-19 in Wuhan, China: A retrospective cohort study. The Lancet, 2020, 395: 1054-1062. DOI:10.1016/S0140-6736(20)30566-3 |
| [84] |
Tong Y G, Shi W F, Liu D, et al. Genetic diversity and evolutionary dynamics of Ebola virus in Sierra Leone. Nature, 2015, 524: 93-96. DOI:10.1038/nature14490 |
| [85] |
Shi Y, Zhang W, Wang F, et al. Structures and receptor binding of hemagglutinins from human-infecting H7N9 influenza viruses. Science, 2013, 342: 243-247. DOI:10.1126/science.1242917 |
| [86] |
Yan H, Zhong G C, Xu G W, et al. Sodium taurocholate cotransporting polypeptide is a functional receptor for human hepatitis B and D virus. eLife, 2012, 1: e00049. DOI:10.7554/eLife.00049 |
| [87] |
Li S, Tian Y H, Wu K, et al. Modulating plant growth metabolism coordination for sustainable agriculture. Nature, 2018, 560: 595-600. DOI:10.1038/s41586-018-0415-5 |
| [88] |
Huang X H, Yang S H, Gong J Y, et al. Genomic architecture of heterosis for yield traits in rice. Nature, 2016, 537: 629-633. DOI:10.1038/nature19760 |
| [89] |
Guo X J, Yu Q Q, Chen D F, et al. 4-vinylanisole is an aggregation pheromone in locusts. Nature, 2020, 584: 584-588. DOI:10.1038/s41586-020-2610-4 |
| [90] |
Lu Y H, Wu K M, Jiang Y Y, et al. Widespread adoption of Bt cotton and insecticide decrease promotes biocontrol services. Nature, 2012, 487: 362-365. DOI:10.1038/nature11153 |
| [91] |
Xie Z X, Li B Z, Mitchell L A, et al. "Perfect" designer chromosome V and behavior of a ring derivative. Science, 2017, 355: eaaf4704. DOI:10.1126/science.aaf4704 |
| [92] |
Shen Y, Wang Y, Chen T, et al. Deep functional analysis of synⅡ, a 770-kilobase synthetic yeast chromosome. Science, 2017, 355: eaaf4791. DOI:10.1126/science.aaf4791 |
| [93] |
Zhang W M, zhao G H, Luo Z Q, et al. Engineering the ribosomal DNA in a megabase synthetic chromosome. Science, 2017, 355: eaaf3981. DOI:10.1126/science.aaf3981 |
| [94] |
Shao Y Y, Lu N, Wu Z F, et al. Creating a functional single chromosome yeast. Nature, 2018, 560: 331-335. DOI:10.1038/s41586-018-0382-x |
| [95] |
Si L L, Xu H, Zhou X Y, et al. Generation of influenza A viruses as live but replication-incompetent virus vaccines. Science, 2016, 354: 1170-1173. DOI:10.1126/science.aah5869 |
| [96] |
Li S P, Jiang Q, Liu S L, et al. A DNA nanorobot functions as a cancer therapeutic in response to a molecular trigger in vivo. Nature Biotechnology, 2018, 36(3): 258-264. DOI:10.1038/nbt.4071 |
| [97] |
Cai T, Sun H B, Qiao J, et al. Cell-free chemoenzymatic starch synthesis from carbon dioxide. Science, 2021, 373: 1523-1527. DOI:10.1126/science.abh4049 |
| [98] |
Xiao L, Liu G X, Gong F Y, et al. A minimized synthetic carbon fixation cycle. ACS Catalysis, 2022, 12(1): 799-808. DOI:10.1021/acscatal.1c04151 |
| [99] |
Zheng T T, Zhang M L, Wu L H, et al. Upcycling CO2 into energy-rich long-chain compounds via electrochemical and metabolic engineering. Nature Catalysis, 2022. DOI:10.1038/s41929-022-00775-6 |
| [100] |
Yang M A, Fan X C, Sun B, et al. Ancient DNA indicates human population shifts and admixture in northern and southern China. Science, 2020, 369: 282-288. DOI:10.1126/science.aba0909 |
| [101] |
Fan J X, Shen S Z, Erwin D H, et al. A high-resolution summary of Cambrian to Early Triassic marine invertebrate biodiversity. Science, 2020, 367: 273-277. |
| [102] |
Zong W J, Wu R L, Li M L, et al. Fast high-resolution miniature two-photon microscopy for brain imaging in freely behaving mice. Nature Methods, 2017, 14(7): 713-719. DOI:10.1038/nmeth.4305 |
| [103] |
Shi F Z, Zhang Q, Wang P F, et al. Protein imaging.Single-protein spin resonance spectroscopy under ambient conditions. Science, 2015, 347: 1135-1138. DOI:10.1126/science.aaa2253 |



