2. 北京干细胞与再生医学研究院 北京 100101;
3. 中国科学院 前沿科学与教育局 北京 100864
2. Beijing Institute for Stem Cell and Regenerative Medicine, Beijing 100101, China;
3. Bureau of Frontier Sciences and Education, Chinese Academy of Sciences, Beijing 100864, China
2007年,图灵奖得主吉姆·格雷(Jim Gray)提出了科学研究的四类范式,这些范式基本上被科学界广泛认可。第一范式是实验(经验)科学,主要通过实验或经验来描述自然现象并总结规律;第二范式是理论科学,科学家通过数学模型进行归纳总结形成科学理论;第三范式是计算科学,利用计算机对科学实验进行模拟仿真;第四范式是数据科学,利用仪器收集或仿真计算产生的大量数据进行分析与知识提取。科学研究的范式变革体现了人类对宇宙探索的深度、广度、方式和效率的演进。
生命科学的发展经历了多个阶段,其研究范式的演进也有其独特的学科属性。在生命科学早期发展阶段,生物学家主要通过观察不同生物体的形态和行为模式来探索生物存在的一般形式和演化的共同规律,这一阶段的代表是达尔文,通过全球考察积累了大量物种的表象描述资料,并以此提出了进化论。从20世纪中叶开始,以DNA双螺旋结构的揭示为标志,生命科学研究进入了分子生物学时代,生物学家开始在更深层次水平研究生命的基本组成和运作规律。在这一阶段,生物学家仍主要通过对生物现象的观察和实验来总结规律与知识。随着生命科学的进一步发展和新型生物技术的快速涌现,科学家可以对生命科学在不同层级和不同分辨率下进行更为广泛的探索,这也使得生命科学领域的数据呈现爆发性增长。通过高通量、多维度组学数据分析与实验科学结合的方式对生物过程进行更加精细的描述和解析,成为现代生命科学研究的常态。
然而,生命系统具有多层面的复杂性,涵盖了从分子、细胞到个体不同层次,以及个体间的种群关系、机体与环境的互作关系,展现出多层级、高维度、高度互联、动态调控的特点。现有的实验科学研究范式在面对如此复杂的生命系统时,往往只能从特定尺度对有限数量的样本进行观察描述和研究,难以全面理解生物网络的运作机制;并且高度依赖人的经验和先验知识对特定生物关系进行探索,难以从大规模、多样性、高维度数据中高效提取隐匿的关联和机制。面对生命现象中复杂的非线性关系和难以预测的特征,人工智能(AI)技术展现出强大的能力,并且已经在蛋白质结构预测、基因调控网络模拟解析方面表现出颠覆性的应用潜力,将生命科学研究由实验科学为主的第一范式推向以人工智能驱动的生命科学研究新范式——第五范式(图 1)。
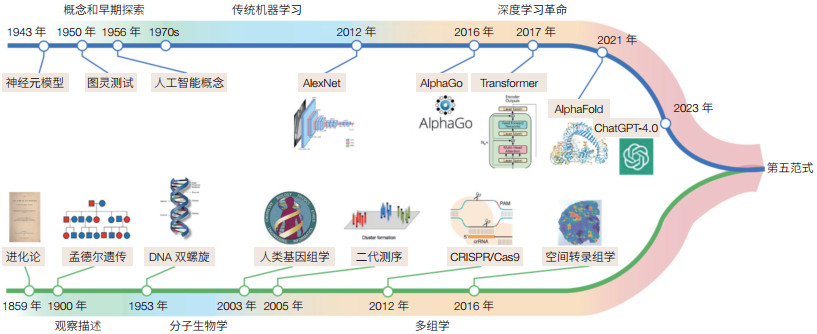
|
| 图 1 生命科学与人工智能的发展简述 Figure 1 Brief overview of development of life sciences and artificial intelligence |
本文将从AI驱动的生命科学研究典型范例、生命科学研究新范式的内涵和关键要素、新范式赋能的生命科学研究前沿及我国面临的挑战3个方面进行系统论述。
1 人工智能驱动的生命科学研究典型范例生命是一个多层次、多尺度、动态互联、相互影响的复杂系统。在面对生命现象的极端复杂性、多尺度跨越和时空动态变化时,传统的生命科学研究范式往往只能从局部入手,通过实验验证或有限层次的组学数据分析建立有限生物分子和表型的关联关系。然而,即使花费巨大成本,也通常只能发现特定情境下的单一线性关联机制,与生命活动的非线性属性在复杂度上存在显著差异[1],难以全面理解整个网络的运作机制。
AI技术,尤其是深度学习和预训练大模型等技术,以其优越的模式识别和特征提取能力,能够在庞大的参数堆叠情况下超越人类理性推理能力,从数据中更好地理解复杂生物系统中的规律。现代生物技术的不断发展,使生命科学领域的数据呈现跨越式增长,在过去全球范围生命科学研究中,人类已经积累了大量基于实验描述和验证的数据,为AI破解生命科学底层规律创造了基础[2]。当拥有充足且高质量的数据和适配于生命科学的算法时,AI模型就能够在多层次的海量数据中以“低维”数据预测“高维”信息及规律,实现从基因序列和表达等低维数据到细胞、机体等高维复杂生物过程规律揭示的跨越,解析复杂的非线性关系,如生物大分子结构生成规律、基因表达调控机制,甚至个体发育、衰老等多因素交叉的复杂生物系统中的底层规律。在此发展趋势下,近年来生命科学领域涌现出了蛋白质结构解析、基因调控规律解析等一批AI驱动生命科学研究发展的典型范例。
1.1 蛋白质结构解析范例蛋白质作为生物体内关键功能的执行者,其结构直接影响运输、催化、结合和免疫功能等重要的生物过程。虽然测序技术可以揭示蛋白质所包含的氨基酸序列,但任何一个已知氨基酸序列的蛋白质链有可能折叠成天文数字中的任何一种可能构象,这使得准确解析蛋白质结构成为长期以来的挑战。利用传统技术如核磁共振、X射线晶体分析、冷冻电子显微镜等解析已知序列的蛋白质结构方法,需要数年时间才能描绘出单个蛋白质的形状,昂贵耗时且不能保证成功解析其结构。因此,捕获蛋白质折叠的底层规律从而实现对蛋白质结构的精准预测,一直是结构生物学领域最重要的挑战之一。
AlphaFold 2利用基于注意力机制的深度学习算法,对大量蛋白质序列和结构数据进行训练,并结合物理学、化学和生物学的先验知识,构建了包含特征提取、编码、解码模块的蛋白质结构解析模型[3]。在2020年国际蛋白质结构预测竞赛(CASP14)中,AlphaFold 2取得了瞩目的成绩,其蛋白质三维结构预测准确性甚至可与实验解析的结果相媲美。这一突破为生命科学领域带来了全新的视角和前所未有的机遇,主要体现在3点。
(1)对药物发现领域产生了直接影响。大多数药物通过与体内蛋白质特殊结构域的结合而引发蛋白质功能的变化,AlphaFold 2能够快速计算出海量目标蛋白质的结构,从而有针对性地设计药物以有效地与这些蛋白质结合[4]。
(2)对蛋白质的理性设计提供了新的可能性。一旦AI对蛋白质折叠的底层规律有了深刻理解,就可以利用这一知识设计出折叠成所需结构的蛋白质序列。这使得生物学家可以根据需求自由设计和改造蛋白质或酶的结构,如设计更高活性的基因编辑酶[5],甚至是自然界中不存在的蛋白质结构[6]。同时也推动了人们对基因编码信息在蛋白质层面结构投射规律的理解,并将大幅提高人类对生命的改造能力。
(3)AlphaFold 2彻底改变蛋白质结构解析领域的研究范式。从只能通过费时费力的传统实验技术解析蛋白质结构转变为低门槛、高精度、高通量地预测蛋白质三维结构的新范式,证明通过将蛋白质知识和AI技术相结合,可以提取和学习到高维、复杂的知识,促进对蛋白质物理结构和功能的更深入理解。
1.2 基因调控规律解析范例人类基因组计划被誉为20世纪人类三大科学计划之一,揭开了生命奥秘的序幕。尽管编码生命个体的遗传信息存储在DNA序列中,但每个细胞的命运和表型却因其独特的时空背景而千差万别。这种复杂的生命过程由精细的基因表达调控系统所控制,而探索生命普遍存在的基因调控机制是继人类基因组计划之后最为重要的生命科学问题之一。不同细胞的基因表达谱是理解生物系统内基因调控活动的理想窗口。然而,仅通过生物学实验全面解读基因调控机制,需要捕获不同生物个体的不同细胞类型在不同环境背景下的对照试验来观察。传统生物信息分析方法只能处理少量数据,对大规模、高维度且缺乏准确标注的生物组大数据难以捕捉数据中复杂的非线性关系。
近年来,自然语言处理技术的不断突破,特别是大语言模型的迅猛发展,能够通过训练语料数据使模型具有理解人类语言描述知识的能力,为解决这一领域问题带来了新思路。国际多个研究团队借鉴大语言模型的训练思路,相继基于数以千万计的人类单细胞转录组谱数据和庞大的算力资源,利用Transformer等先进算法和多种生物学知识,构建了多个具有理解基因动态关系能力的生命基础大模型,如GeneCompass[7]、scGPT[8]、Geneformer[9]和scFoundation[10]等。这些生命基础大模型以基因表达等底层生命活动信息为训练基础,利用机器来学习理解这些“低维”的生命科学数据与复杂“高维”的基因表达调控网络、细胞命运转变等底层生命机制之间的关联性和对应规律,实现以低维数据对高维信息的有效模拟和预测。这种对基因表达调控网络的模拟可以在广泛的下游任务中表现出卓越性能,为深入理解基因调控规律提供了全新的途径。
现有的AI驱动生命科学研究的成功案例向我们证明,面对更深入、更系统的生命科学问题,AI有望突破传统研究方法难以解决的困境、构建从基础生物层次到整个生命系统的投射理论体系,并进一步推动生命科学向更高阶段发展,开启生命科学研究的新范式。
2 生命科学研究新范式的内涵和关键要素随着生物技术的不断进步、生命科学数据的快速增长、AI技术的飞速发展及其与生命领域的深度交叉融合,AI展示出了对生命科学知识的深入理解和泛化能力,不仅提高了生命科学的研究高度和广度,也促使生命科学研究由实验科学为主的第一范式,跨越进入AI驱动的生命科学研究新范式(第五范式,以下简称“新范式”)。
通过深入剖析AI驱动生命科学研究的典型范例,笔者认为,生命科学研究的新范式正如一台智能化的新能源汽车,对标新能源汽车的电池系统、电控系统、电机系统、辅助驾驶系统、底盘系统等核心技术,新范式应具备生命科学大数据、智能算法模型、算力平台、专家先验知识和交叉研究团队五大关键要素(图 2)。犹如电池系统为车辆提供能量,生命科学大数据为科学研究提供基础资源;算法模型则像智能电控系统,赋能深入理解生物系统的运行机制;算力平台可比喻为电机系统,负责处理海量的科学数据和复杂的计算任务;专家先验知识则像辅助驾驶系统,为科学家提供方向引领和实施经验;交叉研究团队类似于底盘系统,负责整合不同领域的知识和技能,通过跨学科合作提高研究效率,推动生命科学的发展。
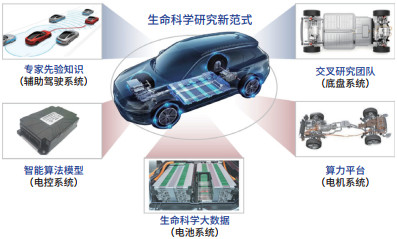
|
| 图 2 生命科学研究新范式的五大关键要素 Figure 2 Five key elements of new paradigm in life sciences research |
生命科学大数据是新范式“汽车”的“电池”系统。随着新型生物技术的发展,具有多模态、多维度、分布分散、关联隐匿、多层次交汇等特点的生命科学大数据逐渐形成;只有对生命科学大数据进行有效整合并利用创新AI技术充分挖掘数据,才能够打破人类科学家的认知局限、促进新发现的产生并拓展生命科学的探索范围。例如医疗视觉大模型[11-13],通过整合多来源、多模态、多任务的医疗图像数据,实现了在少样本和零样本条件下的多种应用;跨物种生命基础大模型GeneCompass[7],通过有效整合全球开源的单细胞数据,在超过1.2亿个单细胞的训练数据集上实现了对基因表达调控规律的全景式学习理解等多个生命科学问题的分析。
2.2 关键要素二:智能算法模型智能算法模型是新范式“汽车”的“电控”系统。从浩如烟海的生命科学大数据中涌现生命的新规律和新知识,需要创新AI算法和模型;如何研发利用生命科学适配的AI算法、提取有效的生物特征、构建大规模生物过程动态模型,是当前新范式的中心问题[14]。例如,Gerstein团队[15]使用贝叶斯网络算法预测蛋白质相互作用的成果发表于Science,为经典机器学习在生物信息领域发展奠定了基础;图卷积神经网络算法被用于分析蛋白质—蛋白质相互作用网络[16]和基因调控网络[17]等生物分子网络,拓展了生命科学领域的研究方向;AlphaFold 2[4]使用Transformer模型,能够在高准确度的基础上快速计算出大量蛋白质的结构,都展示出了AI算法模型在生命科学研究新范式中的重要性。
2.3 关键要素三:算力平台算力平台是新范式“汽车”的“电机”系统。算力是实现AI运行的基础,深度学习、大模型技术等适用于生命科学研究新范式的AI算法模型的不断发展,使AI模型训练需要更强大、更高效的算力平台支持。面向新范式,未来应构建能够支撑AI赋能生命科学研究的硬件能力平台,包括建设高速大容量存储系统、构建高性能高吞吐量超级计算机、研发专门用于处理生命科学数据的芯片、设计用于加速生物模型推理和训练的专用处理器等,为生命科学研究提供高效、可靠的计算和处理能力,以应对生命科学领域产生的海量数据、满足生命科学领域复杂模型构建的计算需求,保障AI在生命科学领域的应用和创新。
2.4 关键要素四:专家先验知识专家先验知识是新范式“汽车”的“辅助驾驶”系统。新范式下,已有的生命科学知识将为AI算法模型提供宝贵的训练约束条件、重要的背景和特征关系,帮助解释和理解生命科学数据的复杂性、验证和优化AI在生命科学领域的应用;能够在AI算法设计和模型构建时发挥重要的指导作用,促进更加准确、高效地解决生命科学问题,推动生命科学研究向更深入、全面的方向发展。例如,通过嵌入生命科学专家先验知识和人类注释信息编码,新型基因表达预训练大模型[7]提高了对生物数据间复杂特征关联关系的解释,展示出更为优异的模型表现。
2.5 关键要素五:交叉研究团队交叉研究团队是新范式“汽车”的“底盘”系统。新范式下,一支由AI专家、数据科学家、生物学家和医学家等组成的多学科交叉研究团队对于实现跨越式的生命科学发现至关重要。多元背景紧密协作的交叉研究团队能够整合AI、生物学、医学等领域的专业知识,提供多元化的视角和方法,为全面理解和解决生命科学中的复杂机制问题提供牢固基础,为创新性解决方案提供更多可能性,从而推动生命科学领域的突破性发现和进展。
3 新范式赋能的生命科学研究前沿及我国面临的挑战传统的研究范式对生命的探索如同管中窥豹,生物学家在生命科学的不同细分领域各自奋战。随着新范式的不断发展,生命科学研究将迎来以AI预测、指导、提出假说、验证假设为特点的新型研究模态,迸发出一批快速发展的生命科学新范式前沿研究方向,并展现出新范式变革带来的发展增益。然而,在当前条件下加速推进我国生命科学研究新范式的建立和推广,仍面临一系列巨大的挑战。
3.1 新范式赋能的生命科学研究前沿(1)结构生物学。目前在结构生物学领域,以AlphaFold为代表的AI应用技术仍停留在“从序列到结构”的蛋白质结构预测和设计阶段[6, 18, 19],还无法实现复杂生理条件下蛋白质结构和功能的模拟与预测。更高质量、更大规模的蛋白质数据和新型算法的出现,将有望对不同生理状态和时空条件下的生物大分子结构和功能进行系统解析,并实现蛋白质“从序列到功能”甚至“从序列到多尺度相互作用”的智能化结构解析与精细设计。
(2)系统生物学。当前的组学数据分析仍局限于较低维度的生物组学观测水平,还未形成从基因水平到细胞水平甚至生物个体乃至群体组学水平的全维度观测。新范式将融通多维度、多模态的生物大数据和专家先验知识,提取生物表型的关键特征,构建多尺度生物过程解析模型,还原复杂生物系统运行的底层规律,形成基础而广泛适用的系统生物学研究新体系。
(3)遗传学。随着多组学数据的积累和新型基因大模型的出现,遗传学研究已进入新范式推动的快速发展阶段,基于基因表达谱数据的自监督预训练大模型有望成为解析基因调控规律、预测疾病靶点的有力工具[9],拓展遗传学研究的探索边界。
(4)药物设计开发。随着AlphaFold的出现和一批分子动力学模型的发展,AI模型已经被用于预测和筛选药物候选分子。未来新范式将进一步推动该领域的发展,有望出现AI辅助的全流程药物设计开发体系,能够自主完成药物结构和性质的优化设计、实现候选药物的有效性和安全性模拟预测、生成药物的高效合成和生产工艺方案,极大加速药物的开发和生产过程。
(5)精准医学。计算机视觉、自然语言处理和机器学习等AI技术已广泛渗透到生物影像、医学影像、疾病智能分析及靶点预测等精准医学子领域。例如,基于AI的诊断系统在准确度上已经可以媲美甚至在某些方面超过资深的临床医生[20]。然而,现有的模型大多受制于数据的偏好性,存在鲁棒性差、通用性低等问题,随着新范式驱动的通用精准医学模型的出现,将有助于更加快速准确地诊断疾病、解析疾病的分子机制、发现新的治疗靶点,提高人类的健康水平。
3.2 我国生命科学研究新范式面临的挑战面对生命科学研究新范式发展的新形势、新要求,我国仍面临高质量生命科学数据资源体系缺乏、AI关键技术与基础设施不足、新范式下的交叉创新科研新生态匮乏等方面的巨大挑战。
3.2.1 高质量生命科学数据资源体系缺乏尽管我国在生命领域的科研投入持续增加,但在一些前沿领域,我国科学家仍依赖国外高质量数据,而国内数据的建设和使用相对滞后,我国生命科学数据资源还存在分布不均衡问题,需要更好地统筹协调和资源整合,实现高质量生命科学数据资源的高效汇聚和系统化提升。此外,在生命科学数据的收集、传输和存储过程中,数据安全问题亟待加强,特别是生物数据的隐私和安全问题仍需要引起重视。
面对这些挑战,我国需要加强科学数据资源的整合与共享,推动生命科学数据资源的可持续发展,提高数据的质量和安全性,加强数据管理与供给模式的变革,推动跨领域多模态科技资源融合服务能力的提升,以满足新范式下科研需求的发展。
3.2.2 AI关键技术与基础设施不足我国AI驱动新科研范式的核心技术相对匮乏,自主原创的算法、模型、工具仍待大力发展。针对生命科学大数据的海量、高维、稀疏分布等特征,亟需发展复杂数据的先进计算与分析方法。未来应开发更加适合生命科学应用的硬件、软件和新计算介质,并在生命科学和计算科学的融合过程中,探索新的计算-生物交互模式。简而言之,新范式研究对数据、网络、算力等资源的综合能力提出了新的要求,需要加快推进新一代信息基础设施建设,解决算力“卡脖子”问题。
3.2.3 新范式下的交叉创新科研新生态匮乏现有AI驱动的生命科学研究方式大多为课题组自发组合的“小作坊”模式,缺乏新范式发展所需的交叉创新环境。美国在2023年发布的《国家人工智能研发战略计划》更新版本中也着重强调了人工智能研究的跨学科交叉发展的重要性。因此,新范式下的科研生态应鼓励更为广泛的多学科“大交叉”“大融合”,建立干湿结合、理实交融的新型研究模式,持续培养高水平复合型交叉研究人才。
在新形势下我国也已经开始广泛布局和推进交叉学科的发展。《中华人民共和国国民经济和社会发展第十四个五年规划和2035年远景目标纲要》中指出要推动互联网、大数据、人工智能等同各产业的深度融合。结合我国生命科学领域的实际发展情况,我国生命科学领域发展更应着眼于将AI赋能生命科学研究的范式变革融入我国新时代的国家发展远景布局中,实现以点带面的整体效应建立更加开放的新型科研生态和发展环境。
4 结语近年来,生命科学领域正经历着前所未有的巨变,这一领域的发展不仅受到生物技术和信息技术的双重推动,更受到AI技术进步的巨大影响。这一变革的核心在于从传统的主要依赖于人经验的假说和实验驱动的科研范式向大数据和AI驱动的新研究范式的演变。这意味着我们不再仅仅依赖于实验和假说,而是通过大数据分析和AI技术主动揭示生命的奥秘。更广泛的,这个演变将广泛改变或促进不同层面的科学研究活动的变革,涵盖了认识论、方法论、研究组织形式、经济社会及伦理法律等众多层面。
综合而言,我们正身临着一个充满变革和希望的时代,生命科学的革新与科技的进步共同绘制出人类对生命奥秘更深层次探索的未来蓝图。可以预见,随着通用AI的进一步发展,生命科学研究将在不远的未来实现干湿融合、人机协同的新模式,迎来AI自驱抽象新知识、新规律的“预人所未见,思人所未思”的科学新时代。
| [1] |
Wang H, Fu T, Du Y, et al. Scientific discovery in the age of artificial intelligence. Nature, 2023, 620: 47-60. DOI:10.1038/s41586-023-06221-2 |
| [2] |
Erbe R, Gore J, Gemmill K, et al. The use of machine learning to discover regulatory networks controlling biological systems. Molecular Cell, 2022, 82(2): 260-273. DOI:10.1016/j.molcel.2021.12.011 |
| [3] |
Jumper J, Evans R, Pritzel A, et al. Highly accurate protein structure prediction with AlphaFold. Nature, 2021, 596: 583-589. DOI:10.1038/s41586-021-03819-2 |
| [4] |
Borkakoti N, Thornton J M. AlphaFold2 protein structure prediction: Implications for drug discovery. Current Opinion in Structural Biology, 2023, 78: 102526. DOI:10.1016/j.sbi.2022.102526 |
| [5] |
Huang J Y, Lin Q P, Fei H Y, et al. Discovery of deaminase functions by structure-based protein clustering. Cell, 2023, 186(15): 3182-3195. e14. DOI:10.1016/j.cell.2023.05.041 |
| [6] |
Madani A, Krause B, Greene E R, et al. Large language models generate functional protein sequences across diverse families. Nature Biotechnology, 2023, 41(8): 1099-1106. DOI:10.1038/s41587-022-01618-2 |
| [7] |
Yang X D, Liu G L, Feng G H, et al. GeneCompass: Deciphering universal gene regulatory mechanisms with knowledge-informed cross-species foundation model. (2023- 09-28). https://www.biorxiv.org/content/10.1101/2023.09.26.559542v1.
|
| [8] |
Cui H T, Wang C, Maan H, et al. scGPT: Towards building a foundation model for single-cell multi-omics using generative AI. (2023-05-01). https://www.biorxiv.org/content/10.1101/2023.04.30.538439v1.
|
| [9] |
Theodoris C V, Xiao L, Chopra A, et al. Transfer learning enables predictions in network biology. Nature, 2023, 618: 616-624. DOI:10.1038/s41586-023-06139-9 |
| [10] |
Hao M S, Gong J, Zeng X, et al. Large scale foundation model on single-cell transcriptomics. (2023-05-31). https://www.biorxiv.org/content/10.1101/2023.05.29.542705v1.
|
| [11] |
Singhal K, Azizi S, Tu T, et al. Large language models encode clinical knowledge. Nature, 2023, 620: 172-180. DOI:10.1038/s41586-023-06291-2 |
| [12] |
Moor M, Banerjee O, Abad Z S H, et al. Foundation models for generalist medical artificial intelligence. Nature, 2023, 616: 259-265. DOI:10.1038/s41586-023-05881-4 |
| [13] |
Li C Y, Wong C, Zhang S, et al. LLaVA-med: Training a large language-and-vision assistant for biomedicine in one day. (2023-06-02). https://arxiv.org/abs/2306.00890.
|
| [14] |
Alber M, Buganza Tepole A, Cannon W R, et al. Integrating machine learning and multiscale modeling-perspectives, challenges, and opportunities in the biological, biomedical, and behavioral sciences. NPJ Digital Medicine, 2019, 2: 115. DOI:10.1038/s41746-019-0193-y |
| [15] |
Jansen R, Yu H Y, Greenbaum D, et al. A Bayesian networks approach for predicting protein-protein interactions from genomic data. Science, 2003, 302: 449-453. DOI:10.1126/science.1087361 |
| [16] |
Wu Y F, Gao M, Zeng M, et al. BridgeDPI: A novel Graph Neural Network for predicting drug-protein interactions. Bioinformatics, 2022, 38(9): 2571-2578. DOI:10.1093/bioinformatics/btac155 |
| [17] |
Gan Y L, Hu X, Zou G B, et al. Inferring gene regulatory networks from single-cell transcriptomic data using bidirectional RNN. Frontiers in Oncology, 2022, 12: 899825. DOI:10.3389/fonc.2022.899825 |
| [18] |
Lutz I D, Wang S Z, Norn C, et al. Top-down design of protein architectures with reinforcement learning. Science, 2023, 380: 266-273. DOI:10.1126/science.adf6591 |
| [19] |
Watson J L, Juergens D, Bennett N R, et al. De novo design of protein structure and function with RFdiffusion. Nature, 2023, 620: 1089-1100. DOI:10.1038/s41586-023-06415-8 |
| [20] |
Kermany D S, Goldbaum, Cai, W.M. & Leveraging big data and AI in medical diagnosis. [2023-12-30]. https://www.nature.com/articles/d42473-022-00035-y.
|




