2. 工业生物催化教育部重点实验室 北京 100084;
3. 洛阳华清天木生物科技有限公司 洛阳 471023;
4. 北京联合大学 生物化学工程学院 生物质废弃物资源化利用北京市重点实验室 北京 100023;
5. 清华大学 合成与系统生物学中心 北京 100084;
6. 清华大学 深圳国际研究生院 生物医药与健康工程研究院 深圳 518055
2. Key Laboratory of Industrial Biocatalysts, Ministry of Education, Beijing 100084, China;
3. TMAXTREE Biotechnology Co. Ltd., Luoyang 471023, China;
4. Beijing Key Laboratory of Biomass Waste Resource Utilization, College of Biochemical Engineering, Beijing Union University, Beijing 100023, China;
5. Center for Synthetic and Systems Biology, Tsinghua University, Beijing 100084, China;
6. Institute of Biopharmaceutical and Health Engineering, Tsinghua Shenzhen International Graduate School, Shenzhen 518055, China
工程细胞是绿色生物制造的“芯片”,在医药、化学品、材料、燃料等各类物质的生物加工过程中充当核心执行者角色[1]。目前,工程细胞的构建往往依托设计—构建—测试—学习(DBTL)循环策略[2],首先基于先验知识和计算模型设计生物合成路径,利用基因合成、组装和编辑等技术进行工程细胞的构建,进而对所构建工程细胞进行测试,如基因型测试,以及包括细胞生长、目标产物产量和质量在内的表型测试,最后对测试结果进行综合评估分析,用于进一步优化设计,提高工程细胞工作效率[3]。由于生命系统的复杂性,人们对代谢网络和多层次调控机制认知有限,往往需要构建海量基因型进行大规模表型测试,才能获得性能优越的工程细胞底盘。因此,在DBTL循环中,工程细胞的高通量表型测试是最为关键的环节之一。
仪器设备是实现工程细胞高通量表型测试的基础。纵观工程细胞表型测试技术与装备发展历程,经历了平板、微孔板、自动化工作站和微流控4个阶段。19世纪80年代,为了解决试管或烧瓶中单克隆难以观察和操作的问题,德国微生物学家Julius Richard Petri发明了Petri平板培养皿[4],开启了平板测试时代,这种用于单克隆分离培养的平板技术沿用至今。随着测试通量需求的提高,20世纪50年代,德国微生物学家Gyula Takatsy发明了微孔板测试方法,集成单克隆培养与检测,通量一般为103/天—104/天[5, 6]。由于微孔板操作耗时耗力,20世纪80年代,自动化工作站时代到来,并在后期逐渐形成集成克隆挑取、孔板培养、检测、筛选自动化操作模块于一体的集成平台,每天实现104—105样品高通量测试[7]。20世纪90年代,Manz等[8]首次提到微流控这一词汇,定义为一种在微纳米尺度空间中精确控制和操纵微纳米流体的科学技术。21世纪初,微流控技术迎来迅猛发展,由于样品操作体积小、检测参数多样(如荧光、散射光、吸光度、拉曼)、检测通量高(每天测试样品最高达到108—109)[9]、成本低(试剂消耗比微孔板降低可达106倍)等巨大优势[10],微流控装备成为工程细胞高通量表型测试研究热点。面向合成生物学单细胞分析与高通量筛选等表型测试需求,近年来发展了非培养类型的单细胞测试[11]、培养类型的液滴微流控测试[12]与微腔室测试技术与装备[13],为合成生物学的发展提供了重要的装备支撑。总的来说,微流控技术的应用,代表了工程细胞表型测试技术与装备高通量、自动化、微型化、集成化、多参数的发展趋势。本文将重点阐述基于微流控技术的非培养类型与培养类型工程细胞高通量表型测试技术与装备研究进展,并展望其发展方向,为面向绿色生物制造的工程细胞表型测试提供借鉴。
1 单细胞高通量表型测试技术与装备单细胞表型测试技术是指基于单细胞自身特性如光学性质、胞内代谢产物、形状特征、毒性耐受、电学性质等的检测与分选技术。通过散射光与荧光[14, 15]、质谱信号[16]、拉曼光谱[17]、显微成像[18]、磁信号[19]等技术识别目标细胞信息后,利用电场、磁场、光场、声场、流体力场、重力场等方法驱动细胞向收集处运动,最终分选出目标单细胞[20]。下文对4类典型的单细胞表型测试技术与装备进行总结。
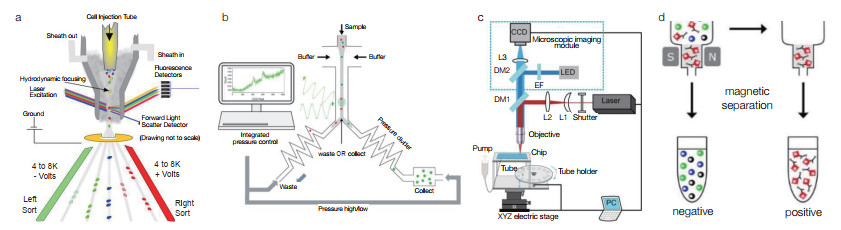
1.1 荧光激活细胞分选技术与装备荧光激活细胞分选(fluorescence activated cell sorting,FACS)是对荧光标记的单细胞进行高速、多参数定量分析和分选的技术(图 1a),由控制细胞流动的流体系统、光学系统、捕获发射荧光和散射信号的电子系统和数据采集系统组成。其原理是利用激光作为光源照射单细胞产生散射光和荧光信号,并将这些光学信号通过检测器读取、转换为电子信号输出,从而对单个细胞进行快速分析和筛选。
|
| 图 1 单细胞高通量表型测试装备技术原理 Figure 1 Principle of single-cell high-throughput phenotype testing equipment (a)荧光激活细胞分选装备技术原理;(b)拉曼激活细胞分选装备技术原理;(c)图像激活细胞分选装备技术原理;(d)磁激活细胞分选装备技术原理 (a) Principle of fluorescence activated cell sorting equipment; (b) Principle of Raman activated cell sorting equipment; (c) Principle of image activated cell sorting equipment; (d) Principle of magnetically activated cell sorting equipment |
FACS技术用于荧光标记的单细胞高通量测试,每天测试通量可达到108以上。近年来,基于荧光探针[21]、细胞表面展示[22]、生物传感器[23]等荧光标记技术,FACS在蛋白质工程和工业菌株育种领域取得显著进展,例如纤维素酶[24]等定向进化,高产L-半胱氨酸大肠杆菌[25]、高产L-赖氨酸谷氨酸棒状杆菌[26]等典型工业菌株的高通量选育。然而,FACS单细胞表型测试技术受限于荧光标签的开发,以及胞内与胞膜物质的测试,同时流式细胞仪细胞分选前的高压充电过程和分选过程中的高速喷射过程均对细胞产生一定损伤,致使活力下降。为了避免这些问题,研究者开发了双乳化水包油包水液滴(W/O/W)[27]、凝胶微球(gel-droplet)[28]等技术,将单细胞包裹进水相液滴或水相微球中进行后续培养和FACS筛选。然而,这些方法由于步骤烦琐、液滴易破损未得以广泛应用。
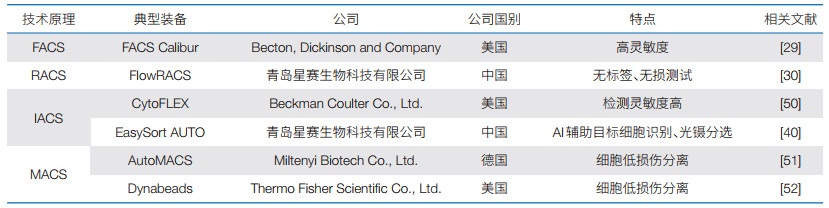
FACS技术装备化方面,近年来,我国上海纬冉科技有限公司自主研发的SE420流式细胞分选仪实现了细胞样本的全面分析与高通量分选,成都赛雷纳医疗科技有限公司研发的小型Sparrow流式细胞仪以及深圳迈瑞生物医疗电子股份有限公司的BriCyte E6流式细胞仪,目前一般用于单细胞的分析与检测。在进口品牌方面,美国BD公司的FACS Calibur、FACS Melody、FACS Jazz、FACS Aria系列[29],美国Beckman Coulter公司的CytoFlex SRT、EPIC XL系列,日本On-chip Biotechnologies公司的On-chip Sort细胞分选仪均可进行多参数、高分辨率和灵敏度细胞分析与分选。可见我国FACS整体技术水平与国外仍有差距,在市场认可度、仪器检测精度、灵敏度、稳定性及多参数检测能力等方面有待提高。因此,需不断加强基础研究和技术创新,加大对关键零部件研发的投入,提高仪器的核心性能和自主可控性,加速技术转化和人才培养,提升我国在流式细胞术领域的整体技术水平。
1.2 拉曼激活细胞分选技术与装备拉曼激活细胞分选(Raman activated cell sorting,RACS)是基于拉曼光谱检测的单细胞分析和分选技术(图 1b)。拉曼光谱是一种散射光谱,每个散射峰都对应于一种特定的分子键振动,因此可以识别单细胞内部的全景信息,允许对单个细胞进行无损、无标记的化学分析,并根据其分子组成进行物理分选,被视为一种快速、低成本的单细胞表型测试技术[30]。根据分选时单细胞的运动状态,RACS测试分为静态细胞分析与捕获、流动细胞分析与捕获2种类型。前者是指在细胞静止或相对静止的状态下,基于拉曼光谱信息分选出特定类型细胞至单管中,如拉曼弹射分选(Raman-activated cell ejection,RACE)[31]、重力驱动拉曼光镊液滴分选(Raman-activated gravity-driven encapsulation,RAGE)[32]等技术,其优势为可以对接下游的单细胞培养、单细胞测序等研究,然而静态单点捕获通量过低。后者是指细胞悬浮在流动相中,在流动状态下对单细胞进行拉曼光谱检测,分选收集优势表型细胞,如拉曼微液滴分选(Raman-activated droplet sorting,RADS)[33]、介电捕获拉曼激活液滴分选(positive dielectrophoresis-based RADS,pDEPRADS)[34]等技术,单细胞在拉曼检测后随着流动相流动,通过油相剪切形成单细胞液滴进而分选至收集管,其优势为高通量,更适用于文库中目标表型细胞的测试。
RACS静态单细胞测试技术主要应用于单细胞组学研究,Song等[17]利用该技术从海水样品中分离出富含类胡萝卜素单细胞,并在分离后对单细胞进行测序,发现了新型类胡萝卜素合成基因;Su等[35]通过对分离的单细胞全基因组扩增测序,实现了95%的基因组覆盖度。而RACS流动单细胞测试技术主要应用于单细胞底物代谢、产物合成和细胞的分析鉴定研究,通量每天可达到104以上。在细胞代谢测试中,通过使用 13C、15N和 2H等同位素标记底物从而改变分子质量,细胞摄入底物后,拉曼光谱发生变化,实现细胞代谢的分析研究。例如,Kumar等[36]将 13C标记的糖类物质等添加到底盘细胞培养基中,通过分析蛋白中13C拉曼光谱位移变化,揭示了细胞对碳源底物代谢的抑制机制。在胞内产物合成测试中,拉曼光谱可以在无损且非标记的状态下同步检测不同代谢产物,如色素[37]、淀粉[38]等物质,为高产菌株的高通量筛选和定量分析提供新思路。此外,由于每种单细胞拉曼光谱具有特异性,可作为单细胞特有的“分子指纹图”,进而反映出特定细胞内化学物质成分及含量的多维信息。因此,RACS还被用于单细胞分析鉴定,如Yan等[39]结合机器学习算法和拉曼光谱,在单细胞水平上鉴定出食源性病原体。
我国拉曼光谱单细胞表型测试装备处于国际领跑地位。青岛星赛生物科技有限公司率先开发了全球首台高通量流式拉曼分选仪FlowRACS,该装备能够直接鉴定单细胞种类并测试代谢相关表型。吉林长光辰英科技有限公司开发了PRECI SCS-R300拉曼单细胞分选仪,实现了单细胞识别与分离研究。
1.3 图像激活细胞分选技术与装备图像激活细胞分选(image activated cell sorting,IACS)是一种基于显微成像的细胞分选技术(图 1c)。IACS技术的核心在于利用高分辨率显微成像系统捕获细胞的图像,然后通过图像分析软件识别和分类细胞[40]。这些图像可以提供细胞的大小、形状、纹理等信息,常用于特定细胞的高通量分离实验。例如,Nitta等[41]将三维成像技术和薄膜微阀流体驱动技术结合,获取高质量的细胞三维图像,并通过薄膜阀驱动目标细胞至收集管路中,以此完成细胞的图像分析和分选。Akihiro等[42]基于IACS技术,集成了高通量的光学显微镜、细胞聚焦、细胞排序和深度学习算法,开发了iIACS系统,实现了数据采集、处理、智能决策和执行的自动化操作。Zhao等[43]将iIACS系统与人工智能(AI)图像处理结合,进一步提高了基于图像的单细胞分选通量。
基于IACS技术研发的装备包括美国BD公司的ImageStream X MkII系统[44]、美国Amnis Corporation公司的ImageStream系统[45]、美国Beckman Coulter公司的CytoFLEX系列产品,实现了分选前的细胞图像信息采集。我国青岛星赛生物科技有限公司开发了EasySort AUTO系统,基于显微成像与AI图像分析技术,在该系统中,AI辅助目标检测模型实现了对目标细胞的高精度识别,系统集成的光镊模块能够将细胞自动转移到收集管中[40]。目前,我国在IACS领域的研究发展迅速,但由于起步较晚,还处于基础技术的开发和优化阶段。因此,需要加强基础研究、促进跨学科合作与国际合作交流,以逐步缩小我国IACS装备与国际先进水平的差距。
1.4 磁激活细胞分选技术与装备磁激活细胞分选技术(magnetic activated cell sorting,MACS)是一种基于磁场和磁性标记的细胞分离技术(图 1d),其核心在于使用超顺磁性微珠标记特异性抗体[46],这些抗体能够识别并结合目标细胞表面的特定抗原,一旦标记完成,细胞混合物被引入磁场中,磁性微珠会被迅速吸附到磁场的一侧,从而将标记的细胞与未标记的细胞分离,其通量为每天109样品[47, 48]。MACS分离方式快速、高效,且对细胞的损伤小,适合于后续的细胞培养、分子分析,常用于动物细胞的分离。Munz等[49]利用MACS技术成功分离小鼠脾细胞中的树突状细胞(DCs),并研究了其在免疫应答中的作用。然而,该技术面临特异性抗体标记的问题,难以实现细胞的普适性测试。在装备研究中,德国Miltenyi Biotec公司的AutoMACS、美国Thermo Fisher Scientific公司的Dynabeads,均成功实现了磁激活细胞分选设备商业化。此外,美国BD公司将MACS与FACS技术结合,开发了FACSAria Ⅲ产品,为用户提供了更多的选择。可见国内MACS装备产业化程度相对较低,缺乏具有国际竞争力的品牌,因此需要投入更多资源进行MACS技术的基础研究,以提升我国MACS技术创新能力。
基于FACS[29]、RACS[30]、IACS[40, 50]、MACS[51, 52]技术原理开发的非培养类型单细胞高通量表型测试典型商业化装备如表 1所示。
|
液滴微流控技术(droplet-based microfluidics)是一种在微纳米尺度上操控和处理微液滴的技术,通过在微通道内操控互不相溶的多相流体,基于微流控芯片实现皮升(pL)至微升(μL)尺度液滴的单元操作,包括液滴的生成、注入、分裂、融合、信号检测和分选等[53, 54]。与单细胞测试工具相比,液滴可以作为独立的反应单元培养单细胞,并进行后续胞内、胞膜、胞外、无细胞体系相关物质的高通量检测与分选,具有体积小、单分散性好、无交叉污染等优点。典型模式菌株如大肠杆菌、酵母菌等直径在10微米以下,100皮升以内的液滴即可满足培养需求;而动物细胞、放线菌等直径在10微米以上,需要将液滴体积增加至几百皮升乃至纳升级别才可以进行培养,丝状真菌菌丝密集坚硬[55],在皮纳升液滴中培养易造成液滴之间的融合,通常需要微升液滴体系才可长期培养。可见,不同表型测试场景下的液滴微反应器尺度需求不同,下文将分别阐述皮纳升液滴与微升级液滴测试技术与装备。
2.1 皮纳升液滴培养技术与测试装备皮纳升液滴是指体积范围在1皮升—100纳升的液滴,一般以油相作为连续相,水相作为分散相,当两相流体经过毛细管共轴聚焦、微流控芯片流动聚焦等结构时,油相剪切水相形成均匀的单分散液滴。通过泊松分布理论,单细胞被包裹在液滴中进行生长代谢,随后基于不同的分选技术,如荧光激活液滴分选(FADS)、吸光度激活液滴分选(AADS)、质谱激活液滴分选(MADS)、成像激活液滴分选(IADS)实现目标表型细胞的分选和收集。
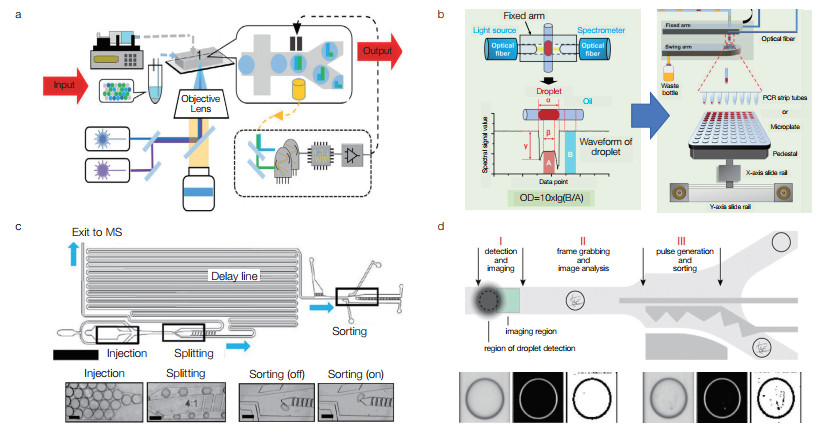
FADS技术是目前使用最广泛的皮纳升液滴筛选技术(图 2a),最早于2009年被提出[56],经过10余年的发展,技术不断迭代升级,已经形成较为成熟的商业化装备。FADS技术由驱动系统、成像系统、光学系统、电学系统、微流控芯片系统等组成[57],通过微泵驱动液滴运动,激光器激发液滴荧光后,光学系统将光信号转化为电学信号输出;当信号位于设定的阈值时,通过介电泳等方式将液滴分选至芯片收集通道[58]。该技术中的关键挑战在于开发荧光探针,实现荧光信号与细胞表型的耦合。针对细胞表达的生物酶活性测试,开发了荧光基团修饰底物检测体系[59, 60];针对小分子代谢物,开发了酶联荧光探针传感器[61, 62]、全细胞类[63]与拟荧光蛋白类生物传感器[64],极大拓展了FADS技术在合成生物制造领域的应用。
|
| 图 2 微液滴高通量表型测试装备技术原理 Figure 2 Principle of microdroplets high-throughput phenotype testing equipment (a)荧光激活液滴分选装备技术原理;(b)吸光度激活液滴分选装备技术原理;(c)质谱激活液滴分选装备技术原理;(d)图像激活液滴分选装备技术原理 (a) Principle of fluorescence activated droplet sorting equipment; (b) Principle of absorbance activated droplet sorting equipment; (c) Principle of mass activated droplet sorting equipment; (d) Principle of image activated droplet sorting equipment |
由于FADS技术需要开发相应的荧光检测体系,在具体使用场景受到一定限制,近年来还发展了AADS、MADS、IADS等无标签检测分选技术。AADS技术是基于吸收光谱法的微液滴检测技术(图 2b),Gielen等[65]通过在液滴检测口两侧,芯片上内置两根光纤,分别连接光源和检测器,液滴流过时引起光谱吸收变化输出信号,根据光吸收变化对感兴趣的目标液滴进行分选。该装置用于苯丙氨酸脱氢酶的定向进化,酶活性增加了2.7倍。然而,由于皮纳升体积液滴反应器检测光程过短、检出信号困难,AADS技术仍处于底层技术研究阶段。MADS技术是将微流控芯片通过接口连接ESI电离喷雾质谱(图 2c),在微流控芯片上完成液滴的分裂,一部分液滴通过接口进入质谱进行破坏性检测,另一部分液滴备份。当质谱输出符合预期的信号时,基于介电泳将备份的液滴分选至芯片收集通道,该装置用于含有体外表达转氨酶的液滴筛选,实现了0.7个/秒的液滴筛选速率,准确率为98%[66]。IADS技术是一种基于液滴图像识别、处理与分析的无标记分选技术(图 2d),首先将细胞细胞悬浮液与试剂混合,进行单个细胞的封装,在微环境中培养后通过显微成像、荧光成像技术测试培养后的细胞群。Zang等[67]通过对液滴成像,检测液滴内放线菌的生长量,实现了100个/秒目标液滴的分选。
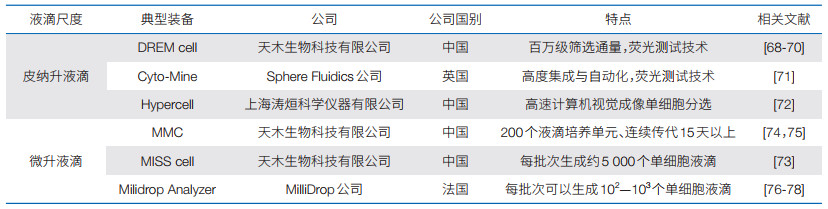
国内外报道了众多基于FADS技术的商业化皮升纳液滴装备。我国洛阳华清天木生物科技有限公司开发了商业化的高通量皮升级液滴单细胞分选系统DREM cell,实现了每天超百万液滴的筛选通量[64]。Ma等[57]基于该设备将酯酶对映体选择性提高了700倍以上。Yu等[68]通过在目标蛋白上添加四半胱氨酸,利用其与双砷反应产生荧光信号,将分泌蛋白产量提高2.5倍以上。Li等[63]通过构建液滴生成、注入、分选流程,结合生物传感器,有效提高了目标小分子等代谢物的产量。DREMcell还用于微生物培养组学研究,如蜜蜂肠道菌群培养[69]、作物致病拮抗菌株的资源挖掘[70]。英国Sphere Fluidics公司研制了纳升级CytoMine设备,液滴操作体积为0.3纳升,是一款单细胞包裹、检测、分选与克隆验证集成于单一平台的单细胞分析筛选仪器,常用于快速检测单个细胞的外泌分子(如IgG、抗原)等,然后根据液滴荧光信号强度选择特定的单细胞[71]。此外,浙江达普生物技术有限公司的CytosparkTM MSP皮升级液滴系统、深圳华大基因股份有限公司MGIDS-1000P多功能液滴分选一体机、浙江墨卓生物科技有限公司的MobiNova-S1单细胞液滴分选仪、大连华微科技有限公司的HWSeaBreeze X等均实现了皮纳升液滴分选技术与装备开发。上海涛烜科学仪器有限公司基于IADS技术研发了Hypercell高通量单细胞分选平台,每天可测试105—106产生分泌物的目标单细胞[72]。
2.2 微升级液滴培养技术与测试装备微升级液滴培养技术指的是基于微升级别体积的油包水液滴的单细胞培养和分选技术,每天可以完成104—105个样本的测试。在培养方面,微升级液滴按序收集于透气性管路中,管壁良好的气体交换性能为细胞培养提供了硬件基础。同时由于微升液滴比皮纳升液滴体积更大,因此能够支持更长时间、更多种类(放线菌、霉菌等大型细胞)的微生物培养,微生物浓度达到105 CFU/mL①以上。在检测与分选方面,微升级液滴可搭载吸光度、荧光、质谱等各种检测方式,实现细胞的多表型测试。在分选方面,常规使用的电场、光镊等难以产生足够大的驱动力将液滴分选至收集通道。本文作者团队[73]开发了重力场驱动微升液滴至微孔板的分选与收集方法,形成了我国具有自主知识产权的微升液滴分选技术。
① 表示每毫升含有细菌群落的总数。
我国洛阳华清天木生物科技有限公司开发了商业化微生物微液滴培养系统MMC与高通量微升级液滴培养组学系统MISScell装备。MMC系统主要用于微生物的连续进化研究,通过集成液滴识别、光谱检测、微流控芯片和进样模块等功能,实现了对微生物液滴的精确操作,包括发生、培养、监测、分割、融合和分选等过程。MMC液滴体积为2—3微升,一批次产生200个液滴培养单元并可连续传代15天以上,最终分选出具有显著生长优势的底盘细胞。MMC已成功应用于耐高浓度D-山梨醇和耐高温Gluconobacter oxydans菌株[74]、甲醇利用型大肠杆菌[75]等菌株适应性进化。MISScell系统主要用于单细胞高通量培养筛选研究,每批次生成约5 000个2微升单细胞液滴,液滴存储在高透气性管路中进行细胞培养(0—8天),通过光学信号(如光学密度、荧光等)检测分选,搭载机械臂搬运孔板,一批次最多可以收集1 000株优良表型细胞。本文作者团队[73]使用带有荧光标记的大肠杆菌验证了MISScell基于泊松分布包裹单细胞的可行性,并利用该装备实现了谷氨酸棒状杆菌的高通量筛选,从502株突变体中分选出的优势菌株谷氨酸产量提高了25% 以上。此外,法国MilliDrop公司的Milidrop Analyzer液滴培养仪也是一款微升级液滴装备[76],每批次可以生成102—103个细菌、酵母等单细胞微生物液滴,在追踪细菌在不同抗生素压力下的适应性进化[77]、量化肠道细菌的多样性[78]等科学研究中得到应用。
基于FADS、AADS、MADS、IADS技术原理开发的培养类型高通量表型测试典型商业化液滴微流控装备如表 2所示。
|
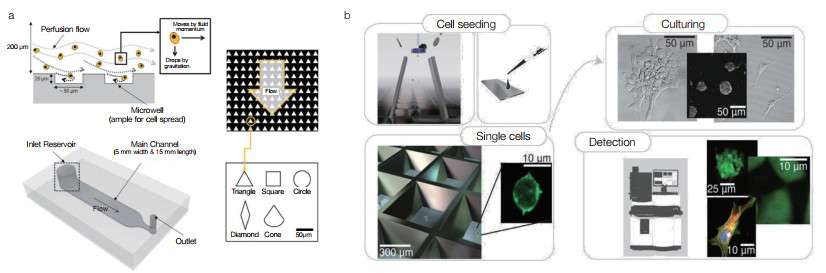
微腔室反应器是指基于微加工技术在硅、玻璃等基板上制作微孔阵列,根据不同需求制作不同形状的腔室,这些腔室具有无菌透气、透明性、低毒性等特点以满足单细胞的培养与代谢[79]。例如,聚合物聚二甲基硅氧烷(PDMS)材料具有疏松多孔、易于加工、生物兼容性好、透明性高等优点,广泛用于细胞的生长代谢观察,其微孔体积包括皮升至微升级别,覆盖微生物到动物细胞所需反应器的体积[80]。微腔室生物反应器中的单细胞研究包括单细胞的捕获、培养和检测分选,单细胞捕获可通过重力驱动[81]、有限稀释法[82]、光电驱动[83]等技术方法将单个细胞导入微腔室(图 3a),接着对微腔室周围环境进行适宜的温度控制和氧气供应,满足细胞在微腔室中的培养需求,最后通过荧光显微等技术,对细胞的生长代谢状态进行连续观察分析,进而挑选出合适的目标细胞(图 3b)。
|
| 图 3 微腔室高通量表型测试装备技术原理 Figure 3 Principle of microchamber high-throughput phenotype testing equipment (a)微腔室单细胞分离;(b)微腔室单细胞培养与检测筛选 (a) Isolation of single cell in microchambers; (b) Cultivation and testing of single cell in microchambers |
皮纳升微腔室是指通过数值模拟和理论分析的方法对微流控芯片尺寸进行精确设计的皮纳升微小孔洞阵列。当样品悬液通入到芯片后,根据泊松分布原理,单个细胞会温和分布至各个微腔室进行生长代谢,单细胞培养后可通过明场成像、荧光成像等检测技术识别单克隆,并基于机械臂(Cobot)挑取、光镊技术(optical tweezers,OT)、光电定位技术(optoelectronic positioning,OEP)将细胞转移至特定位置[83]。
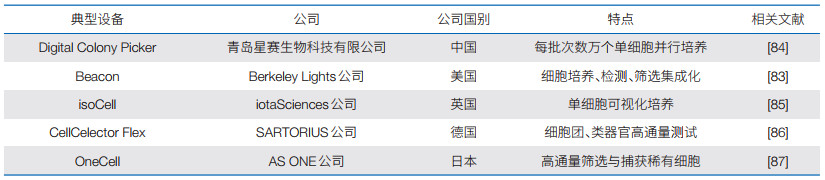
我国青岛星赛生物科技有限公司开发了数字化克隆挑选仪(DCP),该设备搭配的静态皮升级微腔阵列芯片,可容纳数万个单细胞并行培养[84];培养后通过自动对焦系统对每个微腔室进行高分辨率成像,并基于OT技术,将单克隆包裹于微液滴中高效导出,通量为1 000单克隆/小时。美国Berkeley Lights Co., Ltd. 开发了Beacon纳升微腔室细胞表型测试系统,结合光流体芯片(纳升级培养小室和微流管道构成的流体管路系统)和OEP技术,实现了数千个单细胞并行培养、检测、筛选和导出,在抗体筛选、免疫细胞筛选等领域广泛应用[83]。英国iotaSciences公司开发了isoCell高通量、高自动化单细胞可视化培养系统,在培养皿上雕刻出单独的小孔形成纳升级微腔室(6厘米培养皿包含256个腔室)用于单细胞自动化培养与测试[85],每天测试通量达到103以上。此外,德国SARTORIUS公司的CellCelector Flex和日本AS ONE公司的OneCell基于微腔室芯片技术,每批次可分离培养数十万个单细胞,并通过偶联目标抗体或抗原,检测并筛选出目标表型细胞[86, 87]。
3.2 微升级腔室培养技术与测试装备微升级腔室培养技术通常指iChip(isolation chip)技术[88],核心为一种由数百个微型扩散室组成的微型隔离芯片,每个微腔室接种单个细胞后使用滤膜封闭,特定的膜孔径使环境中营养物质、信号分子等可以通过扩散作用进入培养室内,为细胞提供生长所需的养分,但是细胞无法侵入腔室[89, 90],因此可以进行原位环境培养[91, 92]。目前,iChip一般在实验室中自制使用,目前还未报道商业化装备。
基于微腔室培养类型的单细胞高通量表型测试典型商业化装备如表 3所示。
|
本文系统综述了基于微流控技术的工程细胞高通量表型测试技术与装备,包括基于单细胞测试的非培养型技术与装备,基于微液滴、微腔室的单细胞培养测试技术与装备。非培养类型的单细胞测试通常是基于细胞自身或经过生化反应标记的信号进行检测与筛选,适用于胞内、胞膜表型测试。培养类型的细胞表型测试通常需要微型生物反应器来支持单细胞生长代谢,可以实现胞内、胞膜、胞外等多种细胞表型测试。总体来看,在单细胞测试中,FACS与MACS装备通量最高,但是FACS受限于荧光标签的开发[93, 94];MACS依赖细胞表面特定的标志物才能实现抗原抗体结合与磁激活分选;RACS技术在去标签、多参数检测上取得了重要进展,实现了细胞代谢产物、细胞形态、细胞毒性耐受等多表型测试,然而拉曼光谱仍面临背景噪声高、抗干扰能力差,导致测试准确性和通量降低等方面的挑战;IACS在细胞几何结构表型测试中表现出巨大优势,但深度学习算法与商业化装备的集成仍存在局限性。对于细胞培养型表型测试,基于FADS、AADS、IADS、MADS技术,国内外近年来涌现了大量工程细胞高通量表型测试液滴微流控装备[95-97],在高通量、集成化、自动化、多参数检测上取得了关键突破,实现了皮纳升液滴与微升液滴不同尺度下的单细胞培养,然而液滴微流控装备需结合微流控芯片操作,技术操作复杂,门槛较高。此外,微腔室装备历经多年发展,也逐渐形成了单细胞捕获、培养、检测、筛选功能一体化装备,但由于OEP、OT等细胞分离技术通量低,限制了细胞表型测试效率。对比非培养类型的单细胞表型测试,培养类型表型测试技术在细胞生长代谢、细胞环境表型测试中体现出更大优势,而单细胞的优势更多体现在通量、细胞物理参数与几何结构的表型测试中。
对于工程细胞表型测试微流控技术与装备研发的发展方向,本文认为:
(1)发展表型组检测集成,及其与基因型数字化关联。现有微流控技术的高通量表型测试,常以单类型的检测方式为主,如荧光检测、拉曼检测、图像检测等,但在实际实验过程中,单一类型的表型检测方式常无法满足工程细胞的多维度检测需求,从而造成表型数据单一、假阳性结果较多等问题,对后期的数据分析产生干扰。因此,不同检测方式的自由组合,实现工程细胞的多个维度表型参数同时检测,将为工程细胞分析提供更加精确、丰富的表型数据结果。同时结合高通量建库测序技术、生信分析技术、人工智能技术等,实现表型组和基因型的数字化关联,对工程细胞进行系统性深度研究分析,为其改造设计提供精准理性指导。
(2)微流控技术与传统孔板-移液机器机器人技术有机结合,铸造工程细胞高通量表型测试装备集成平台。工程细胞表型测试具有多维度、跨尺度等特性,微流控表型测试技术虽然能够支持实现对多个表型维度进行高通量测试,其尺度往往局限于微升级体积以下,部分表型信号较弱甚至缺乏表达。同时,对于基因型的获得仍需经过PCR扩增、核酸提取等手段获得核酸样本,工作量大且过程较为烦琐。现有的机器人移液技术及孔板自动操控技可以提供孔板水平(百微升—毫升级)规模的移液操作和检测,能够有效解决微流控表型测试及筛选后下游的烦琐和受限工作。因此,微流控技术与传统孔板-移液机器机器人技术的有机结合,实现以多孔板为标准物理接口的自动化对接,有望为工程细胞高通量表型测试和表型-基因型数字化关联提供一站式的完整解决方案。同时,结合具体典型应用场景中工程细胞的实验流程,串联多种不同关键技术,实现工程细胞测试的全流程固化,实现工程细胞高通量表型测试自动化平台。
在科学仪器的国产化研究方面,历经几十年不断发展,特别是“十二五”以来,在国家自然科学基金科研仪器专项和科学技术部科研仪器专项的支持下,我国仪器装备行业已经逐步形成了相对完善的科技创新体系,取得重要突破。然而,国际科学仪器产业仍然以发达国家主导,美国、欧洲和日本的企业占据了高端市场的主要份额,我国科学仪器行业面临着以下关键问题:①科学仪器对国外依存度高,国产仪器使用率不高;②产业发展集聚度低,缺少行业龙头企业;③科学仪器自主研制面临管控禁运的挑战。
因此,对于我国高端仪器装备的发展,提出以下建议,以期最终实现我国科学仪器领域的自主创新能力和产业竞争力提高:①坚定自主研究战略;②以大科学设施集群为引领,推动空间集聚发展;③坚持科学引领,制造技术、资本支持协同提升;④加大专业人才队伍建设力度;⑤坚持资源统筹,持续完善创新生态。
| [1] |
Bashor C J, Hilton I B, Bandukwala H, et al. Engineering the next generation of cell-based therapeutics. Nature Reviews Drug Discovery, 2022, 21(9): 655-675. DOI:10.1038/s41573-022-00476-6 |
| [2] |
Liu Y, Nielsen J. Recent trends in metabolic engineering of microbial chemical factories. Current Opinion in Biotechnology, 2019, 60: 188-197. DOI:10.1016/j.copbio.2019.05.010 |
| [3] |
Opgenorth P, Costello Z, Okada T, et al. Lessons from two design-build-test-learn cycles of dodecanol production in Escherichia coli aided by machine learning. ACS Synthetic Biology, 2019, 8(6): 1337-1351. DOI:10.1021/acssynbio.9b00020 |
| [4] |
Bhunia A K, Singh A K, Parker K, et al. Petri-plate, bacteria, and laser optical scattering sensor. Frontiers in Cellular and Infection Microbiology, 2022. DOI:10.3389/fcimb.2022.1087074 |
| [5] |
Abatzoglou I, Zois C E, Pouliliou S, et al. Establishment and validation of a method for multi-dose irradiation of cells in 96-well microplates. Biochemical and Biophysical Research Communications, 2013, 431(3): 456-459. DOI:10.1016/j.bbrc.2012.12.146 |
| [6] |
Zeng W Z, Guo L K, Xu S, et al. High-throughput screening technology in industrial biotechnology. Trends in Biotechnology, 2020, 38(8): 888-906. DOI:10.1016/j.tibtech.2020.01.001 |
| [7] |
Huang Y M, Sheth R U, Zhao S J, et al. High-throughput microbial culturomics using automation and machine learning. Nature Biotechnology, 2023, 41(10): 1424-1433. DOI:10.1038/s41587-023-01674-2 |
| [8] |
Manz A, Graber N, Widmer H M. Miniaturized total chemical analysis systems: A novel concept for chemical sensing. Sensors and Actuators B: Chemical, 1990, 1(6): 244-248. |
| [9] |
Sciambi A, Abate A R. Accurate microfluidic sorting of droplets at 30 kHz. Lab on a Chip, 2015, 15(1): 47-51. DOI:10.1039/C4LC01194E |
| [10] |
Whitesides G M. The origins and the future of microfluidics. Nature, 2006, 442: 368-373. DOI:10.1038/nature05058 |
| [11] |
Zhang J, Ren L, Zhang L, et al. Single-cell rapid identification, in situ viability and vitality profiling, and genome-based source-tracking for probiotics products. iMeta, 2023. DOI:10.1002/imt2.117 |
| [12] |
Nawar S, Stolaroff J K, Ye C W, et al. Parallelizable microfluidic dropmakers with multilayer geometry for the generation of double emulsions. Lab on a Chip, 2020, 20(1): 147-154. DOI:10.1039/C9LC00966C |
| [13] |
Bhatia S N, Ingber D E. Microfluidic organs-on-chips. Nature Biotechnology, 2014, 32(8): 760-772. DOI:10.1038/nbt.2989 |
| [14] |
Hodne K, Weltzien F A. Single-cell isolation and gene analysis: Pitfalls and possibilities. International Journal of Molecular Sciences, 2015, 16(11): 26832-26849. DOI:10.3390/ijms161125996 |
| [15] |
Jakobsson O, Grenvall C, Nordin M, et al. Acoustic actuated fluorescence activated sorting of microparticles. Lab on a Chip, 2014, 14(11): 1943-1950. DOI:10.1039/C3LC51408K |
| [16] |
Newell E W, Sigal N, Bendall S C, et al. Cytometry by time-of-flight shows combinatorial cytokine expression and virus-specific cell niches within a continuum of CD8+ T cell phenotypes. Immunity, 2012, 36(1): 142-152. DOI:10.1016/j.immuni.2012.01.002 |
| [17] |
Song Y Z, Kaster A K, Vollmers J, et al. Single-cell genomics based on Raman sorting reveals novel carotenoid-containing bacteria in the Red Sea. Microbial Biotechnology, 2017, 10(1): 125-137. DOI:10.1111/1751-7915.12420 |
| [18] |
Hasle N, Cooke A, Srivatsan S, et al. High-throughput, microscope-based sorting to dissect cellular heterogeneity. Molecular Systems Biology, 2020, 16(6): e9442. DOI:10.15252/msb.20209442 |
| [19] |
Kim U, Tom Soh H. Simultaneous sorting of multiple bacterial targets using integrated dielectrophoretic-magnetic activated cell sorter. Lab on a Chip, 2009, 9(16): 2313-2318. DOI:10.1039/b903950c |
| [20] |
Johansson L, Nikolajeff F, Johansson S, et al. On-chip fluorescence-activated cell sorting by an integrated miniaturized ultrasonic transducer. Analytical Chemistry, 2009, 81(13): 5188-5196. DOI:10.1021/ac802681r |
| [21] |
Robinson J P, Ostafe R, Iyengar S N, et al. Flow cytometry: The next revolution. Cells, 2023. DOI:10.3390/cells12141875 |
| [22] |
Grzeschik J, Yanakieva D, Roth L, et al. Yeast surface display in combination with fluorescence-activated cell sorting enables the rapid isolation of antibody fragments derived from immunized chickens. Biotechnology Journal, 2019. DOI:10.1002/biot.201800466 |
| [23] |
Hewitt B M, Singhal N, Elliot R G, et al. Novel fiber optic detection method for in situ analysis of fluorescently labeled biosensor organisms. Environmental Science Technology, 2012, 46(10): 5414-5421. DOI:10.1021/es300164p |
| [24] |
Ostafe R, Prodanovic R, Commandeur U, et al. Flow cytometry-based ultra-high-throughput screening assay for cellulase activity. Analytical Biochemistry, 2013, 435(1): 93-98. DOI:10.1016/j.ab.2012.10.043 |
| [25] |
Gao J S, Du M H, Zhao J H, et al. Design of a genetically encoded biosensor to establish a high-throughput screening platform for L-cysteine overproduction. Metabolic Engineering, 2022, 73: 144-157. DOI:10.1016/j.ymben.2022.07.007 |
| [26] |
Kortmann M, Mack C, Baumgart M, et al. Pyruvate carboxylase variants enabling improved lysine production from glucose identified by biosensor-based high-throughput fluorescence-activated cell sorting screening. ACS Synthetic Biology, 2019, 8(2): 274-281. DOI:10.1021/acssynbio.8b00510 |
| [27] |
Wagner J M, Liu L Q, Yuan S F, et al. A comparative analysis of single cell and droplet-based FACS for improving production phenotypes: Riboflavin overproduction in Yarrowia lipolytica. Metabolic Engineering, 2018, 47: 346-356. DOI:10.1016/j.ymben.2018.04.015 |
| [28] |
Ma C X, Tan Z L, Lin Y, et al. Gel microdroplet–based high-throughput screening for directed evolution of xylanase-producing Pichia pastoris. Journal of Bioscience and Bioengineering, 2019, 128(6): 662-668. DOI:10.1016/j.jbiosc.2019.05.008 |
| [29] |
Song H K, Kim J M, Noh E M, et al. Role of NOX1 and NOX5 in protein kinase C/reactive oxygen species-mediated MMP-9 activation and invasion in MCF-7 breast cancer cells. Molecular Medicine Reports, 2024, 30(4): 188. DOI:10.3892/mmr.2024.13312 |
| [30] |
Song Y Z, Yin H B, Huang W E. Raman activated cell sorting. Current Opinion in Chemical Biology, 2016, 33: 1-8. DOI:10.1016/j.cbpa.2016.04.002 |
| [31] |
Wang Y, Ji Y T, Wharfe E S, et al. Raman activated cell ejection for isolation of single cells. Analytical Chemistry, 2013, 85(22): 10697-10701. DOI:10.1021/ac403107p |
| [32] |
Xu T, Gong Y H, Su X L, et al. Phenome-genome profiling of single bacterial cell by Raman-activated gravity-driven encapsulation and sequencing. Small, 2020. DOI:10.1002/smll.202001172 |
| [33] |
Wang X X, Ren L H, Su Y T, et al. Raman-activated droplet sorting (RADS) for label-free high-throughput screening of microalgal single-cells. Analytical Chemistry, 2017, 89(22): 12569-12577. DOI:10.1021/acs.analchem.7b03884 |
| [34] |
Wang X X, Xin Y, Ren L H, et al. Positive dielectrophoresis-based Raman-activated droplet sorting for culture-free and label-free screening of enzyme function in vivo. Science Advances, 2020. DOI:10.1126/sciadv.abb352 |
| [35] |
Su X L, Gong Y H, Gou H L, et al. Rational optimization of Raman-activated cell ejection and sequencing for bacteria. Analytical Chemistry, 2020, 92(12): 8081-8089. DOI:10.1021/acs.analchem.9b05345 |
| [36] |
Vinay Kumar B N, Guo S X, Bocklitz T, et al. Demonstration of carbon catabolite repression in naphthalene degrading soil bacteria via Raman spectroscopy based stable isotope probing. Analytical Chemistry, 2016, 88(15): 7574-7582. DOI:10.1021/acs.analchem.6b01046 |
| [37] |
Kaczor A, Baranska M. Structural changes of carotenoid astaxanthin in a single algal cell monitored in situ by Raman spectroscopy. Analytical Chemistry, 2011, 83(20): 7763-7770. DOI:10.1021/ac201302f |
| [38] |
Ji Y T, He Y H, Cui Y B, et al. Raman spectroscopy provides a rapid, non-invasive method for quantitation of starch in live, unicellular microalgae. Biotechnology Journal, 2014, 9(12): 1512-1518. DOI:10.1002/biot.201400165 |
| [39] |
Yan S S, Wang S Y, Qiu J X, et al. Raman spectroscopy combined with machine learning for rapid detection of food-borne pathogens at the single-cell level. Talanta, 2021, 226: 122195. DOI:10.1016/j.talanta.2021.122195 |
| [40] |
Diao Z D, Kan L Y, Zhao Y L, et al. Artificial intelligence-assisted automatic and index-based microbial single-cell sorting system for One-Cell-One-Tube. mLife, 2022, 1(4): 448-459. DOI:10.1002/mlf2.12047 |
| [41] |
Nitta N, Sugimura T, Isozaki A, et al. Intelligent image-activated cell sorting. Cell, 2018, 175(1): 266-276. DOI:10.1016/j.cell.2018.08.028 |
| [42] |
Isozaki A, Mikami H, Hiramatsu K, et al. A practical guide to intelligent image-activated cell sorting. Nature Protocols, 2019, 14(8): 2370-2415. DOI:10.1038/s41596-019-0183-1 |
| [43] |
Zhao Y Q, Isozaki A, Herbig M, et al. Intelligent sort-timing prediction for image-activated cell sorting. Cytometry Part A, 2023, 103(1): 88-97. DOI:10.1002/cyto.a.24664 |
| [44] |
Headland S E, Jones H R, D'Sa A S V, et al. Cutting-edge analysis of extracellular microparticles using ImageStream (X) imaging flow cytometry. Scientific Reports, 2014, 4: 5237. DOI:10.1038/srep05237 |
| [45] |
Zuba-Surma E, Kucia M, Ratajczak M. "Decoding the dots": The ImageStream System (ISS) as a novel and powerful tool for flow cytometric analysis. Open Life Sciences, 2008, 3(1): 1-10. DOI:10.2478/s11535-007-0044-8 |
| [46] |
Welzel G, Seitz D, Schuster S. Magnetic-activated cell sorting (MACS) can be used as a large-scale method for establishing zebrafish neuronal cell cultures. Scientific Reports, 2015, 5: 795. |
| [47] |
Adams J D, Kim U, Tom Soh H. Multitarget magnetic activated cell sorter. PNAS, 2008, 105(47): 18165-18170. DOI:10.1073/pnas.0809795105 |
| [48] |
Pan J, Wan J. Methodological comparison of FACS and MACS isolation of enriched microglia and astrocytes from mouse brain. Journal of Immunological Methods, 2020, 486: 112834. DOI:10.1016/j.jim.2020.112834 |
| [49] |
Münz C, Steinman R M, Fujii S I. Dendritic cell maturation by innate lymphocytes: Coordinated stimulation of innate and adaptive immunity. Journal of Experimental Medicine, 2005, 202(2): 203-207. DOI:10.1084/jem.20050810 |
| [50] |
Chavarria V, Ortiz-Islas E, Salazar A, et al. Lactate-loaded nanoparticles induce glioma cytotoxicity and increase the survival of rats bearing malignant glioma brain tumor. Pharmaceutics, 2022, 14(2): 327. DOI:10.3390/pharmaceutics14020327 |
| [51] |
Kulis M, Merkel A, Heath S, et al. Whole-genome fingerprint of the DNA methylome during human B cell differentiation. Nature Genetics, 2015, 47(7): 746-756. DOI:10.1038/ng.3291 |
| [52] |
Laghmouchi A, Hoogstraten C, Frederik Falkenburg J H, et al. Long-term in vitro persistence of magnetic properties after magnetic bead-based cell separation of T cells. Scandinavian Journal of Immunology, 2020. DOI:10.1111/sji.12924 |
| [53] |
Mazutis L, Gilbert J, Lloyd Ung W, et al. Single-cell analysis and sorting using droplet-based microfluidics. Nature Protocols, 2013, 8(5): 870-891. DOI:10.1038/nprot.2013.046 |
| [54] |
Shembekar N, Hu H X, Eustace D, et al. Single-cell droplet microfluidic screening for antibodies specifically binding to target cells. Cell Reports, 2018, 22(8): 2206-2215. DOI:10.1016/j.celrep.2018.01.071 |
| [55] |
Zhang C T, Wu X H, Song F Q, et al. Core-shell droplet-based microfluidic screening system for filamentous fungi. ACS Sensors, 2023, 8(9): 3468-3477. DOI:10.1021/acssensors.3c01018 |
| [56] |
Baret J C, Miller O J, Taly V, et al. Fluorescence-activated droplet sorting (FADS): Efficient microfluidic cell sorting based on enzymatic activity. Lab on a Chip, 2009, 9(13): 1850-1858. DOI:10.1039/b902504a |
| [57] |
Ma F Q, Chung M T, Yao Y, et al. Efficient molecular evolution to generate enantioselective enzymes using a dual-channel microfluidic droplet screening platform. Nature Communications, 2018, 9(1): 1030. DOI:10.1038/s41467-018-03492-6 |
| [58] |
Jiang J J, Yang G Y, Ma F Q. Fluorescence coupling strategies in fluorescence-activated droplet sorting (FADS) for ultrahigh-throughput screening of enzymes, metabolites, and antibodies. Biotechnology Advances, 2023. DOI:10.1016/j.biotechadv.2023.108173 |
| [59] |
Shi L X, Liu P, Tan Z J, et al. Complete depolymerization of PET wastes by an evolved PET hydrolase from directed evolution. Angewandte Chemie (International Ed), 2023. DOI:10.1002/anie.202218390 |
| [60] |
Yuan H L, Tu R, Tong X W, et al. Ultrahigh-throughput screening of industrial enzyme-producing strains by droplet-based microfluidic system. Journal of Industrial Microbiology Biotechnology, 2022. DOI:10.1093/jimb/kuac007 |
| [61] |
Abalde-Cela S, Gould A, Liu X, et al. High-throughput detection of ethanol-producing cyanobacteria in a microdroplet platform. Journal of the Royal Society, Interface, 2015. DOI:10.1098/rsif.2015.0216 |
| [62] |
Wang B L, Ghaderi A, Zhou H, et al. Microfluidic high-throughput culturing of single cells for selection based on extracellular metabolite production or consumption. Nature Biotechnology, 2014, 32(5): 473-478. DOI:10.1038/nbt.2857 |
| [63] |
Li S, Zhang Y, Li L, et al. Establishment of picodroplet-based co-culture system to improve erythritol production in Yarrowia lipolytica. Biochemical Engineering Journal, 2023. DOI:10.1016/j.bej.2023.109036 |
| [64] |
Li S, Liao X H, Yu X Y, et al. Combining genetically encoded biosensors with droplet microfluidic system for enhanced glutaminase production by Bacillus amyloliquefaciens. Biochemical Engineering Journal, 2022. DOI:10.1016/j.bej.2022.108586 |
| [65] |
Gielen F, Hours R, Emond S, et al. Ultrahigh-throughput-directed enzyme evolution by absorbance-activated droplet sorting (AADS). PNAS, 2016, 113(47): 7383-7389. |
| [66] |
Holland-Moritz D A, Wismer M K, Mann B F, et al. Mass activated droplet sorting (MADS) enables high-throughput screening of enzymatic reactions at nanoliter scale. Angewandte Chemie (International Ed), 2020, 59(11): 4470-4477. DOI:10.1002/anie.201913203 |
| [67] |
Zang E, Brandes S, Tovar M, et al. Real-time image processing for label-free enrichment of Actinobacteria cultivated in picolitre droplets. Lab on a Chip, 2013, 13(18): 3707-3713. DOI:10.1039/c3lc50572c |
| [68] |
Yu X Y, Li S, Feng H B, et al. CRISPRi-microfluidics screening enables genome-scale target identification for high-titer protein production and secretion. Metabolic Engineering, 2023, 75: 192-204. DOI:10.1016/j.ymben.2022.12.004 |
| [69] |
Meng Y J, Li S, Zhang C, et al. Strain-level profiling with picodroplet microfluidic cultivation reveals host-specific adaption of honeybee gut symbionts. Microbiome, 2022, 10(1): 140. DOI:10.1186/s40168-022-01333-9 |
| [70] |
Wei L F, Li S, Pan H, et al. Ultrahigh-throughput screening of antagonistic bacteria against Erwinia carotovoraEcc15 based on droplet microfluidics. Food Science and Human Wellness, 2024. DOI:10.26599/FSHW.2024.9250263 |
| [71] |
Gaa R, Menang-Ndi E, Pratapa S, et al. Versatile and rapid microfluidics-assisted antibody discovery. mAbs, 2021, 13(1): 1978130. DOI:10.1080/19420862.2021.1978130 |
| [72] |
Yu T, Hull J, Ruiz A, et al. Expediting antibody discovery using Bioelectronica's HypercellTM platform. The Journal of Immunology, 2020, 204(1_Supplement): 86-36. |
| [73] |
Jian X J, Guo X J, Cai Z S, et al. Single-cell microliter-droplet screening system (MISS Cell): An integrated platform for automated high-throughput microbial monoclonal cultivation and picking. Biotechnology and Bioengineering, 2023, 120(3): 778-792. DOI:10.1002/bit.28300 |
| [74] |
Liu L, Zeng W Z, Yu S Q, et al. Rapid enabling of Gluconobacter oxydans resistance to high D-sorbitol concentration and high temperature by microdroplet-aided adaptive evolution. Frontiers in Bioengineering and Biotechnology, 2021, 9: 731247. DOI:10.3389/fbioe.2021.731247 |
| [75] |
Jian X J, Guo X J, Wang J, et al. Microbial microdroplet culture system (MMC): An integrated platform for automated, high-throughput microbial cultivation and adaptive evolution. Biotechnology and Bioengineering, 2020, 117(6): 1724-1737. DOI:10.1002/bit.27327 |
| [76] |
Jiang L, Boitard L, Broyer P, et al. Digital antimicrobial susceptibility testing using the MilliDrop technology. European Journal of Clinical Microbiology Infectious Diseases, 2016, 35(3): 415-422. DOI:10.1007/s10096-015-2554-z |
| [77] |
Postek W, Garstecki P. Droplet microfluidics for high-throughput analysis of antibiotic susceptibility in bacterial cells and populations. Accounts of Chemical Research, 2022, 55(5): 605-615. DOI:10.1021/acs.accounts.1c00729 |
| [78] |
Villa M M, Bloom R J, Silverman J D, et al. Interindividual variation in dietary carbohydrate metabolism by gut bacteria revealed with droplet microfluidic culture. mSystems, 2020, 5(3): e00864-19. |
| [79] |
Sun L L, Zhang L K, Yang X, et al. A simple and low-cost method for fabrication of polydimethylsiloxane microfludic chips. Journal of Nanoscience and Nanotechnology, 2021, 21(11): 5635-5641. DOI:10.1166/jnn.2021.19464 |
| [80] |
Gambardella G, Viscido G, Tumaini B, et al. A single-cell analysis of breast cancer cell lines to study tumour heterogeneity and drug response. Nature Communications, 2022, 13(1): 1714. DOI:10.1038/s41467-022-29358-6 |
| [81] |
Park J Y, Morgan M, Sachs A N, et al. Single cell trapping in larger microwells capable of supporting cell spreading and proliferation. Microfluidics and Nanofluidics, 2010, 8(2): 263-268. DOI:10.1007/s10404-009-0503-9 |
| [82] |
Lindström S, Andersson-Svahn H. Single-cell culture in microwells. Single-Cell Analysis. Totowa: Humana Press, 2012: 41-52.
|
| [83] |
Le K, Tan C, Le H, et al. Assuring clonality on the beacon digital cell line development platform. Biotechnology Journal, 2020, 15(1): e1900247. DOI:10.1002/biot.201900247 |
| [84] |
Wang X X, Ren L H, Diao Z D, et al. Robust spontaneous Raman flow cytometry for single-cell metabolic phenome profiling via pDEP-DLD-RFC. Advanced Science, 2023, 10(16): e2207497. DOI:10.1002/advs.202207497 |
| [85] |
Vallone V F, Telugu N S, Fischer I, et al. Methods for automated single cell isolation and sub-cloning of human pluripotent stem cells. Current Protocols in Stem Cell Biology, 2020, 55(1): e123. DOI:10.1002/cpsc.123 |
| [86] |
Matochko W L, Nelep C, Chen W C, et al. CellCelectorTM as a platform in isolating primary B cells for antibody discovery. Antibody Therapeutics, 2022, 5(1): 11-17. DOI:10.1093/abt/tbab030 |
| [87] |
Yoshimoto N, Kida A, Jie X, et al. An automated system for high-throughput single cell-based breeding. Scientific Reports, 2013, 3: 1191. DOI:10.1038/srep01191 |
| [88] |
Berdy B, Spoering A L, Ling L L, et al. In situ cultivation of previously uncultivable microorganisms using the ichip. Nature Protocols, 2017, 12(10): 2232-2242. DOI:10.1038/nprot.2017.074 |
| [89] |
Nichols D, Cahoon N, Trakhtenberg E M, et al. Use of ichip for high-throughput in situ cultivation of "uncultivable" microbial species. Applied and Environmental Microbiology, 2010, 76(8): 2445-2450. DOI:10.1128/AEM.01754-09 |
| [90] |
Li M, Raza M, Song S, et al. Application of culturomics in fungal isolation from mangrove sediments. Microbiome, 2023, 11(1): 272. DOI:10.1186/s40168-023-01708-6 |
| [91] |
Liu H Z, Xue R, Wang Y L, et al. FACS-iChip: A highefficiency iChip system for microbial 'dark matter' mining. Marine Life Science Technology, 2020, 3(2): 162-168. |
| [92] |
Lodhi A F, Zhang Y, Adil M, et al. Antibiotic discovery: Combining isolation chip (iChip) technology and co-culture technique. Applied Microbiology and Biotechnology, 2018, 102(17): 7333-7341. DOI:10.1007/s00253-018-9193-0 |
| [93] |
Herzenberg L A, Parks D, Sahaf B, et al. The history and future of the fluorescence activated cell sorter and flow cytometry: A view from Stanford. Clinical Chemistry, 2002, 48(10): 1819-1827. DOI:10.1093/clinchem/48.10.1819 |
| [94] |
Uchinomiya S, Nagaura T, Weber M, et al. Fluorescence-based detection of fatty acid β-oxidation in cells and tissues using quinone methide-releasing probes. Journal of the American Chemical Society, 2023, 145(14): 8248-8260. DOI:10.1021/jacs.3c02043 |
| [95] |
Tu R, Li L P, Yuan H L, et al. Biosensor-enabled droplet microfluidic system for the rapid screening of 3-dehydroshikimic acid produced in Escherichia coli. Journal of Industrial Microbiology Biotechnology, 2020, 47(12): 1155-1160. DOI:10.1007/s10295-020-02316-1 |
| [96] |
He R L, Ding R H, Heyman J A, et al. Ultra-high-throughput picoliter-droplet microfluidics screening of the industrial cellulase-producing filamentous fungus Trichoderma reesei. Journal of Industrial Microbiology Biotechnology, 2019, 46(11): 1603-1610. DOI:10.1007/s10295-019-02221-2 |
| [97] |
Qiao Y X, Zhao X Y, Zhu J, et al. Fluorescence-activated droplet sorting of lipolytic microorganisms using a compact optical system. Lab on a Chip, 2017, 18(1): 190-196. |




